简介
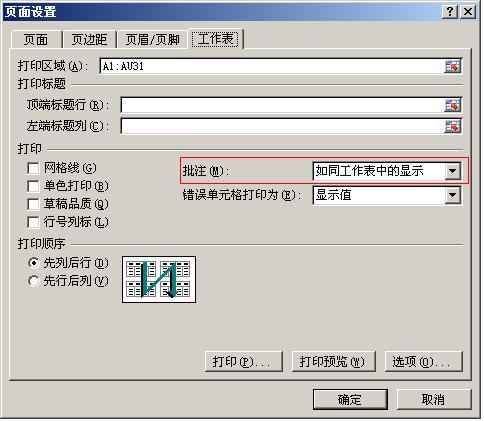
建立模型进行分子动力学模拟后,对得到的轨迹进行主成分分析(PCA),绘制相关性矩阵(Correlation Matrix)和自由能井图(Free Energy Landscape)和dssp图(Definition of Secondary Structure of Proteins)
绘制相关性矩阵图:
使用cpptraj计算轨迹的相关性矩阵
#进入amber的cpptraj程序:
cpptraj##第一步,计算轨迹的平均结构:
parm tyr.prmtop
trajin tyr_500.crd#计算非H原子的rmsd,保存为数据集rmsd-dataset,选择第一帧为参考文件:
rms rmsd-dataset first !@H=#根据轨迹计算结构的平均构象,保存为数据集average-dataset:
average crdset average-dataset
run计算Cα原子的相关性矩阵,输出为ca_matrix.gnu文件:
#利用1~30帧,计算所有CA的相关性矩阵,
#输出文件为ca_matrix.gnu,保存数据集为ca_matrix:
matrix covar out ca_matrix.gnu name ca_matrix start 1 stop 30 @CA给数据前后补上脚本,具体例子如下:
#设置保存为pdf文件
set terminal pdfcairo enhanced color size 8,6 linewidth 3 font "Time New Roman,20"
set output "Correlation Matrix.pdf"set size square
set pm3d map
#设置色条颜色,rainbow色条:
set palette rgb 30,31,32
#差值法解决马赛克问题:0,0是插入的值,设置越大,马赛克消除效果越好。
#0,0表示让系统自动优化:
set pm3d interpolate 0,0
#设置标题和轴标签:
set title "Correlation matrix" font "Time New Roman,14"
set xlabel "atom CA" font "Time New Roman,10"
set ylabel "atom CA" font "Time New Roman,10"
#隐藏图例:
unset key
#设置x,y轴范围:
set yrange [1:265]
set xrange [1:265]
#设置轴的小刻度线:
set mxtics 3
set mytics 3
#刻度线向外:
set xtics out
set ytics out
#设置色条的刻度朝内:
set cbtics in
#隐藏对称的刻度轴:
set tics nomirror
#设置色条的刻度长度:
set cbtics scale 3
#绘图数据:
splot "-" with pm3d title "pca-out.gnu"1.000 1.000 1.0001.000 2.000 0.9041.000 3.000 0.7031.000 4.000 0.5231.000 5.000 0.7091.000 6.000 0.7531.000 7.000 0.6381.000 8.000 0.6491.000 9.000 0.6941.000 10.000 0.6651.000 11.000 0.6211.000 12.000 0.5981.000 13.000 0.5861.000 14.000 0.5491.000 15.000 0.5091.000 16.000 0.5051.000 17.000 0.5111.000 18.000 0.5491.000 19.000 0.6081.000 20.000 0.5721.000 21.000 0.582
[过多的数据省略...]set output
end
pause -1在gnuplot里拖入该gun文件。
查看绘制的效果:
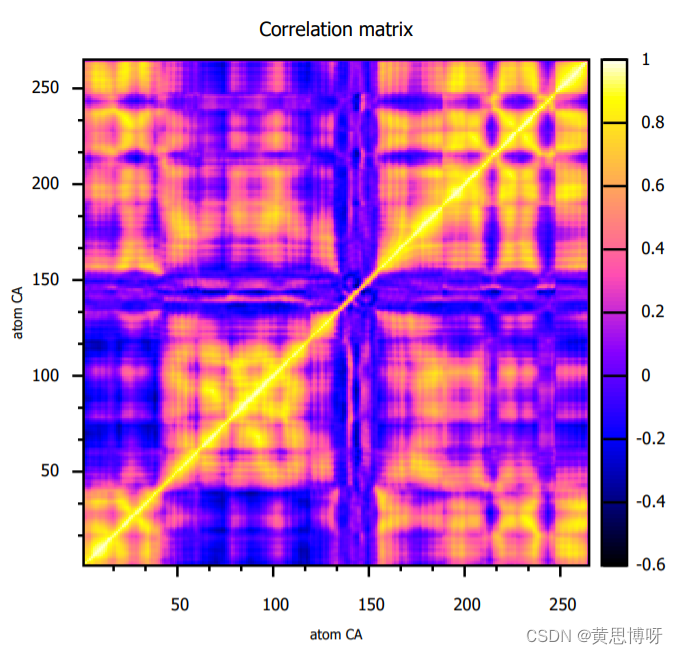
相关性矩阵的图像呈对角线对称,色条两端颜色代表的区域对应的残基有着越强的运动相关性,颜色越浅的区域对应的残基运动相关性越弱。
用cpptraj进行蛋白质二级结构分析(DSSP)
编写cpptraj脚本dssp.in
parm tyr.parm7 [top] #导入拓扑文件
trajin tyr.nc parm [top] #导入100frame的轨迹文件center origin :236&!@H= mass #将残基236的质心作为盒子的中心对体系进行移动
image origin center familiar
reference tyr.pdb #导入坐标参考文件
secstruct :15-68 out dssp15_68.gnu #计算15-68残基的dssp
run
quit注意:这里reference tyr.pdb很重要,不用pdb文件,它识别不了提取的残基id
运行脚本:
cpptraj dssp.in运行gnuplot进行绘制:
gnuplot dssp15_68.gnu调整gnu脚本:
#设置保存为pdf文件
set terminal pdfcairo enhanced color size 8,6 linewidth 3 font "Time New Roman,20"
set output "DefinitionofSecondaryStructureofProteins.pdf"
set size square
set pm3d map corners2color c1
#隐藏图例:
unset key
#范围:
set yr [1.000:54.000]
set xrange [1.000:100.000]
#刻度值:
set ytics ("ILE:19" 5.000,"GLY:29" 15.000,"ARG:39" 25.000,"PHE:49" \35.000,"GLN:59" 45.000,"PRO:68" 54.000) font "Times New Roman,10"
set xtics ("0" 0,"20" 10,"40" 20,"60" 30,"80" 40,"100" 50,"120" 60,"140" 70,"160"\80,"180" 90,"200"100) font "Times New Roman,10"#设置整个图表的空间(面积)大小:
##数值表示距离窗口左边界的距离:
set lmargin at screen 0.15
set rmargin at screen 0.85
##数值表示距离窗口下边界的距离:
set tmargin at screen 0.78
set bmargin at screen 0.20
#标题和刻度标签:
set xlabel "Time (ps)"
set ylabel "residues"
set title "Definition of Secondary Structure of Proteins 15-68"
#刻度线条外翻:
set tics out
#色条的范围是 -0.5~7.5
set cbrange [-0.0500:0.7500]
#调整色条刻度值:
set cbtics("None" -0.0500,"Extended" 0.05000,"Bridge" 0.1500,"3-10" \
0.2500,"Alpha" 0.3500,"Pi" 0.4500,"Turn" 0.5500,"Bend" 0.6500)
set palette maxcolors 8
set palette defined (0 "#000000",1 "#0000FF",4 "#00FF00",7 "#FF0000")
#调整色条刻度线:
set cbtics in
set cbtics scale 2.8
set tics nomirror
splot "-" with pm3d title "dssp15_68.gnu"1.000 1.000 0.01.000 2.000 0.01.000 3.000 0.71.000 4.000 0.01.000 5.000 0.01.000 6.000 0.01.000 7.000 0.11.000 8.000 0.11.000 9.000 0.11.000 10.000 0.1[过多的数据省略]set output
end
pause -1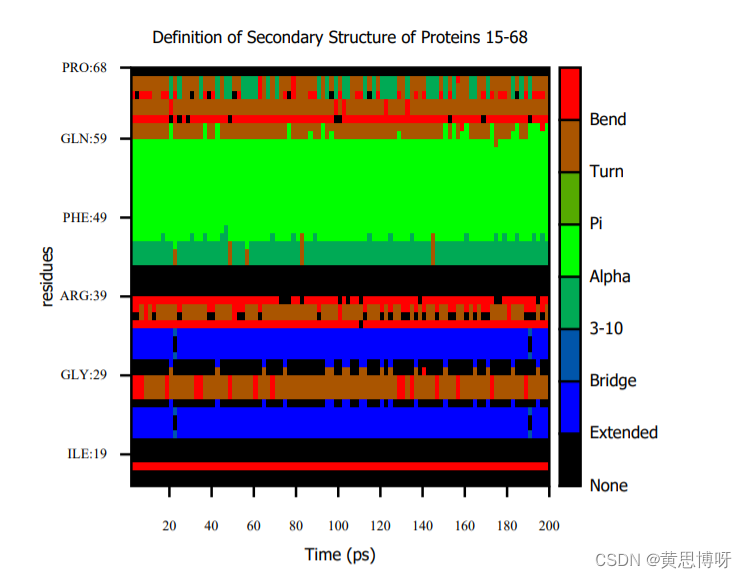
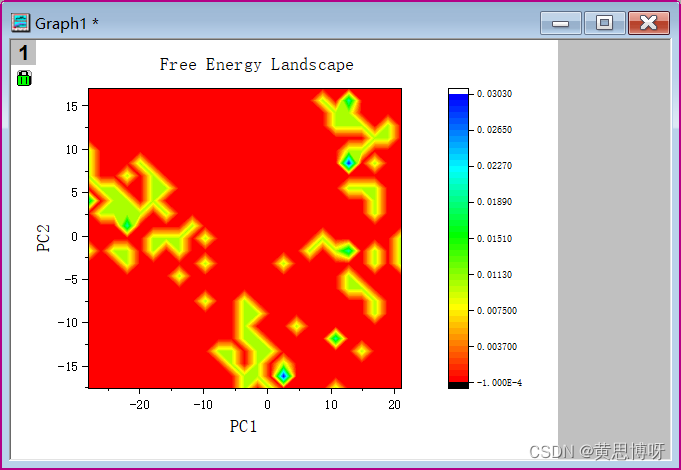
利用cpptraj进行PCA分析绘制自由能井图
编写脚本pca.in:
parm tyr.prmtop [p]
trajin tyr.crd parm [p]
rms rms-data :1-264&!@H= first #生成rmsd数据集
run
average crdset average-dataset :1-264&!@H= #生成平均构象数据集
run
createcrd tyr-trajectories-dataset #建立一个轨迹数据集
runcrdaction tyr-trajectories-dataset \ #计算这个轨迹数据集的rmsd,参考平均构象数据集
rms ref average-dataset :1-264&!@H=crdaction tyr-trajectories-dataset \ #计算协方差矩阵
matrix covar name tyr-covar-dataset :1-264&!@H=
runrunanalysis diagmatrix tyr-covar-dataset out tyr-evecs.dat \ #获取前3个特征向量
vecs 3 name my-evecs \
nmwiz nmwizvecs 3 nmwizfile tyr.nmd nmwizmask :1-264&!@H=crdaction tyr-trajectories-dataset projection TYR modes my-evecs \
beg 1 end 3 :1-264&!@H=hist TYR:1 bins 25 out tyr-hist.agr norm name TYR-1
hist TYR:2 bins 25 out tyr-hist.agr norm name TYR-2
hist TYR:3 bins 25 out tyr-hist.agr norm name TYR-3
hist TYR:1 TYR:2 bins 25 out pca-out.gnu norm name all_1_2 #将PC1和PC2输出为一个gnu文件
run运行脚本:
cpptraj pca.in得到的图像有点马赛克感觉;而且色图外围还有一圈白色的留白;还有颜色也不是我们熟悉的红蓝色条的热图。
进行脚本的润色:
#设置保存为pdf文件:
set terminal pdfcairo enhanced color size 8,6 linewidth 3 font "Time New Roman,20"
set output "Free Energy Landscape.pdf"
#必须要在splot末尾加上"set output"语句,否则保存的文件会损坏!
set size square
set pm3d map
#设置色条颜色,rainbow色条:
set palette rgb -31,13,22
#差值法解决马赛克问题:0,0是插入的值,设置越大,马赛克消除效果越好。
#0,0表示让系统自动优化:
set pm3d interpolate 0,0
#设置标题和轴标签:
set title "Free Energy Landscape" font "Time New Roman,20"
set xlabel "PC1" font "Time New Roman,20"
set ylabel "PC2" font "Time New Roman,20"
#隐藏图例:
unset key
#这里必须根据实际数据进行修改:
set yrange [ -18.955: 22.277]
set xrange [ -25.709: 22.280]
#设置轴的小刻度线:
set mxtics 3
set mytics 3
#刻度线向外:
set tics out
#隐藏对称的刻度轴:
set tics nomirror........(表示数据)#输出:
set output
end
pause -1查看保存的pdf图片:
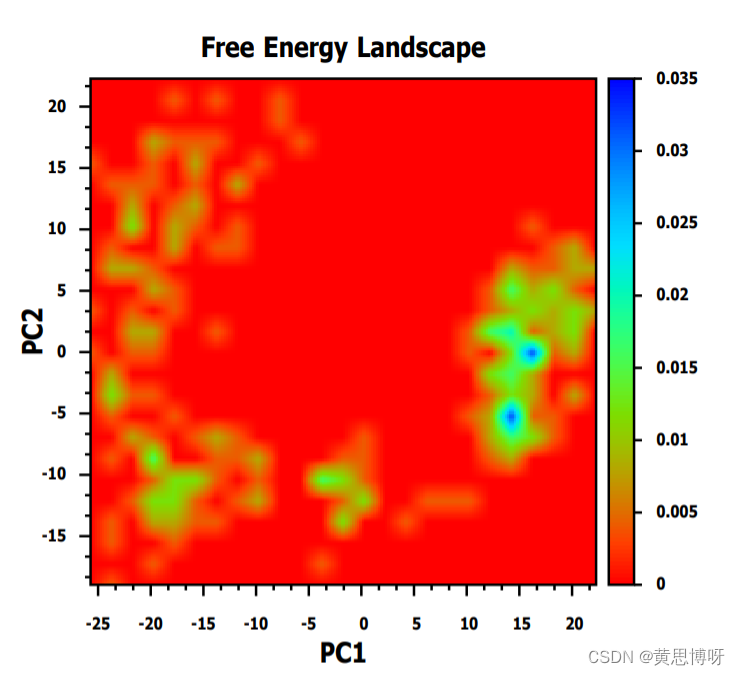
编写脚本将概率值转换为相对自由能,再绘制FEL图:
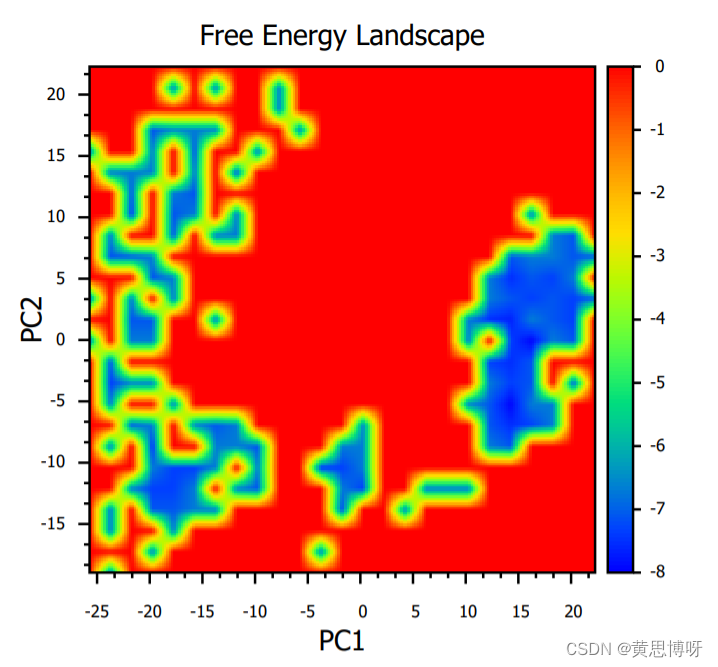
使用origin软件绘制自由能井图:
将数据文件pca-out.gnu改为pca-out.dat,将里面和gnuplot有关的命令语法删掉,只保留数据值,导入origin里:
将第3列数据表示强度的数据列改为Z轴:
对3列数据全选,在【工作表】-->【转化为矩阵】-->【xyz网格化】生成一个虚拟矩阵。
选择虚拟矩阵,选择【绘图】-->【等高线图】,调整调整
关于origin软件保存图片带有demo的水印的问题(提醒自己):
发现用修复工具之后,一段时间demo由会像蘑菇一样冒出来(F**k!),修复操作如下:
水印修复工具(就是那个Origin.exe文件),复制(不要移动)到安装目录里,替换里面原有的Origin.exe
打开安装目录下的Origin64.exe就能去掉水印啦。