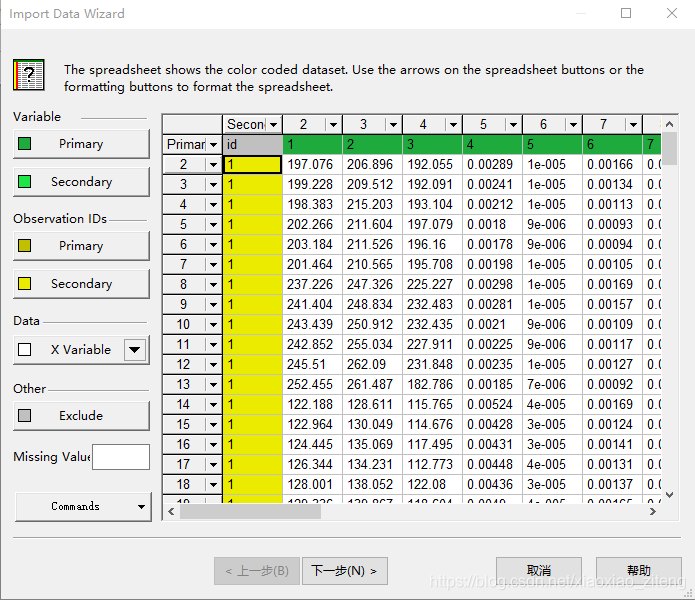
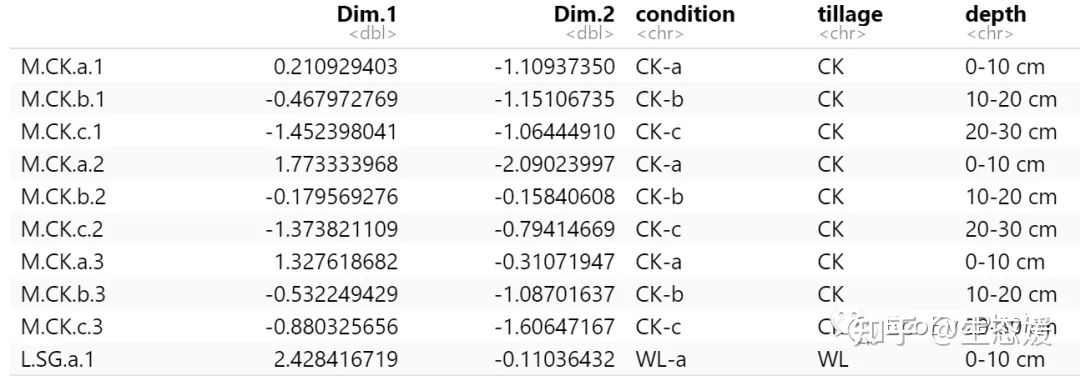
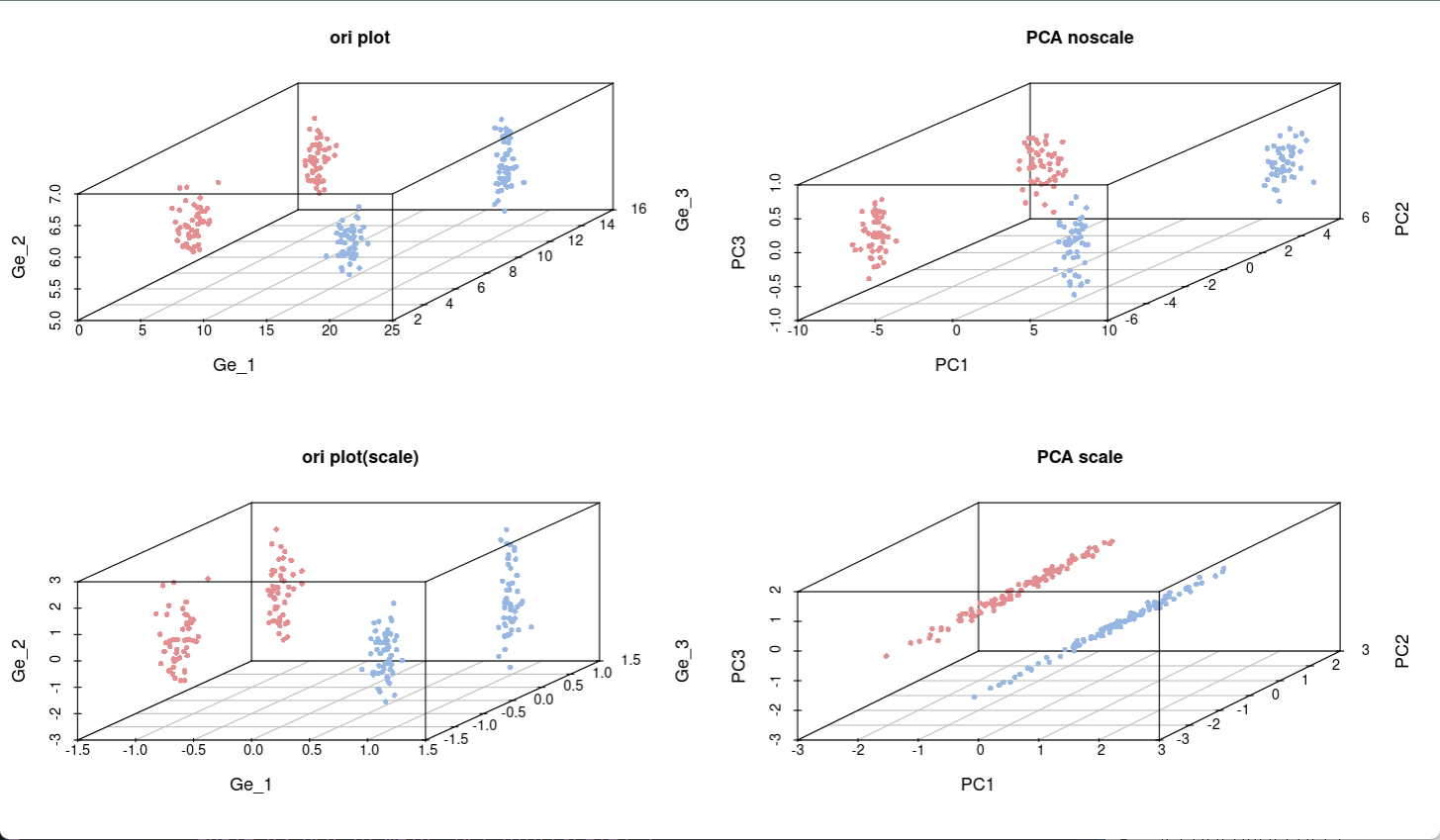
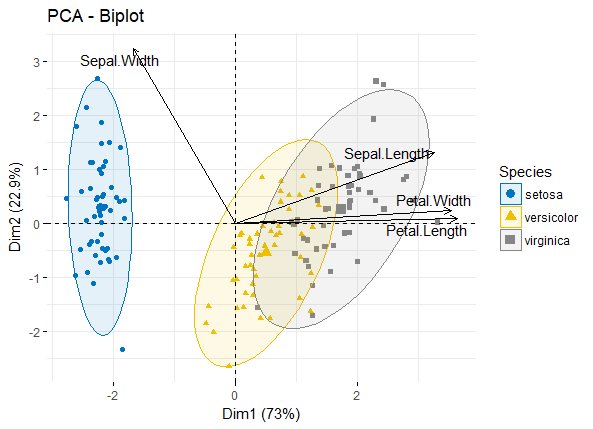
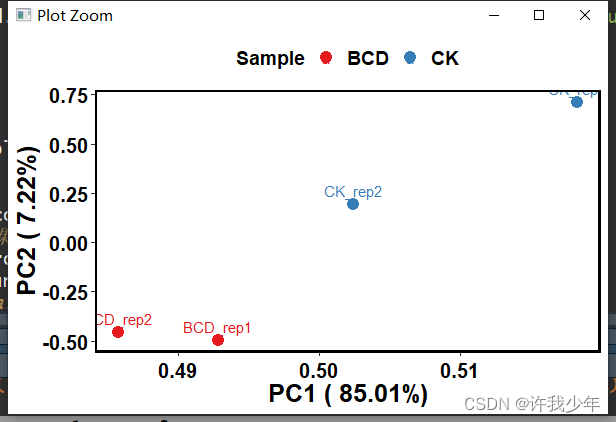
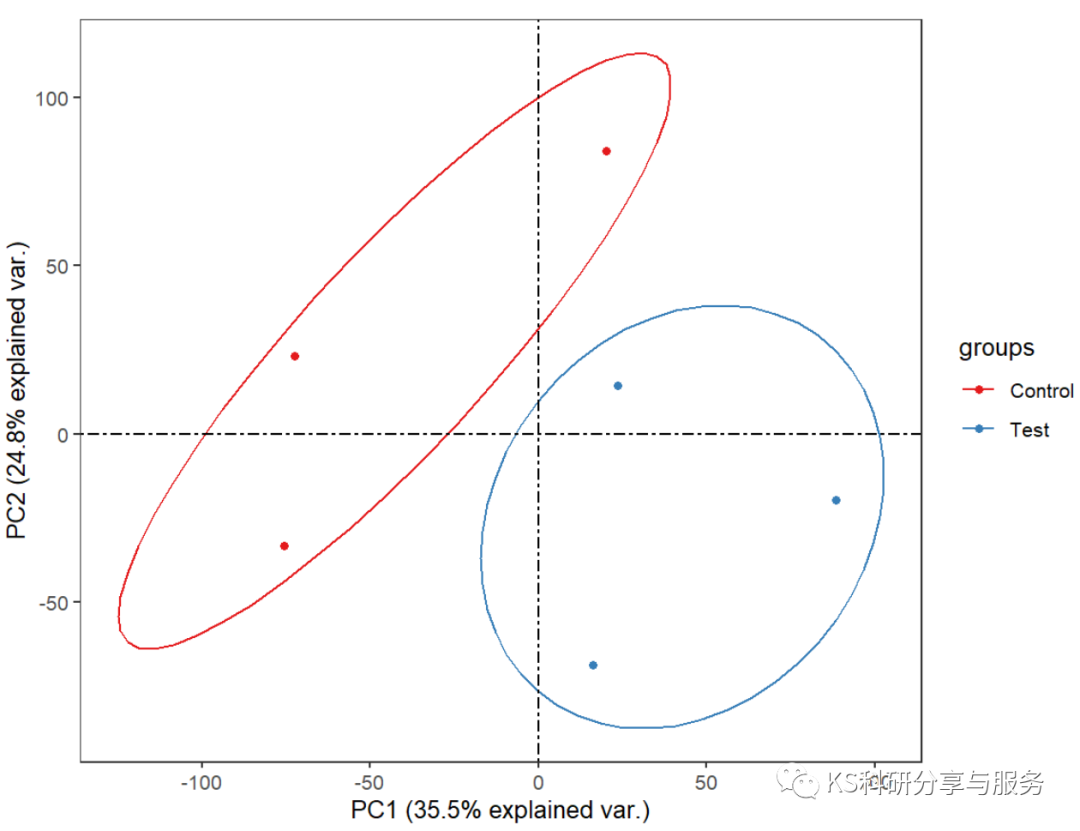
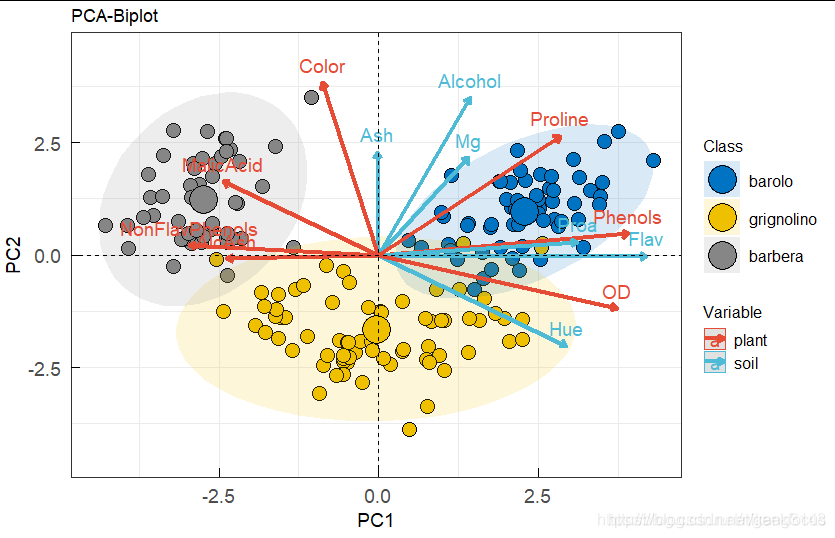
主成分分析(Principal component analysis, PCA)是一种数学算法,它可以在减少数据维度的同时,保留数据集中绝大多数的变量[1]。
PCA通过对主成分进行辨别,找出一个方向,并沿此方向分布的数据为最大值,从而减少数据维度。通过采用这样的主成分,便可以只选用若干变量而不是上千的变量来对一种样品进行分析了。
这样,就可以将样品有关变量绘制成图,使得样品间的相似性和相异之处一目了然,对不同样品是否可以归为一组,也一清二楚。这里我们主要解析如何利用R语言,基于表达谱中基因的表达值做PCA分析,并输出三维的PCA结果图。
1. 在线安装并加载rgl包
source("http://bioconductor.org/biocLite.R")biocLite(“rgl”)library(rgl)
2. 输入表达谱数据(示例以一组随意获得的表达谱中的表达谱值为例)
A=read.table("e:/diff-exp-zhuan.txt",header=T)#输入表达谱数据,注意这里基因数目一定不能少于样本数目B=as.data.frame(scale(A[2:21]))#指定参与做PCA分析的数据
3. 用之前指定