PCA分析步骤:
第一步,对所有样本进行中心化
第二步,求特征协方差矩阵
第三步,求协方差矩阵的特征值和特征向量
第四步,将特征值按照从大到小的顺序排序,选择其中最大的k个,然后将其对应的k个特征向量分别作为列向量组成特征向量矩阵
第五步,将样本点投影到选取的特征向量上(用欧氏距离方程计算点间距离)
getwd()#导入三张表gene_exp<-read.csv("gene_exp.csv",header = T,row.names = 1)
gene_info<-read.csv("gene_info.csv",header = T,row.names = 1)
sample_info<-read.csv("sample_info.csv",header = T,row.names = 1)#BiocManager::install("PCAtools")
library(PCAtools)一、样本相关性分析
sample_cor<-cor(gene_exp)#计算样本相关性
sample_cor2<-round(sample_cor,digits = 2)#保留两位小数
library(pheatmap)
pheatmap(sample_cor2)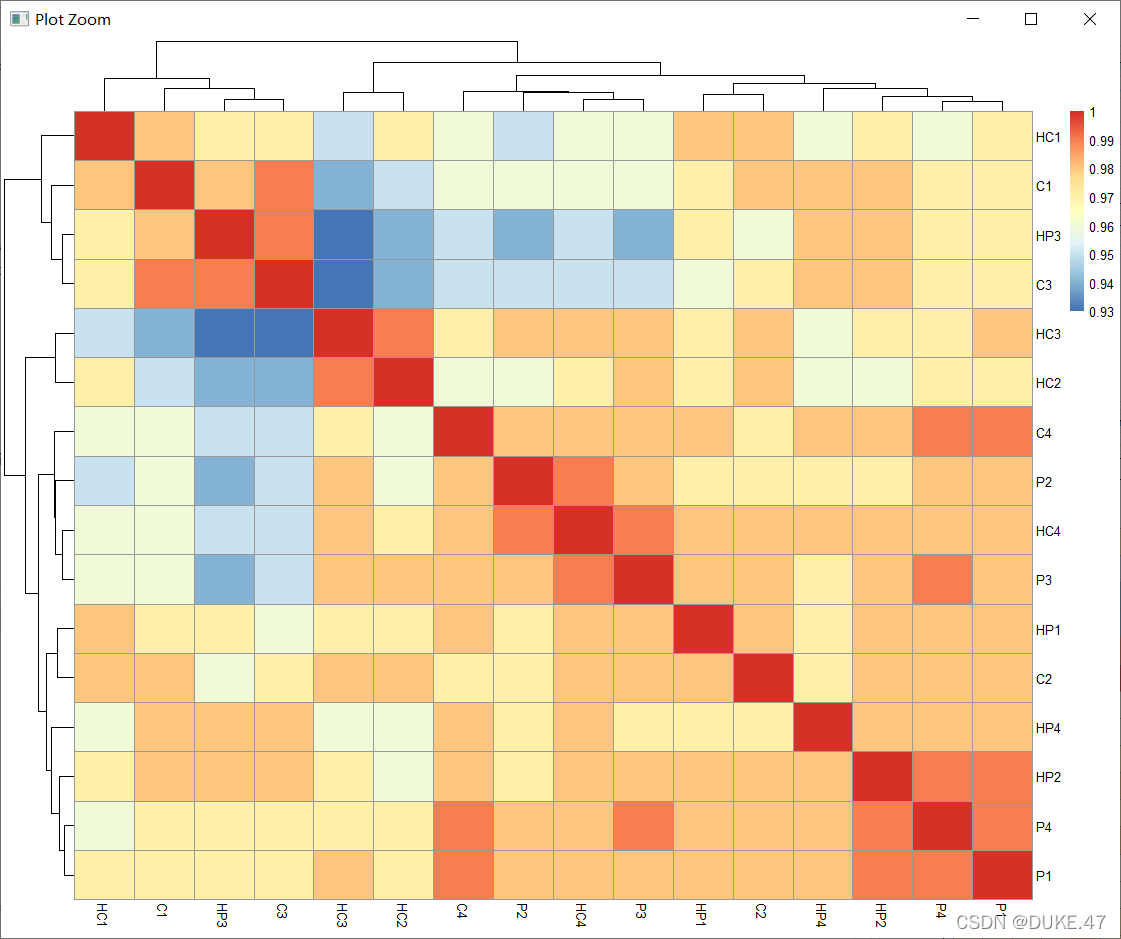
二、样本聚类
##计算距离矩阵
sample_dist<-dist(t(gene_exp))#先转置在计算距离矩阵##聚类
sample_hc<-hclust(sample_dist)
plot(sample_hc)
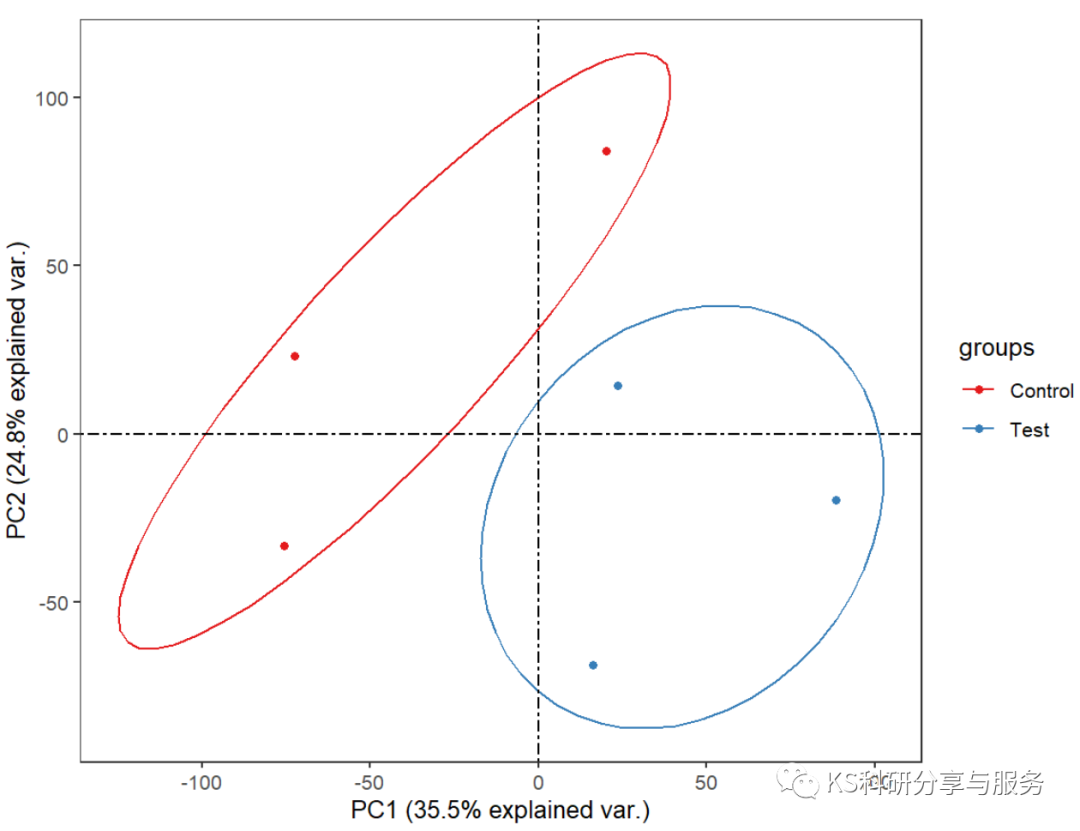
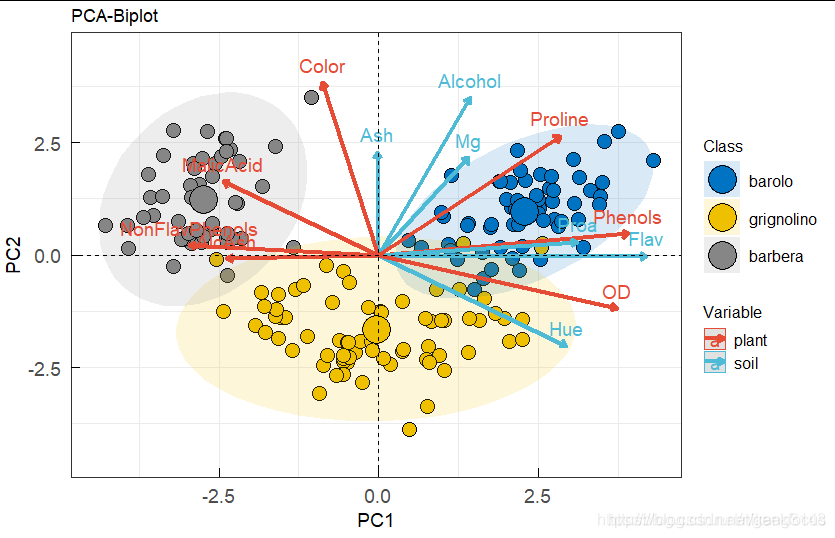
三、主成分分析
library(PCAtools)##主成分分析
pca<-pca(gene_exp,metadata=sample_info,removeVar = 0.1)#样本名字顺序要完全一致!
pca_loadings<-pca$loadings#提取loadings信息
pca_loadings[1:4,1:4]#打印出1-4行/1-4列
pca_rotated<-pca$rotated#提取rotated信息
pca_rotated[1:4,1:4]screeplot(pca)#画图:主成分对样本差异的解释度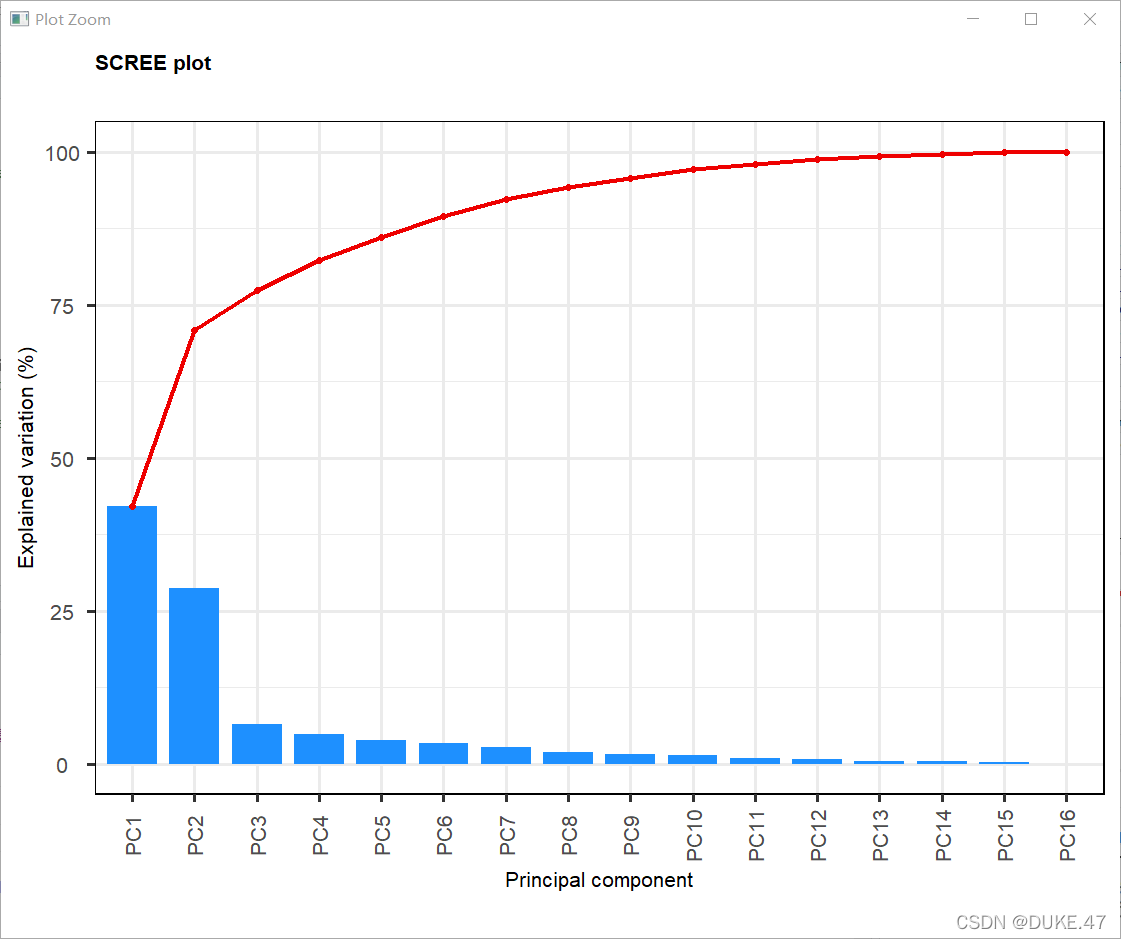
biplot(pca,#画图:PC1/PC2图x="PC1",y="PC2",#PC1/PC2可换shape = "group",)#形状=分组信息
plotloadings(pca)#查看哪些gene对PCA的贡献大