关注 M r . m a t e r i a l , \color{Violet} \rm Mr.material\ , Mr.material , 更 \color{red}{更} 更 多 \color{blue}{多} 多 精 \color{orange}{精} 精 彩 \color{green}{彩} 彩!
主要专栏内容包括:
†《LAMMPS小技巧》: ‾ \textbf{ \underline{\dag《LAMMPS小技巧》:}} †《LAMMPS小技巧》: 主要介绍采用分子动力学( L a m m p s Lammps Lammps)模拟相关安装教程、原理以及模拟小技巧(难度: ★ \bigstar ★)
††《LAMMPS实例教程—In文件详解》: ‾ \textbf{ \underline{\dag\dag《LAMMPS实例教程—In文件详解》:}} ††《LAMMPS实例教程—In文件详解》: 主要介绍采用分子动力学( L a m m p s Lammps Lammps)模拟相关物理过程模拟。(包含:热导率计算、定压比热容计算,难度: ★ \bigstar ★ ★ \bigstar ★ ★ \bigstar ★)
†††《Lammps编程技巧及后处理程序技巧》: ‾ \textbf{ \underline{\dag\dag\dag《Lammps编程技巧及后处理程序技巧》:}} †††《Lammps编程技巧及后处理程序技巧》: 主要介绍针对分子模拟的动力学过程(轨迹文件)进行后相关的处理分析(需要一定编程能力。难度: ★ \bigstar ★ ★ \bigstar ★ ★ \bigstar ★ ★ \bigstar ★ ★ \bigstar ★)。
††††《分子动力学后处理集成函数—Matlab》: ‾ \textbf{ \underline{\dag\dag\dag\dag《分子动力学后处理集成函数—Matlab》:}} ††††《分子动力学后处理集成函数—Matlab》: 主要介绍针对后处理过程中指定函数,进行包装,方便使用者直接调用(需要一定编程能力,难度: ★ \bigstar ★ ★ \bigstar ★ ★ \bigstar ★ ★ \bigstar ★)。
†††††《SCI论文绘图—Python绘图常用模板及技巧》: ‾ \textbf{ \underline{\dag\dag\dag\dag\dag《SCI论文绘图—Python绘图常用模板及技巧》:}} †††††《SCI论文绘图—Python绘图常用模板及技巧》: 主要介绍针对处理后的数据可视化,并提供对应的绘图模板(需要一定编程能力,难度: ★ \bigstar ★ ★ \bigstar ★ ★ \bigstar ★ ★ \bigstar ★)。
††††††《分子模拟—Ovito渲染案例教程》: ‾ \textbf{ \underline{\dag\dag\dag\dag\dag\dag《分子模拟—Ovito渲染案例教程》:}} ††††††《分子模拟—Ovito渲染案例教程》: 主要采用 O v i t o \rm Ovito Ovito软件,对 L a m m p s \rm Lammps Lammps 生成的轨迹文件进行渲染(难度: ★ \bigstar ★ ★ \bigstar ★)。
专栏说明(订阅后可浏览对应专栏全部博文): ‾ \color{red}{\textbf{ \underline{专栏说明(订阅后可浏览对应专栏全部博文):}}} 专栏说明(订阅后可浏览对应专栏全部博文):
注意: \color{red} 注意: 注意:如需只订阅某个单独博文,请联系博主邮箱咨询。 l a m m p s _ m a t e r i a l s @ 163. c o m \rm lammps\_materials@163.com lammps_materials@163.com
♠ \spadesuit ♠ † \dag † 开源后处理集成程序:请关注专栏《LAMMPS后处理——MATLAB子函数合集整理》
♠ \spadesuit ♠ † \dag † † \dag † 需要付费定制后处理程序请邮件联系: l a m m p s _ m a t e r i a l s @ 163. c o m \rm lammps\_materials@163.com lammps_materials@163.com
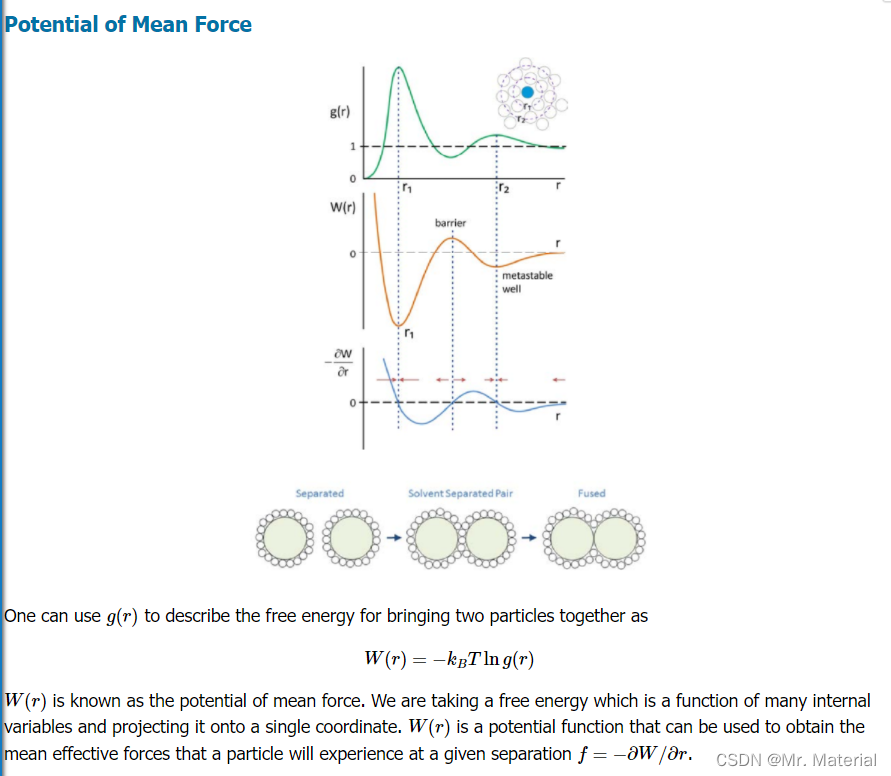
融化过程中RDF的变化
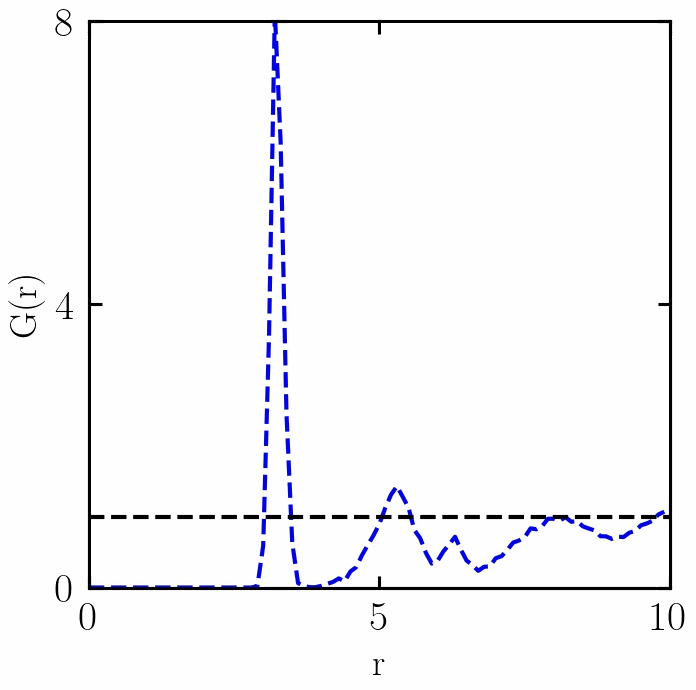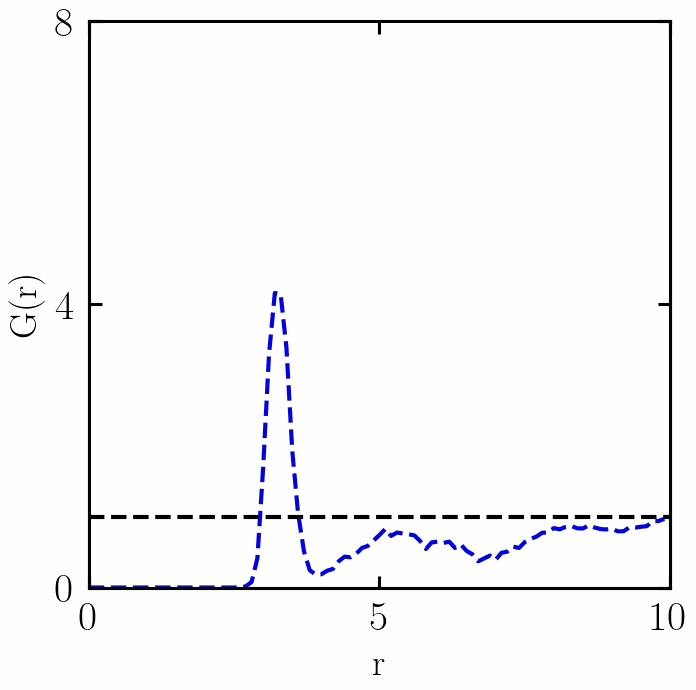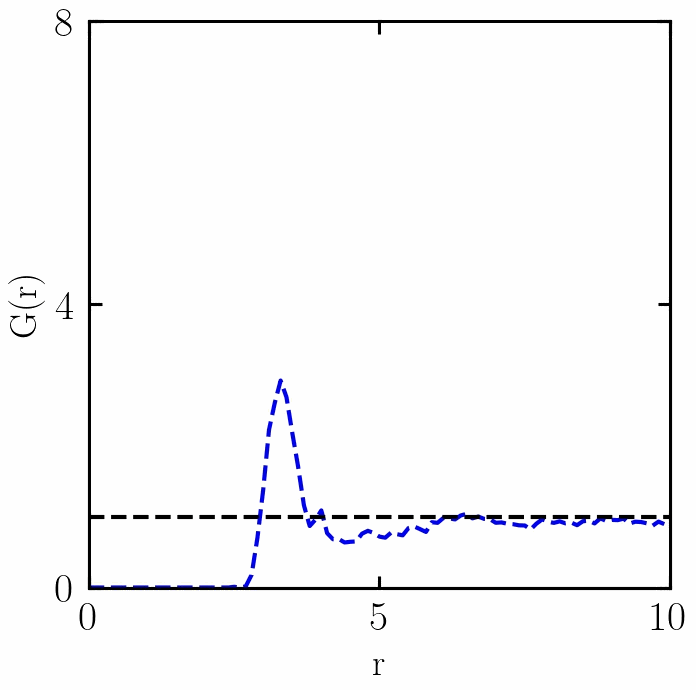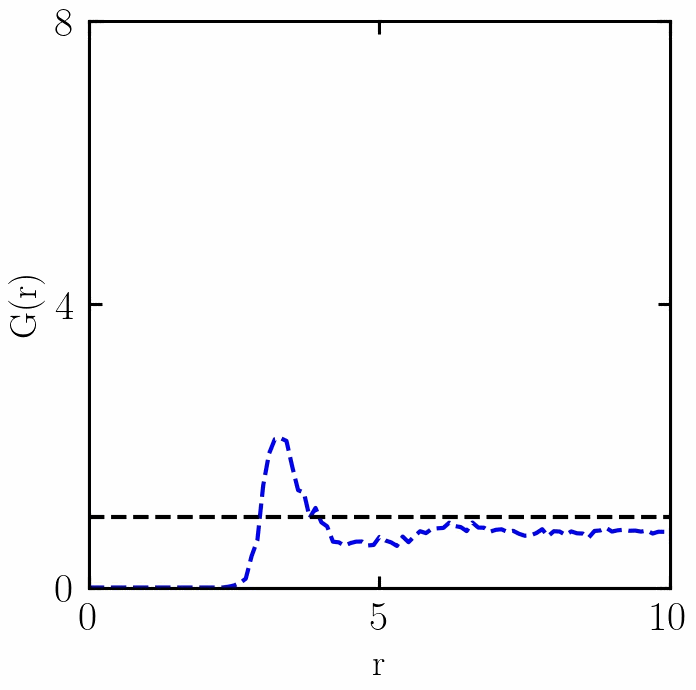
感谢 w e i x i n _ 50560116 weixin\_50560116 weixin_50560116朋友,提出两个错误:(1)已经更新了PBC函数,(2)t1if 改为 if (复制错误);
Lammps-如何采用MATLAB计算径向分布函数(RDF)
- 一. 读取dump
- 二. 计算RDF
- 三. 写出文件python画图
- 全部代码如下
下文翻译自:(请点击)
“结构”意味着粒子的定位是规则的和可预测的。考虑到粒子的短程位置和堆积,这在某种程度上是可能的。局部颗粒密度变化应显示统计平均意义上的某种结构。结构需要一个参考点,在流体的情况下,我们选择单个粒子作为参考,并描述其他粒子相对于该点的位置。由于流体的每个粒子都经历不同的局部环境,因此必须对这些信息进行统计平均,这是我们第一个相关函数的例子。对于大于“相关长度”的距离,我们应该失去预测特定粒子对相对位置的能力。在这个较长的尺度上,流体是均匀的。
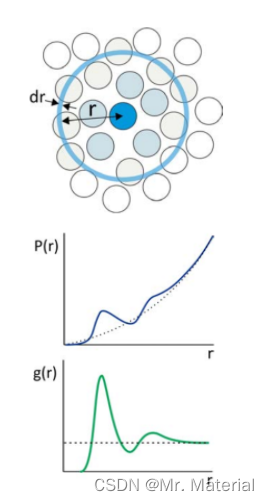
径向分布函数 g ( r ) g(r) g(r) 是分子长度尺度上流体“结构”的最有用的度量。尽管它引用了连续统描述,但“流体”指的是任何稠密、无序的系统,其组成粒子的位置存在局部变化,但在宏观上是各向同性的。 g ( r ) g(r) g(r) 通过描述中心参考粒子周围粒子的平均分布,提供了系统的局部堆积和粒子密度的统计描述。我们将径向分布函数定义为 ⟨ ρ ( r ) ⟩ ⟨ρ(r)⟩ ⟨ρ(r)⟩, 距离r处颗粒的平均局部数密度与颗粒的体积密度 ρ \rho ρ :
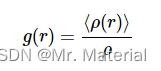
在稠密系统中, g ( r ) g(r) g(r) 从零开始(因为它不计算参考粒子),在表征参考粒子周围的第一层粒子(即第一溶剂化层)的距离处上升到峰值,并且在各向同性介质中长距离接近 1。在厚度为dr的壳中距离r处发现粒子的概率为 P ( r ) = 4 π r 2 g ( r ) d r P(r)=4πr2g(r)dr P(r)=4πr2g(r)dr,因此对 ρ ⋅ g ( r ) ρ⋅g(r) ρ⋅g(r)在中的第一个峰上给出了第一个壳中粒子的平均数量。
径向分布函数最常用于气体、液体和溶液,因为它可以用于计算热力学性质,如系统的内能和压力。但在任何大小的尺度上都是相关的,例如胶体的堆积,并且在复杂的异质介质中是有用的,例如 D N A DNA DNA 周围的离子分布。为了关联不同类型粒子的位置,径向分布函数被定义为距离 “ a ” “a” “a” 粒子 r r r 处 “ b ” “b” “b” 粒子的局部密度之比 g a b ( r ) = ⟨ ρ a b ( r ) ⟩ / ρ g_{ab}(r) = ⟨ρ_{ab}(r)⟩/ρ gab(r)=⟨ρab(r)⟩/ρ 在实践中, ρ a b ( r ) ρ_ab(r) ρab(r) 是通过从距离为 r r r 且厚度为 d r dr dr 的壳层上的 “ a ” “a” “a” 粒子径向观察,计算该壳层内 “ b ” “b” “b” 粒子的数量,并用该壳层的体积归一化该数量来计算的。
让我们看得更深一点,考虑到相同类型的粒子,如原子液体或颗粒材料中的粒子。如果体积V中有 N N N 个粒子,且第 i t h i^{th} ith 个粒子的位置为 r i ‾ \overline{r_{i}} ri,则数密度描述粒子的位置,
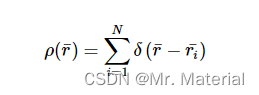
X ( r ) X(r) X(r) 给出的径向变化特性的平均值由下式确定:
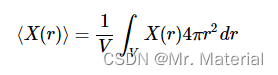
在一个体积上积分 ρ ( r i ) ‾ \overline{\rho(r_{i})} ρ(ri) 可以得到该体积中的粒子数。
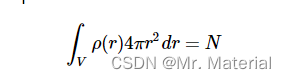
当积分在整个体积上时,我们可以使用它来获得平均粒子密度:
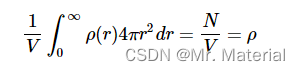
接下来,我们可以考虑两个粒子 i i i 和 j j j 之间的空间相关性

这描述了在位置 r i r_{i} ri 处找到粒子 i i i 和在位置 r j r_{j} rj 处找到粒子 j j j 的条件概率。根据 i = j i=j i=j 还是 i i i,我们可以将 ρ ( r ‾ , r ⃗ ′ ) \rho(\overline{r} ,\vec{r}^{\prime}) ρ(r,r′) 展开并因子化为两项 i = j i=j i=j or i i i 不等于 j j j:
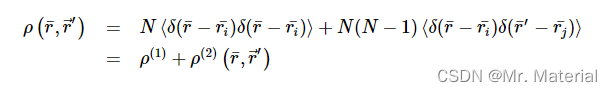
第一项描述了自相关,其中有N项:每个原子一项。

第二项描述了两种身体相关性,其中有N(N‐1)项。
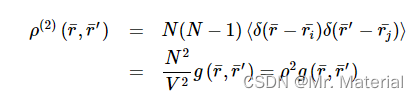
g ( ) = ρ ( 2 ) ( r ‾ , r ⃗ ′ ) / ρ 2 g()=\rho^{(2)}(\overline{r} ,\vec{r}^{\prime})/\rho^{2} g()=ρ(2)(r,r′)/ρ2 是双粒子分布函数,描述了两个原子或分子之间的空间相关性。对于各向同性介质,它仅取决于粒子之间的距离, g ( ∣ r ‾ , r ⃗ ′ ∣ ) = g ( r ) g(\left | \overline{r} ,\vec{r}^{\prime} \right |) =g(r) g(∣r,r′∣)=g(r),因此也称为径向对分布函数。
我们可以通过写 g a b ( r ) g_{ab}(r) gab(r)将 g ( r ) g(r) g(r) 推广为 a a a 和 b b b 粒子的混合物:
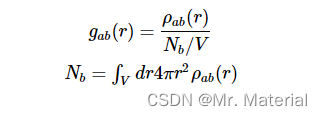
一. 读取dump
读取dump文件,请见博客:
python 读取:https://blog.csdn.net/qq_43689832/article/details/113812304?spm=1001.2014.3001.5502
matlab读取:https://blog.csdn.net/qq_43689832/article/details/108943190?spm=1001.2014.3001.5501
C++ 读取:https://blog.csdn.net/qq_43689832/article/details/104686674
二. 计算RDF
%=========================================================%
bin = 100;
cutoff = 5;
%=========================================================%
bin_size = cutoff/bin; calcu_data = atom_data;
all_frame = size(atom_data,3);Gr = zeros(bin,all_frame);
N = length(atom_data);vol = xl*yl*zl;
globel_density =N/vol;
%----------------------------------------------------------%for frame = 1:30:all_framenow_frame = calcu_data(:,:,frame);xl = x_bound(1,2)-x_bound(1,1);yl = y_bound(1,2)-y_bound(1,1);zl = z_bound(1,2)-z_bound(1,1);type = now_frame(:,2);XYZ = now_frame(:,3:5);for ii = 1:Nif(1)for jj = 1:Nif(ii~=jj)dx = XYZ(jj,1)-XYZ(ii,1);dy = XYZ(jj,2)-XYZ(ii,2);dz = XYZ(jj,3)-XYZ(ii,3);PBC(dx,dy,dz,xl,yl,zl);dis = dx^2+dy^2+dz^2;if(sqrt(dis)<cutoff)index = ceil(sqrt(dis)/bin_size); Gr(index,frame) = Gr(index,frame) + 1;endendendendfor kk = 1:bindV = 4 * pi * (bin_size * kk)^2 * bin_size; Gr(kk,frame) =Gr(kk,frame)./N./dV./globel_density;end
end
%----------------------------------------------------------%
Gr(:,all(Gr == 0,1)) =[];
%----------------------------------------------------------%
%pbc
function [dx,dy,dz] = PBC(dx,dy,dz,xl,yl,zl)
%-------------------------------------------------%if (dx>xl/2)dx = dx - xl;elseif (dx<(-xl/2))dx = dx + xl;endif (dy>yl/2)dy = dy - yl;elseif (dy<(-yl/2))dy = dy + yl;endif (dz>zl/2)dz = dz - zl;elseif (dz<(-zl/2))dz = dz + zl;end
%-------------------------------------------------%
end三. 写出文件python画图
import os
def make_save_file(filepath,filename,savename):# filepath 存储路径; filename:创建文件夹的名字 savename:存储图片的名字save_path = filepath+os.sep+filename+os.sep+savenameall_path = filepath+os.sep+filenameif not os.path.exists(all_path):os.mkdir(all_path)plt.savefig(save_path,dpi=150)
filepath = r"save path"
font1 = {'family': 'Times New Roman','weight': 'bold','size': 22,}
font2 = {'family': 'Times New Roman','weight': 'normal','size': 11,}
# Global setting# ----------------------------------------------------------------------#
import matplotlib.pyplot as plt
import pandas as pd
import numpy as np
# coding:utf-8
import pylab
import matplotlib.ticker as plticker
from matplotlib.pyplot import MultipleLocator
import xlrd
import matplotlib.pyplot as plt
from matplotlib.lines import Line2D
import math
def func(x,beita_tar):return 0.022/(1+2*x*beita_tar)plt.style.use('science')
# ----------------------------------------------------------------------#
def setup(ax,x_label,y_label):# 边框线的线宽设置width = 1.5ax.spines['top'].set_linewidth(width)ax.spines['bottom'].set_linewidth(width)ax.spines['left'].set_linewidth(width)ax.spines['right'].set_linewidth(width)# 边框上的ticks的出现ax.tick_params(top='on', bottom='on', left='on', right='on', direction='in')# 边框上的ticks对应的lable,即数字的尺寸ax.tick_params(labelsize=20,pad=7)#ax.yaxis.set_ticks_position('right')ax.tick_params(which='major', width=1.50, length=6)ax.tick_params(which='minor', width=0.75, length=0)# 边框上的ticks的出现的间隔locx = plticker.MultipleLocator(base=5) # this locator puts ticks at regular intervalsax.xaxis.set_major_locator(locx)locy = plticker.MultipleLocator(base=4) # this locator puts ticks at regular intervalsax.yaxis.set_major_locator(locy)# 边框上的注释labelslabels = ax.get_xticklabels() + ax.get_yticklabels()[label.set_fontname('Times New Roman') for label in labels]# 边框上的注释labels距离坐标轴的距离xAxisLable = x_label yAxisLable = y_labelax.set_xlabel(xAxisLable, font1, labelpad=5)ax.set_ylabel(yAxisLable, font1, labelpad=5)
# ----------------------------------------------------------------------#
# 读取excel
excel_path = r"\rdf1.xlsx"
work_sheet = xlrd.open_workbook(excel_path);# sheet的数量
sheet_num = work_sheet.nsheets;
# 获取sheet name
sheet_name = []
for sheet in work_sheet.sheets():sheet_name.append(sheet.name)
# ----------------------------------------------------------------------#
# ----------------------------------------------------------------------#
#fig, axe = plt.subplots(1, 1, figsize=(5,5))
for sheet_num in range(100):# 选取sheetnow_sheet = work_sheet.sheets()[0]#now_sheet_ref = work_sheet.sheets()[3]# sheet的行row = now_sheet.nrows# sheet的列ncol = now_sheet.ncols#----------------------------------------------------------------------##----------------------------------------------------------------------#font1 = {'family': 'Times New Roman','weight': 'bold','size': 18,}font2 = {'family': 'Times New Roman','weight': 'normal','size': 11,}#----------------------------------------------------------------------# # stepy1 = now_sheet.col_values(sheet_num)y1_matrix = y1[0:]# tempx = np.arange(100)# stepy2 = now_sheet.col_values(sheet_num)y2_matrix = y2[0:]# press#----------------------------------------------------------------------#markersize = 10 linewidth = 1markevery =1p_1 = dict(marker='o',color = 'r',linestyle = 'none',markersize=markersize,markevery=1, linewidth=linewidth,alpha=0.5)p_2 = dict(color = 'r', linestyle = '-.',linewidth=linewidth)p_3 = dict(color = 'b', linestyle = '--',linewidth=2)p_33 = dict(color = 'r', linestyle = '--',linewidth=2)p_4 = dict(color = 'c', linestyle = '-',linewidth=linewidth)p_5 = dict(color = 'm', linestyle = '-',linewidth=linewidth)p_6 = dict(color = 'y', linestyle = '-',linewidth=linewidth)p_7 = dict(color = 'k', linestyle = '-',linewidth=linewidth)p_8 = dict(color = 'tab:orange',linestyle = '-',linewidth=linewidth)p_9 = dict(color = 'tab:pink',linestyle = '-',linewidth=linewidth) p_10 = dict(color = 'tab:pink',linestyle = '-',linewidth=linewidth)#----------------------------------------------------------------------#p_fit = dict(color = 'b',linestyle = '--',lw=2)fig, axe = plt.subplots(1, 1, figsize=(5,5))skip=3#axe.scatter(x1_matrix, y1_matrix, **p_s_1,label=legend_1)#x1_fit = np.arange(0.3,1.8,0.01)#y1_fit = 156*exp(-((x1_fit-0.11)/0.94)**2)#y1_fit = 156.9*np.exp(-1.16*(x1_fit-0.11)**2)#axe.plot(x, y1_matrix,**p_2,label='Number density')axe.plot(x*np.array(0.1), y2_matrix,**p_3)#axe.plot(x*np.array(0.1),np.array(y2_matrix).sum(axis=0)/np.array(100),**p_3)plt.axhline(1, color='k', linestyle='--',lw=2)#axe.axvline(x=5000,ymin=-6.5,ymax=-5)#plt.axvline(x=1700,color='k',linestyle='dashed')#axe.plot(np.arange(len(y10_matrix))+1,np.array(y10_matrix)+45,label=legend_10,**p_10)axe.set_ylim(0, 8)axe.set_xlim(0, 10)setup(axe,r'$\rm r$',r'$\rm G(r)$')#axe.legend(loc='best', frameon=False, \# labelspacing=0.3)#axe.legend(bbox_to_anchor=(1.10, 1), \# loc='upper left', borderaxespad=0.,fontsize='xx-large')#axe[1].legend(loc='best', frameon=False, labelspacing=0.3)#axe[1].legend(bbox_to_anchor=(1.10, 1), loc='upper left', borderaxespad=0.,fontsize='x-large')# ============================================================#filename = "rdf"savename = str(sheet_num)+'.jpg'make_save_file(filepath,filename,savename)#figureFileName = "Amorphous_overlop.pdf"#print(figureFileName)#plt.savefig(figureFileName, dpi=300)#plt.xscale("log")#plt.yscale("log")# ----------------------------------------------------------------------#plt.show()
全部代码如下
%{
This code is to calculate RDF by LAMMPS dump file
The origin code source is from (see below)
https://github.com/brucefan1983/Molecular-Dynamics-Simulation/tree/master/src/06-properties/rdf
This code is to calculate the each frame from dump file
%}clc;clear;
%=========================================================%
file ="dump.all";
bin = 100; % be carefull to select the bin value based on your simulation system
cutoff = 5; %
%=========================================================%
t_start_all = cputime;
trydump = fopen(file,'r');
catcherror('Dumpfile not found!');
endi=1;
while feof(dump) == 0id = fgetl(dump);if (strncmpi(id,'ITEM: TIMESTEP',numel('ITEM: TIMESTEP')))timestep(i) = str2num(fgetl(dump));elseif (strncmpi(id,'ITEM: NUMBER OF ATOMS',numel('ITEM: NUMBER OF ATOMS')))Natoms(i) = str2num(fgetl(dump));elseif (strncmpi(id,'ITEM: BOX BOUNDS',numel('ITEM: BOX BOUNDS')))x_bound(i,:) = str2num(fgetl(dump));y_bound(i,:) = str2num(fgetl(dump));z_bound(i,:) = str2num(fgetl(dump));elseif (strcmpi(id(1:11),'ITEM: ATOMS'))for j = 1 : 1: Natomsatom_data(j,:,i) = str2num(fgetl(dump));endi=i+1;endend endend
end
t_start_stop = (cputime-t_start_all)/60;
fprintf('Now the used time is: %.1f mins.\n',t_start_stop);
disp("-------------------");
disp("----ALL DONE!!!----");
disp("-------------------");
% 这个是自动关机-
% system('shutdown -s');
bin_size = cutoff/bin; calcu_data = atom_data;
all_frame = size(atom_data,3);Gr = zeros(bin,all_frame);
N = length(atom_data);vol = xl*yl*zl;
globel_density =N/vol;
%----------------------------------------------------------%for frame = 1:30:all_framenow_frame = calcu_data(:,:,frame);xl = x_bound(1,2)-x_bound(1,1);yl = y_bound(1,2)-y_bound(1,1);zl = z_bound(1,2)-z_bound(1,1);type = now_frame(:,2);XYZ = now_frame(:,3:5);for ii = 1:Nif(1)for jj = 1:Nif(ii~=jj)dx = XYZ(jj,1)-XYZ(ii,1);dy = XYZ(jj,2)-XYZ(ii,2);dz = XYZ(jj,3)-XYZ(ii,3);PBC(dx,dy,dz,xl,yl,zl);dis = dx^2+dy^2+dz^2;t1if(sqrt(dis)<cutoff)index = ceil(sqrt(dis)/bin_size); Gr(index,frame) = Gr(index,frame) + 1;endendendendfor kk = 1:bindV = 4 * pi * (bin_size * kk)^2 * bin_size; Gr(kk,frame) =Gr(kk,frame)./N./dV./globel_density;end
end
%----------------------------------------------------------%
Gr(:,all(Gr == 0,1)) =[];
%----------------------------------------------------------%