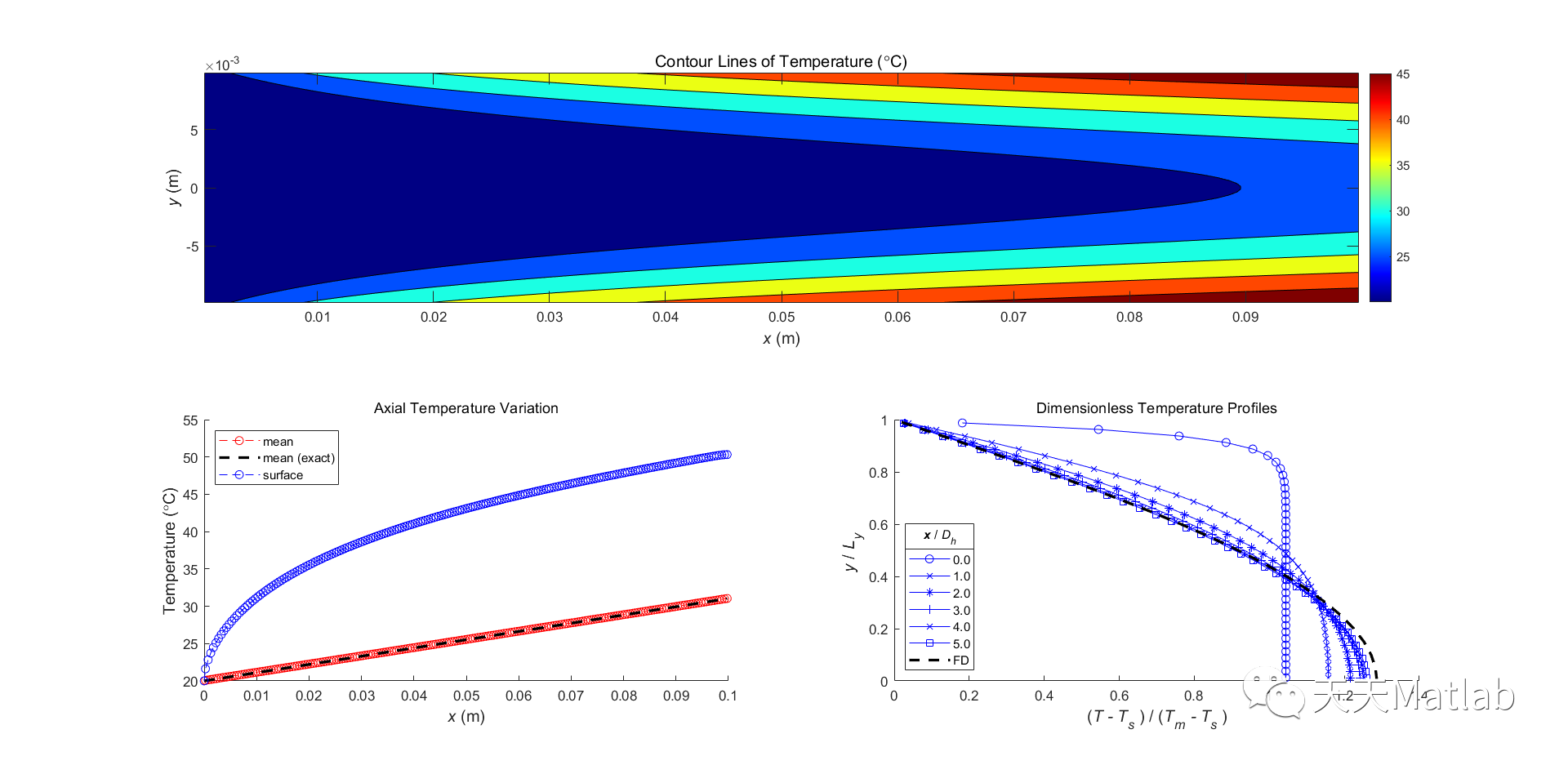
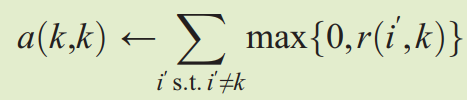第14-16讲 真核生物基因表达调控
文章目录
- 10. 真核生物基因表达调控
- 10.1 转录水平的调控 (transcriptional regulation)
- 10.1.1 转录起始调控 Transcriptional initiation
- 10.1.2 组蛋白修饰
- 10.1.3 DNA 甲基化
- 10.2 转录后水平的调控 (post-transcriptional regulation)
- 10.2.1 前体 RNA 加工
- 10.2.2 mRNA 转运
- 10.2.3 翻译
- 10.2.4 RNA 降解
- 10.2.6 蛋白质降解
10. 真核生物基因表达调控
● 真核生物与原核生物的差别
- 原核生物:单细胞;基因组织方式——操纵子;调控——短期转录。
- 真核生物:
蛋白或者多细胞;稀少的操纵子;
调控——短期转录和长期调控;
目标:在不同的时间不同的细跑中协调新蛋白的产生。
● 真核细胞与原核细胞在基因转录,翻译及 DNA 的空间结构方面存在如下差异:
(1) 在真核细胞中,一条成熟的 mRNA 链只能翻译出一条多肽链,原核生物中常见的多基因操纵子形式在真核细胞中比较少见。
(2) 真核细胞的 DNA 与组蛋白和大量非组蛋白结合,只有一小部分 DNA 是裸露的。
(3) 高等真核细胞 DNA 中大部分不转录,真核细胞中有一部分由几个或几十个碱基组成的 DNA 序列,在整个基因组中重复几百次甚至上百万次。此外,大部分真核细胞的基因中间还存在不被翻译的内含子。
(4) 真核生物能够有序地根据生长发育阶段的需要进行 DNA 片段重排,还能在需要时增加细胞内某些基因的拷贝数,这种能力在原核生物中也是极为鲜见的。
(5) 在原核生物中,转录的调节区都很小,大都位于转录起始位点上游不远处,调控蛋白结合到调节位点上可直接促进或抑制 RNA 聚合酶对它的结合。在真核生物中,基因转录的调节区则大得多,它们可能远离核心启动子几百个甚至上千个碱基对。虽然这些调节区也能与蛋白质结合,但是并不直接影响启动子区对于 RNA 聚合酶的接受程度,而是通过改变整个所控制基因 5’上游区 DNA 构型来影响它与 RNA 聚合酶的结合能力。
(6) 真核生物的 RNA 在细胞核中合成,只有经转运穿过核膜,到达细胞质基质后,才能被翻译成蛋白质。原核生物中不存在这样严格的空间间隔。
(7) 许多真核生物的基因只有经过复杂的成熟和剪接过程,才能被顺利地翻译成蛋白质。
10.1 转录水平的调控 (transcriptional regulation)
- 转录调控决定一个基因是否转录和转录的速度
- 与原核基因表达调控类似,真核基因的表达调控主要是转录调控
● 真核生物转录水平调控的特点
- 核小体结构及其修饰影响调控蛋白与 DNA 的结合和相互作用
- 与原核生物相比,真核生物 DNA 上具有更多的调控结合位点和更多的调控蛋白
10.1.1 转录起始调控 Transcriptional initiation
● 转录起始调控元件:
1)RNA 聚合酶 (RNA Polymerase):
- 真核生物有 3 种 RNA 聚合酶,催化转录不同 RNA 产物。
- 只有 RNA 聚合酶Ⅱ能够转录 mRNA 前体,并在加工成熟后按照三联子密码的原理翻译成蛋白质产物,因此,主要讨论 RNA 聚合酶Ⅱ的基因转录及其调控过程。
- 启动子,调节序列和调节蛋白通过 DNA-蛋白质相互作用、蛋白质-蛋白质相互作用影响 RNA 聚合酶活性。
2)顺式作用元件 (cis-acting elements): 基因组中含有可以调控自身基因表达活性的特异 DNA 序列。promoters/regulatory sequences of genes. 启动子和基因的调节序列。包括:启动子、增强子、沉默子等。
3)反式作用因子 (trans-acting factors): proteins (and RNAs) that bind cis-elements and promote or repress gene expression. 能够结合在顺式作用元件上调控基因表达的蛋白质或 RNA。
RNA 聚合酶Ⅱ
RNA 聚合酶Ⅱ是一类能够直接或间接与启动子核心序列 TATA 区特异结合,并启动转录的调节蛋白。- RNA 聚合酶Ⅱ在转录因子帮助下,形成转录起始复合物。
- RNA 聚合酶Ⅱ由至少 10~12 个亚基组成,其中 2.4×105 最大亚基的羧基末端含有由 7 个氨基酸残基 (
Tyr-Ser-Pro-Thr-Ser-Pro-Ser) 组成的多磷酸化位点重复序列,称为羧基末端结构域 (CTD)。 - 在生理条件下,RNA 聚合酶Ⅱ转录某个基因时通常需要与 20 种以上的蛋白质因子(以 TFⅡ表示)结合形成转录起始复合物。TFⅡD、TFⅡB 和 TFⅡF 与 RNA 聚合酶Ⅱ可在启动子上形成最初级复合物,开始转录 mRNA,加入 TFⅡE 和 TFⅡH 后形成完整的转录复合物并转录出长链 RNA,加入 TFⅡA 可进一步提高转录效率。RNA 聚合酶Ⅱ沿模板滑动时,TFⅡD 及 TFⅡA 滞留在转录起始位点上,其他因子随聚合酶向模板 DNA 的 3’端移动。
● RNA 聚合酶Ⅱ如何整合成为转录起始复合物,有两种假说:

① “一步结合”论 one-step:RNA 聚合酶Ⅱ先同大量的转录因子和转录相关蛋白结合成转录复合体,然后在 TATA 区结合蛋白 (TBP) 的帮助下结合到 DNA 上,起始基因转录。
② “分步结合”理论 multiple-step:顺序组装
- TFⅡD (TBP + 11 TAF, 800KD)与 TATA 区结合是启动的第一步。
TBP: TATA binding protein TATA 区结合蛋白
TAFs: TBP associated factors TBP 相关因子 - TFⅡB 与 DNA 结合并在 RNA 出口位点和活性中心附近与 RNA 聚合酶接触,并将其定向在 DNA 上。
CTD: RNA PolⅡ C 末端结构域
- 开始转录时需要通过 TFⅡH 的激酶活性使 CTD 磷酸化以释放 RNA 聚合酶。
- TFⅡE 和 TFⅡH 融化 (melt) DNA 以使聚合酶移动。
- CTD(通过 TFⅡH 和其他激酶)的磷酸化是延长开始启动 PolⅡ 所必需的。
- CTD 可以协调 RNA 与转录的加工。
- 在真核细胞中,CTD 在空间和时间上进行精确调控基因的转录。
- CTD 含 25~ 52 个氨基酸,都包含 7 元重复序列 Y1S2P3T4S5P6S7,这个 7 元重复序列上 2 位和 5 位的丝氨酸是蛋白质的主要磷酸化位点。
- CTD 重复序列上的磷酸化导致其结构中部分脯氨酸的构象发生变化,使之更容易与其他转录相关因子结合(磷酸化组合方式不同,招募的转录因子不同),从而提供一个动态的有利于形成转录复合物的平台。
- S2和S5:转录调控的触发器。在 RNA 聚合酶Ⅱ参与转录调节的整个过程中,其 CTD 上 2 位和 5 位丝氨酸的磷酸化能够引发并确保转录过程各个步骤的顺利进行。
① 转录起始阶段:CTD 上 5 位丝氨酸的磷酸化 PhosphoS5 是形成转录起始复合物PIC(pre-initiation complex) 的必要前提。并且通过募集加帽酶促进 mRNA 加帽。
② 转录延伸阶段 Elongation:PhosphoS5 逐步去磷酸化,S2 逐步磷酸化。
③ 转录终止过程 Terminating:PhosphoS2 通过触发招募 3’-RNA 加工机器确保高效的 3’-RNA 加工。
④ 转录结束 Ending:CTD 不含磷酸基团,有助于转录复合物的解离,使 RNA 聚合酶Ⅱ与新的启动子相结合,启动新一轮基因转录。
顺式作用元件

● 顺式作用元件能够被转录调节蛋白特异识别和结合,从而影响基因表达活性。
- 核心启动子元件(Core promoter elements):结合 RNAPⅡ和 GTF(general TF)。
- 调控元件(Regulatory Elements):增强子 (enhancer)、沉默子 (silencer): 结合调节蛋白
★ 核心启动子元件(Core promoter elements)
-
真核基因启动子由核心启动子和上游启动子两个部分组成,是在基因转录起始位点 (+1) 及其 5’上游 100~ 200 bp 以内的一组具有独立功能的 DNA 序列,每个元件长度为 7~20bp, 是决定 RNA 聚合酶Ⅱ转录起始位点和转录频率的关键元件。
-
(1) 核心启动子 (core promoter) 是指保证 RNA 聚合酶Ⅱ转录正常起始所必需的、最少的 DNA 序列,包括转录起始位点及转录起始位点上游 -25 ~ -30 bp 处的 TATA 区。核心启动子单独起作用时,只能确定转录起始位点并产生基础水平的转录。

| BRE: TFⅡB recognition element | TATA: TATA Box | TFⅡ: transcription factor for Pol Ⅱ |
|---|---|---|
| TBP: TATA binding protein | Inr: initiator element | DPE: Downstream promoter element |
- (2) 上游启动子元件 (upstream promoter element,
UPE) 包括通常位于 -70 bp 附近的 CAAT 区 (CCAAT) 和 GC 区 (GGGCGG) 等,能通过 TFⅡD 复合物调节转录起始的频率,提高转录效率。
● 调控元件(Regulatory Elements)
- 通常位于核心启动子上游,对于体内有效的转录是必须的。
- 可以分为几类:
① 启动子近端元件;
② 上游激活序列 (UASs, 增强子)
③ 一系列抑制元件(沉默子)
④ 界面元素 (boundary elements)
⑤ 绝缘体 (insulators) - 所有这些 DNA 元件都与调节蛋白(激活剂和阻遏物)结合,从而帮助或阻碍核心转录启动。
- 对于每一个基因,都有一个、几个或许多特定的调控元件进行组合,精细调节其开关。不同调控元件的组合可以调控打开或关闭特定基因的数量和位置。
- 增强子有助于进一步提高基因的转录。
★ 增强子 enhancer
-
增强子是指能使与它连锁的基因转录频率明显增加的 DNA 序列。
-
增强子的特性:
1)增强效应十分明显。
2)增强效应与其位置和取向无关。距离启动子有一定的距离,可以位于其上游或者下游。
3)大多为重复序列,一般长约 50bp,适合与某些蛋白因子结合。常含有一个核心序列:(G)TGGATA/TA/T©,该序列是产生增强效应时所必需的。
4)其增强效应有严密的组织和细胞特异性,说明增强子只有与特定蛋白质(转录因子)相互作用才能发挥功能。
5)没有基因专一性,可以在不同的基因组合上表现增强效应。
6)许多增强子还受外部信号的调控,如金属硫蛋白基因启动区上游所带的增强子,就可以对环境中的锌、镉浓度做出反应。 -
远程调控机制:(为什么增强子的作用与距离和方向无关?)
① 成环(目前普遍接受的机制):影响模板附近的 DNA 双螺旋结构,导致 DNA 双螺旋弯折或在反式作用因子的参与下,以蛋白质之间的相互作用为媒介形成增强子与启动子之间“成环”连接,活化基因转录。Cohesins( 黏连蛋白) 有助于稳定增强子。
② 滑动:将模板固定在细胞核内特定位置,如连接在核基质上,有利于 DNA 拓扑异构酶改变 DNA 双螺旋结构的张力,促进 RNA 聚合酶Ⅱ在 DNA 链上的结合和滑动;
③ 增强子区可以作为反式作用因子或 RNA 聚合酶Ⅱ进入染色质结构的 “入口”。

-
增强子的基本作用是 增加启动子附近的激活子浓度
◆ 增强子可以通过将蛋白质带到启动子附近而起作用。
◆ 增强子不作用于长线性 DNA 相反端的启动子,但当 DNA 通过蛋白质桥连接成环时增强子能发挥作用。
◆ 增强子和启动子位于不同环状 DNA 分子时增强子不起作用,但是当两个环链接在一起时能起作用。
反式作用因子 (
trans-acting factor)
- 能直接或间接与顺式作用元件相互作用,进而调控基因转录的一类调节蛋白,统称为反式作用因子。
- 按照功能分三类:
1)基本转录因子:识别 promoter 元件
2)转录调节因子:识别 enhancer 或 silencer
3)共调节因子:不能进行 DNA-蛋白质相互作用
1)基本转录因子 (general transcription factors, GTFs):是指能够直接或间接与启动子核心序列 TATA 盒特异结合、并启动转录的一类调节蛋白。

2)转录调节因子
◆ 这类调节蛋白能识别并结合转录起始点的上游序列和远端的增强子元件(沉默子),通过 DNA-蛋白质相互作用而调节转录活性。决定不同基因的时间、空间特异性表达。
◆ 常将基本转录因子和转录调节因子统称为转录因子 (transeription factor, TF)。
- 转录激活因子 (transcriptional activator)
- 转录抑制因子 (transcriptional repressor)
3)共调节因子 (transcriptional regulator/ co-factor):本身无 DNA 结合活性,但能与转录因子发生蛋白-蛋白相互作用,进而影响它们的分子构象,以调节转录活性。
- 共激活因子:与转录激活因子有协同作用
- 共抑制因子:与转录抑制因子有协同作用。
- 功能:通过与激活因子和 GTFs(普通转录因子)相互作用参与转录的激活。
- 结构:一种大的多蛋白复合物,不直接与 DNA 结合。
★ 转录因子通常含有结构域
- DNA 结合域 (DNA binding domain,
DBD):碱性氨基酸结合域 Basic AA (K/R) rich, positively charged 带正电(容易与 DNA 结合)【K 赖氨酸 Lys;R 精氨酸 Arg】 - 转录激活域 (trans-activation domain,
TAD):酸性激活域 (D/E-rich); 谷氨酰胺 (Q) 富含域;脯氨酸 P 富含域。【D 天冬氨酸 Asp;E 谷氨酸 Glu】
★ TF 中常见的 DNA 结合域 (
DBD)

(1) Zinc Finger 锌指结构:锌指结构家族蛋白特有的半胱氨酸 C 和组氨酸 H 残基之间氨基酸残基数基本恒定,有锌参与时才具备转录调控活性。
- Cys-X2-4-Cys-X3-Phe-X5-Leu-X2-His-X3-His
- C-terminal: α-helix → binding DNA
- C2H2 (两个半胱氨酸+2 个组氨酸)是最常报道的哺乳动物转录因子中的一类锌指,主要结合在 DNA 双螺旋结构的大沟中。
- 常结合 GC box。
- 识别螺旋位置:螺旋上的-1,2,3 和 6 位置。
- 多肽序列与碱基间的相互作用:每一个锌指的 α-helix 可以特异识别 3-4 个碱基。
(2) 碱性亮氨酸拉链 (basic-leucine zipper), 即 bZIP 结构

- 亮氨酸拉链蛋白是二聚体,拉链螺旋位于蛋白质的 C-末端
- 在螺旋的每第 7 位有 L 亮氨酸 Leu,在二聚体中面对面
- N-末端螺旋具有 正电荷的氨基酸 (K/R) :结合 DNA 大沟
(3) Homeodomain 同源异型域:

- 同源域是指编码 60 个保守氨基酸序列的 DNA 片段,它广泛存在于真核生物基因组内。
- 属于螺旋-转角-螺旋家族 (helix-turn-helix,
HTH) 蛋白,每个同源异形域蛋白包括三个 α 螺旋,第二和第三个螺旋形成螺旋转角-螺旋模体。 - 同源域蛋白通过其第 3 个螺旋与链 DNA 的大沟相结合。其 N 端的延伸部分则与 DNA 的小沟相结合,提高了稳定性。
(4) bHLH 结构 (basic-Helix-Loop-Helix 碱性-螺旋-环-螺旋)

- 来自两个单体中的每一个延伸的 α 螺旋区域插入 DNA 的大沟中
- 二聚化表面由两个螺旋区域形成
- 亮氨酸拉链和 HLH 蛋白通常被称为碱性拉链 bZIP 和碱性 HLH 蛋白 bHLH
- 肌细胞定向分化的调控因子 MyoD-1、原癌基因产物 Myc 及其结合蛋白 Max 等都属于 bHLH 蛋白。
- bHLH 类蛋白只有形成同源或异源二聚体时,才具有足够的 DNA 结合能力。当 HLH 蛋白二聚体中的一方无碱性区时,该二聚体明显缺乏对靶 DNA 的亲和力,不能结合 DNA。
★ TF 常见的转录激活域 (
TAD)
-
在真核生物中,反式作用因子的功能由于受蛋白质-蛋白质之间相互作用的调节变得精密、复杂,完整的转录调控功能通常以复合物的方式来完成,这就意味着并非每个转录因子都直接与 DNA 结合。
-
转录激活/活化域(
trans-activation domain)是反式作用因子中唯一的结构基础。 -
反式作用因子的功能具有多样性,其转录活化域也有多种,通常依赖于 DNA 结合结构域以外的 30 ~ 100 个氨基酸残基。
-
不同的转录活化域大体上有下列几个特征性结构:

(1)GAL4酸性结构域:富含酸性氨基酸(D/E)。哺乳动物细胞中糖皮质激素受体的两个转录活化域,AP1 家族的 Jun 及 GAL4 都有酸性的螺旋结构。特异性诱导转录起始的活性并不是很强,结合 TFⅡD 复合物或 RNA polⅡ,稳定转录起始复合物。(2)
SP1谷氨酰胺结构域:富含谷氨酰胺Gln(Q)。SP1 是启动子 GC 区的结合蛋白,除结合 DNA 的锌指结构以外,SP1 共有 4 个参与转录活化的区域,其中最强的转录活化域很少有极性氨基酸,却富含谷氨酰胺,达该区氨基酸总数的 25% 左右。哺乳动物细胞中的 Oet1/2、Jun、AP2、血清应答因子 (SRF) 等都有相同的富含谷氨酰胺的结构域。(3) 富含脯氨酸结构域:
CTF-NF1因子的羧基端富含脯氨酸 Pro(达 20% ~ 30%),很难形成 α 螺旋(有脯氨酸就会形成拐角)。在 Ocet2、Jun、AP2、SRF 等哺乳动物因子中也有富含脯氨酸的结构域。 -
转录因子的特异性取决于其 DBD(识别 DNA 序列),激活域 TAD 是可以互换的。
活化因子作用于介体复合物

- 介体是一种含有多种 (20-50) 蛋白质的稳定复合物
- 介体与 RNA pol Ⅱ和转录因子结合,并“调节”调节信号至 pol Ⅱ ——一个大的共激活因子!
转录激活机制:激活转录可以不需要激活域
① 变构模型 allosteric model
② 招募模型 recruitment model
- Gal4 DBfused with Gal11 (a TAF)
- LexA DB fused with Gal11 (a TAF)
- HIV
tat蛋白(一种 RNA 结合蛋白)可以刺激转录而不结合 DNA!
tat 蛋白的激活结构域可以通过 与前轮转录的 RNA 产物(tar序列) 结合到启动子附近,激活转录。 - 招募模型共性:
激活子中 DNA-binding domain (DBD) 与增强子、上游启动子元件结合时,或者 tat 蛋白中的 RNA-binding domain 连接到新制造的 RNA 产物时,将 activation domain (TAD) 招募到转录起始点附近。
激活因子功能域的独立性
- 真核生物转录调控因子具有组件式结构 (modular) 特征,这些蛋白往往由两个或两个以上相互独立的结构域,其中 DNA 结合结构域 (DNA binding domain, DBD) 和转录激活结构域 (transcription activation domain, TAD) 是转录激活因了发挥功能所必须的。
- 激活因子的 DNA 结合域和转录激活域彼此独立折叠,形成独立的三维结构,独立行使功能。
- DBD 能与特定基因启动区结合,但不能激活基因转录,由不同转录调控因子的 DBD 和 TAD 所形成的杂合蛋白却能行使激活转录的功能。
- 不同转录因子的 TAD 和 DBD 可以互换,杂合激活因子具有转录活性。

总结:
● 所有激活因子的共性:识别靶位点(启动子、增强子)的特异性由 DNA 结合域决定。
● 直接作用的转录激活因子具有 DNA 结合域 (DBD) 和转录激活域 (TAD)。
● 没有转录激活域 (TAD) 的激活因子可能与具有转录激活域的共激活因子一起行使功能。
● 转录区域中许多元件是激活因子的靶位点,决定了转录的起始。
● RNA 聚合酶和许多基本转录因子相互作用,形成全酶复合物行使功能。其 CTD 协调了转录和 RNA processing。
● 转录因子 DNA 结合域将转录激活域带到基础转录区域附近,可直接或间接(通过中介复合体)与 RNA 聚合酶全酶复合物相互作用(招募模型)。
协同转录
- 协同作用 (
Synergy):由多种因子诱导的高水平转录。 - 转录因子可以以非线性方式增强转录
- 由于与转录机器的多次接触而发生协同激活
- 同一活化因子的多个拷贝也诱导协同激活
● 真核生物只有有限数量的转录调节蛋白
Q: 少量的调节蛋白是如何调控大量的基因转录的?

- Enhancer(增强子):多个顺式元件的协同
- 增强子通常具有几种转录因子的结合位点
- 转录因子可以在相邻位点协同结合
- 组装因子,没有调控结构域,例如 HMGI, 可以协助组装
- 显着提高与 DNA 和转录元件的结合亲和力
● 不同调控元件对转录的净效应是什么?
- 不同调控元件对转录的影响取决于不同蛋白质结合的组合。
- 如果正调控蛋白在转录的增强子和启动子元件激活处都结合;
- 如果负调节蛋白与增强子结合,且正调节蛋白与启动子元件结合,则取决于两种调控蛋白之间的相互作用。如果负调控蛋白质具有很强的作用,则该基因被抑制。(正负 PK,总体促进就促进,总体抑制就抑制)
基于转录调控原理的应用
(1) GR(糖皮质激素受体)inducible system
-
Steriod receptors(类固醇受体)are transcription factors
-
Receptors for many steroid(类固醇) and thyroid hormones(甲状腺激素)具有相似的蛋白组成,具有单独的保守的 N 端 DNA 结合区域和 C 端激素结合区域。属于 Zinc Finger TF。

-
糖皮质激素调节基因转录:糖皮质激素能使糖皮质激素受体(Glucocorticoid Receptor, GR)运输到细胞核中并结合增强子,启动转录。糖皮质激素通过核穿梭 (
nuclear shuttling) 激活下游信号通路。该激素与相应受体结合,改变其构象,使之进入细胞核内,结合在能够促进转录的相应增强子上,从而促进下游基因的转录。利用 GR 的特性构建可诱导表达融合蛋白系统
◆ 目的:转录因子 X 在细胞内调控了哪些蛋白的转录?
◆ Dexamethasone (DEX) : 地塞米松(抗炎药),一种合成的糖皮质激素;
◆ 通过分子克隆的方法将糖皮质激素受体 GR 和要研究的核蛋白 X 构建成融合蛋白,转基因到酵母、动物细胞或者植物中;
◆ 不加外源 DEX 时,融合蛋白与 HSP90 形成复合物,由于构象和空间位阻等原因,融合蛋白存在于胞质中,不能定位到细胞核;
◆ 添加 DEX 时,DEX 扩散入胞与 GR 相结合,融合蛋白改变构象后核定位信号暴露,即可入核行使功能,导致下游基因表达;
◆ 用以研究核蛋白 X 的功能(包括转录因子)。
(2) Activation tagging approaches 激活标签法
- 构建 T-DNA 序列,其中包括 4 倍重复的 CaMV 的 35S 增强子序列,
4×35S 元件可以大大增强相邻基因的转录; - 通过转基因的方法将 T-DNA 片段整合到植物基因组中;
- 植物基因组上与 T-DNA 插入位点相近的基因表达量增高,即得到这个基因(TAIL-PCR) gain-of-function 的转基因植物。
- 突变体筛选
- 找到拟南芥(拟南芥)基因组中的 T-DNA 插入位点 (How? )
- 确定赋了突变表型的正确基因 (How? )
- 雌激素调控的诱导表达系统:雌激素受体调控域 E 与 X、V 结构域融合,导致 XVE 融合蛋白不能入核,加入雌激素后能入核。

(3) Enhancer trap system 增强子捕捉技术

- Enhancer trap 的质粒包含一个报告基因 (lacZ) 和基本启动子,这段启动子不足以启动报告基因的表达,但是对增强子非常敏感。
- 将这样的质粒整合到基因组中,如果 插入位点附近有增强子报告基因就会表达。因此只要通过观测报告基因的表达情况(比如时空上的特异性)就可以知道这个增强子的作用,进而研究由这个增强子调控的内源基因的表达特性。(此方法局限性:增强子与启动子隔得很远)
(4) ZFN 技术
-
锌指核酸酶 (Zinc-finger nucleases,
ZFN) 是人工改造的限制性核酸内切酶,利用不同的锌指结构识别特异 DNA 序列,利用核酸酶切断靶 DNA。
-
锌指结构中每一个 α 螺旋可以特异识别 3-4 个碱基。
-
人工设计识别特异 DNA 序列的 α 螺旋采用如上的通用序列,通过改变其中 7 个 X 来实现识别不同的三联体碱基,TGEK 是多个螺旋间的连接序列;
-
构建成对人工锌指结构域和 FokⅠ核酸酶融合蛋白 (ZFN) 可以在指定区域切断 DNA 双链。
-
Fok1 dimerization is required for DNA cleavage.
-
研究人员可以利用 ZFN 技术进行各种基因编辑,比如基因敲除。已建立有 ZFN 库,识别多种 DNA 序列,但还不能达到识别任意靶 DNA 的目的,其应用受到一定的限制。
(5) TALEN 技术
- TALEN=Transcription Activator- Like Effector+Fokl Nuclease fusion protein
- TALE:来自黄单胞菌 (Xanthomonas) 的转录激活因子样效应子,TALE 可以在发病过程中特异性结合和调节植物基因。
- 长度为 34aa 的重复肽段中的第 12、13 个氨基酸可以特异识别 DNA 单个碱基,形成 2aa→ 1bp 的特殊 codon。利用这个特性可以人工设计识别任意碱基序列的 TALE 蛋白。

详见:【中科院】分子生物学-朱玉贤第四版-笔记-第 11-12 讲 基因功能研究技术 8.1.4 - TALEN 技术存在的问题:
① 构建重复序列难:但氨基酸存在简并性,可以改成氨基酸序列相同,但碱基不同的序列。
② 脱靶效应:结合一段序列的概率高,意味着结合另一段重复序列的概率低。 - TALE reporter 系统:设计 TALE 结合位点接上报告基因 (
mCherry) ,同时构建一个特异识别这种 TALE 结合序列的 TALE 蛋白,将两种质粒共同转入细胞,那么这个人工 TALE 蛋白可以启动报告基因 mCherry 的表达。

◆ TALEs 重复序列后加了 NLS(核定位信号)、VP64(一种转录激活域 TAD,与报告基因结合时激活报告基因的转录)、2A(使得核糖体可以跳过 2A 肽,继续翻译后面的 EGFP 而发光,发绿色荧光表明成功转入质粒)。
◆ TALE reporter 上有 TALE 结合位点,与 TALE 结合后,后面接 minCMV 的启动子,启动 mCherry 基因的转录而发出红色荧光。
(6) 酵母杂交技术
酵母单杂交系统
- 酵母单杂交系统 (
Yeast one-hybrid system) 是上世纪 90 年代中发展起来的研究 DNA-蛋白质之间相互作用的新技术,在酵母细胞内研究真核生物中 DNA-蛋白质之间的相互作用,并通过筛选 DNA 文库直接获得靶序列相互作用蛋白的编码基因。 - 原理:将顺式作用元件与最基本启动子 (minimal promoter,
Pmin) 相连并将报告基因连到 Pmin 下游,将待测转录因子 cDNA 与酵母转录激活结构域 (transcription-activating domain, TAD) 融合表达载体导入酵母细胞,该基因产物如果能够与顺式作用元件相结合,就激活 Pmin 启动子,报告基因表达。(连接入 3 个以上顺式作用元件,可增强转录调控因子的识别和结合效率。)

- 从拟南芥 cDNA 文库中筛选与已知顺式元件 DRE 结合的转录因子
将不同的未知基因分别克隆到 pYF503 中 (GAL4 AD 为 GAL4 激活结构域,empty vector 表示无基因克隆到 pYF503 中),转入酵母中表达 N 端带有 GAL4 DNA 结合结构域的融合蛋白,观察报告基因表达与否。

酵母双杂交系统
- 酵母双杂交系统 (
Yeast two-hybrid system):用于验证两个蛋白质之间的相互作用。 - 原理:利用真核生物转录调控因子的组件式结构 (modular) 特征。单独的 DNA 结合域 BD 能与特定基因的启动区结合,但不能激活基因的转录;而由不同转录调控因子的 BD 和转录激活结构域 AD 所形成的杂合蛋白却能行使激活转录的功能。
- 技术流程:
(简单说:带 BD 的已知靶蛋白、带 AD 的待筛选 cDNA 文库一起转入酵母细胞杂交,捕获筛选与靶蛋白相互作用的未知蛋白)
① 实验中,首先运用基因重组技术把编码已知蛋白的 DNA 序列连接到带有酵母转录调控因子(常为 GALI、GAL4 或 GCNI)DNA BD 编码区的表达载体上。导入酵母细胞中使之表达带有 DNA 结合结构域的杂合蛋白,与报告基因上游的启动调控区相结合,准备作为“诱饵"捕获与已知蛋白相互作用的基因产物。
② 将已知的编码 AD 的 DNA 分别与待筛选的 cDNA 文库中不同插入片段相连接,获得“猎物”载体,转化含有"诱饵”的酵母细胞。
③ 一旦酵母细胞中表达的“诱饵”蛋白与“猎物"载体中表达的某个蛋白质发生相互作用,不同转录调控因子的 AD 和 BD 就会被牵引靠拢,激活报告基因表达。分离有报告基因活性的酵母细胞,得到所需要的“猎物"载体,就能得到与已知蛋白相互作用的新基因。

第 14 讲内容总结:
真核生物转录水平调控
转录起始调控三个基本要素:RNA 聚合酶;顺式作用元件:反式调控蛋白
RNA 聚合酶全酶的装配和 C 端磷酸化调控
顺式作用元件的种类、核心元件、调控元件
反式调控蛋白的种类:基本转录因子;转录调节因子;共调节因子
组合调节/协同调节
转录因子常见结构域:DNA 结合域;转录激活域
DNA 结合域的几种基本类型和特点:锌指;bZIP;同源异形域;bHLH
常见转录激活域及其特点:GAL4
Y2H
转录因子激活转录的分子机理
增强子的协同调节
GR (糖皮质激素受体) inducible system
Activation tagging approaches
Enhancer trap system
ZFN
TALEN
思考题:
怎样把任何序列的一段 DNA 变成 cis element?
★ 染色质结构对基因转录的调控(表观遗传调控)
- 组蛋白修饰
- DNA 甲基化
● Epigenetics (表观遗传学)
表观遗传学是指对基因组功能相关的修饰,不涉及核苷酸序列的变化。它们包括细胞遗传特性的变化,但不代表遗传信息的变化。
10.1.2 组蛋白修饰
- 天然真核基因没有裸露 DNA 分子,DNA 分子与被称为组蛋白的碱性蛋白质形成复合体 ( 染色质)。
- Nucleosome(核小体)是染色质的基本结构单位,由 ~ 200bp DNA 和组蛋白八聚体组成。
- 组蛋白是碱性极强的蛋白质,含有至少 20% 的精氨酸 R 或者赖氨酸 K,在中性 pH 环境中带较强的正电荷:可在强酸中提取组蛋白;与其它大多数蛋白不同,在非变性电泳中向阴极泳动。
- 不同物种中,H2A、H2B 中度保守;H3 和 H4 极端保守;H1 变异较大。
- 在真核生物中,组蛋白基因常常以多拷贝存在。
- 核心组蛋白周围包裹着 146bp 的 DNA 片段。
● 转录过程中核小体的位阻效应:如果组蛋白与 DNA 紧密结合,转录因子(和其他 DNA 结合蛋白)不能有效识别 DNA 元件。
● 核心组蛋白和 H1 抑制转录
- 重建染色质的体外转录:核心组蛋白抑制 75% 的转录活性,剩余 25% 的转录活性由启动子未被核小体核心颗粒覆盖所引起。
含有果蝇 Kruppel 基因的质粒与核心组蛋白重构染色质,用引物延伸实验检测转录效率。
Polyglutamate: 多聚谷氨酸,作为组蛋白返回 DNA 的载体。
Sakosyl: 肌氨酰:阻止再起始,只能发生一次的转录。 - 组蛋白 H1 进一步抑制模板活性;H1 的抑制作用可被转录因子抵消。
● 活跃基因的调控区无核小体
- 真核基因的活跃转录是在常染色质上进行的。转录发生之前,染色质常常会在特定的区域被解旋松弛,形成自由 DNA。这种变化可能包括核小体结构的消除或改变、DNA 本身局部结构的变化甚至从右旋型变为左旋型 (Z-DNA) 等,这些变化可导致结构基因暴露,促进转录因子与启动区 DNA 结合,诱发基因转录。
- DNase Ⅰ敏感性和基因表达:
转录基因区域的染色体结构比无转录活性基因的结构松散。
DNase Ⅰ:一种序列非特异性内切核酸酶,只能切割它可以接触的 DNA。
转录活性 DNA 比转录无活性 DNA 更容易被 DNase I 酶切。 - 活跃状态的 DNA 更易于受核酸酶的攻击而降解。活跃表达基因所在染色质上一般含有一个或数个 DNA 酶Ⅰ超敏感位点 (
hypersensitive site), 它们大多位于基因转录起始上游,包括 启动子区。非活性态基因的 5’端相应位点不表现对 DNA 酶Ⅰ的超敏感性。 - 超敏感位点的产生可能是染色质结构规律性变化的结果。正是由于这种变化,使 DNA 容易与 RNA 聚合酶和其他转录调控因子相结合,从而启动基因表达,同时也更易于被核酸酶所降解。
- 应用 Southern 印记检测 DNase 超敏感区:只有基因活跃时,用 DNase 处理,才能切开 DNA。

● 异染色质 (Heterochromatin):染色质结构非常致密,DNA 不可接近。
- 异染色质中核小体的位阻效应:如果在启动子处形成致密的核小体,则转录机器不能结合 DNA。
● 常染色质 (Euchromatin):染色质结构相对伸展开放,有潜在活性。
染色质状态的转换:

(1)组蛋白替换(histone variants):主要是 H3 和H3.3(组蛋白变体),还有 H2A 和H2A.Z/X。

- 一般而言, H2A/H2B 和 H3/H4 形成的 nucleosome 是最主要的,也是最稳定的形式。
- 当 H3.3 替换 H3, 或 H2A.Z/X 替换 H2A 时,所形成的 nucleosome 不太稳定,因此常常激活基因转录。
(2)染色质重塑(chromatin remodeling):染色质重塑由大型复合物进行,使用 ATP 水解来提供重塑的能量,从特定 DNA 序列中置换核小体。
- 两种主要类型的染色质重塑复合物:SWI/SNF 和 ISW。
- 重塑复合体的核心是它的 ATPase 亚基。重构复合物通常根据 ATP 酶亚基的类型分类。
- 染色质重塑模型:根据核小体与周围包裹的 DNA 的相对位置或组成的变化不同,提出 4 种模型。
a) nucleosome sliding:核小体沿 DNA 滑动,露出先前被遮挡的 DNA 区域(红色标记);
b) histone exchange:变体组蛋白替换为标准组蛋白,形成变异核小体;
c) nucleosome eviction:剔除核小体,打开一大片 DNA 区域。这种机制可能依赖于其他蛋白质,如组蛋白伴侣或 DNA 结合因子;
d) altered nuclesome structure:在核小体表面形成一个环。SWI/SNF 家族的重塑蛋白可能在核小体表面产生稳定的 DNA 环(loop),使核小体的中心位点可用。
Note:限制酶的切割速度很快,不是限速步骤;染色质构象变化后暴露限制酶位点是限速步骤。

- 染色质重建机制尚不清楚,但涉及核小体的移动,伴随 DNA 与核心组蛋白连接的松弛。
- 重塑复合物通过活化因子(或阻遏因子)与染色质结合。基因组研究表明,大多数结合转录因子的位点没有核小体。
(3)组蛋白修饰(histone modifcation)
● 启动子激活涉及:
- 序列特异性转录激活因子 (TF) 的结合
- 重塑复合物的招募和作用
- 乙酰化复合物的招募和作用
- 对组蛋白或 DNA 的更多修饰
● 对组蛋白的化学修饰在基因调控中起重要作用:染色质的化学修饰导致染色质的结构变化(染色质重建,Chromatin remodeling),影响转录因子和转录酶接近 DNA, 形成高转录活性(常染色质)和低转录活性(异染色质)的区域,进而调控基因的转录。
● 组蛋白化学修饰发生在组蛋白 N 端尾部(有大量碱性氨基酸,容易发生修饰),尤其是组蛋白 H3 和 H4 的修饰起始了染色质结构的变化。
H2A 和 H2B 会形成异源二聚体 H2A·H2B dimer;H3 和 H4 也会形成异源四聚体 H3·H4 tetramer。

与转录相关的组蛋白结构
- 组蛋白折叠是由 3 个α 螺旋组成,α螺旋都有仲展的尾部;
- H2B 和 H3 的尾部从 2 个相邻的 DNA 小沟中穿出(说明 H2B 和 H3 与 DNA 有相互作用);
- H4 的尾部暴露在核心颗粒的侧面;
- H4 的尾部富含碱性氨基酸,与相邻核小体的 H2A·H2B dimer 的酸性区域强烈互作,调控核小体间的交联。
组蛋白化学修饰的类型

- 组蛋白的乙酰化和磷酸化(作用于碱性氨基酸,含-NH2)可以减少组蛋白的正电性,从而使其与 DNA 结合变疏松,改变 H2A-H2B 和 H3-H4 间的相互作用,使染色质结构发生变化(浓缩性降低)。
- 更多的时候,组蛋白的化学修饰改变了其 binding features,从而招募不同的 effector proteins.
组蛋白密码

-
组蛋白的氨基末端尾部占核小体质量的四分之一,拥有绝大多数已知的共价修饰位点。
-
Active marks :乙酰化 (Ac 标志)、精氨酸 R 甲基化(黄色 Me 六边形)以及一些赖氨酸 K 甲基化,如 H3K4和 H3K36(绿色 Me 六边形)。H3K79 具有抗沉默功能。
-
Repressvie marks :H3K9、H3K27、H4K20(红色 Me 六边形)
-
相对而言,组蛋白的甲基化修饰方式是最稳定的(形成的键最稳定),所以最适合作为稳定的表观遗传信息。
-
乙酰化修饰具有较高的动态,另外还有其他不稳定的修饰方式,如磷酸化、腺苷酸化、泛素化、SUMO 化、ADP 核糖基化等等。这些修饰更为灵活的影响染色质的结构与功能,通过多种修饰方式的组合发挥其调控功能。
-
在一个基因调控区附近的特定核小体上,组蛋白修饰的组合影响该基因的转录效率。这些能被专一识别的修饰信息为组蛋白密码。
-
这些组蛋白密码组合变化非常多,因此组蛋白共价修饰可能是更为精细的基因表达方式。
组蛋白乙酰化和去乙酰化 (Histone Acetylation and De-acetylation )

- 组蛋白乙酰化:减少组蛋白的正电荷,从而放松染色质→激活转录
★ 乙酰转移酶
- 乙酰转移酶是一种向蛋白质添加乙酰基的酶。
- 组蛋白乙酰转移酶 (
HAT) 通过添加乙酰基来修饰组蛋白;一些转录共激活因子具有 HAT 活性。 - 脱乙酰酶(或脱乙酰转移酶)是从蛋白质中除去乙酰基的酶。
- 组蛋白去乙酰转移酶 (
HDAC或HDA) 从组蛋白中除去乙酰基;它们可能与转录抑制因子有关。 - HAT 和 HDAC 具有低的位点特异性,因此可以在 H3 和 H4 上的多个 Lys 残基处添加/去除乙酰基。

● 组蛋白乙酰化激活转录
许多 HAT 存在于转录激活复合体 (co-activators) 中(如 SAGA 复合体中的 GCN5),且多种 HAT 可同时存在于个辅助激活复合体中(如 P300, ACTR 与 PCAF 的共同作用).
● 阻遏物复合物(例如 Sin3 复合物)含有至少三种组分:DNA 结合亚基(特异性识别 DNA)、辅阻遏物和组蛋白脱乙酰酶 (HDAC)。
● HDAC 是许多转录共抑制复合物中的活性成分。HDACI 和 HDAC2 均与介导转录抑制的 mSin3A 有关。通过与许多序列特异性的转录因子相互作用,可使 HDAC-mSin3 复合物结合到特异性的启动子,抑制相关基因的表达。
● 这些转录因子也可结合 HATs 复合体,激活基因表达。因此,基因的激活或抑制,往往取决于其特异转录因子到底与 HAT 或 HDAC 结合。
● 癌症细胞中的组蛋白去乙酰化
- 在许多肿瘤细胞中,发现 HDAC 高度表达。
- 两种可能的机制:
① 通过改变基因表达程序,包括抑制肿瘤抑制基因(即 p53)
② 在更整体的层面上,组蛋白去乙酰化可能影响基因组稳定性和/或染色体完整性,从而产生更多突变。 - 因此,HDAC 长期以来一直是癌症治疗的有希望的药物靶标(特异性不好,副作用大)。
组蛋白甲基化和去甲基化 (Histone Methylation and Demethylation )

- 比 Histone Acetylation 复杂很多。
- 甲基化位点主要在组蛋白的赖氨酸 (K) 和精氨酸 ® 残基上:
组蛋白 H3 的第 4、9、27 和 36 位,H4 的第 20 位 Lys;
H3 的第 2、17、26 位及 H4 的第 3 位 Arg。 - 甲基化不改变组蛋白的电荷,而是通过不同位点、不同程度的甲基化修饰,来招募不同的 effector 蛋白。

- 组蛋白甲基化是由组蛋白甲基化转移酶 (histone methyl transferase,
HMT) 完成的。由细胞内合成的SAM(S-methyl-AdenoMethionine, S-甲基-腺苷甲硫氨酸) 提供甲基。转移后形成 C-N 键,连接稳定,不易破坏。 - 赖氨酸 K 残基能够发生单、双、三甲基化,而精氨酸 R 残基能够单、双甲基化(对称 SDMA 和不对称 ADMA 双甲基)(R 比 K 更稳定),这些不同程度的甲基化极大地增加了组蛋白修饰和调节基因表达的复杂性。
● 甲基化对转录活性的调控
-
精氨酸 R 甲基化:是一种相对动态的标记,精氨酸甲基化与基因激活相关,而 H3 和 H4 精氨酸的甲基化丢失与基因沉默相关。
-
赖氨酸 K 甲基化:是基因表达调控中一种较为稳定的标记。
➢ H3 第 4 位的赖氨酸残基甲基化 (H3K4me) 与基因激活相关,H3K36和H3K79的甲基化也与基因激活有关。
➢ H3 第 9 位和第 27 位赖氨酸甲基化 (H3K9me,H3K27me) 与基因沉默相关,H4K20的甲基化与基因沉默相关。
➢ 同一位点的甲基化个数与基因沉默和激活的程度相关。组蛋白甲基化 染色体常见分布 主要功能 H3K9me3 中心粒,端粒 组成型异染色质(沉默) H3K27me3 沉默基因 沉默基因 H3K4me3 转录起始位点 转录活性区标记 H3K36me3 转录区 转录活性区标记
● 组蛋白赖氨酸甲基转移酶-HKMTs
- 所有
HKMT都含有甲基转移酶活性的SET 结构域 - HKMT: 一些可以催化组蛋白赖氨酸的单甲基化、二甲基化和三甲基化,而其它只能催化某种特定形式。
- 赖氨酸不同的甲基化修饰程度由什么决定?(由酶的特殊结构决定)
➢ DIM5 可以使 H3K9 三甲基化,但 SET7/9 只能使 H3K4 单甲基化。
➢ 在 DIM5 中,在酶的赖氨酸结合口袋中存在苯丙氨酸 (F281) ,其可以容纳赖氨酸的所有甲基化形式,从而允许酶产生三甲基化产物。
➢ SET7/9 在相应位置具有酪氨酸 (Y305) ,使得它仅能容纳单甲基产物。
➢ 突变研究表明,DIM5 F281Y 的突变将酶转化为单甲基转移酶,而 SET7/9 (Y305F) 中的突变产生了能够使其底物三甲基化的酶。
● 组蛋白赖氨酸去甲基化酶
- HK 甲基化是一种非常稳定的修饰,很难在体外去除
- 第一个 HK 去甲基化酶于 2004 年被鉴定出来——LSD1,一种 H3K4 和 H3K9 去甲基化酶
- 含有 JmjC 结构域的蛋白质 (JmjC-domain-containing proteins) 编码组蛋白 K 去甲基化酶
用于研究组蛋白修饰的全基因组方法
- 组蛋白修饰的组学方法常常结合使用包括 DNA 微阵列和高通量测序的高通量技术。
- ChIP-PCR(染色质免疫沉淀,chromatin immunoprecipitation): 使用对某种组蛋白修饰特异的抗体分离染色质片段。
- ChIP-chip:最普遍的技术,DNA 需要被扩增,杂交;
- ChIP-Seq(最强大):最近开发的技术,非常有限的扩增,更多定量,测序的读数可以直接映射到基因组,可以直接比较不同基因组区域的修饰水平。例如:ChIP-Seq 用于检测小鼠 ES 细胞中的组蛋白修饰。
● 用于分析某位点组蛋白修饰的染色质免疫沉淀方案
- 染色质交联(甲醛):把 DNA 和组蛋白交联起来,得到核小体。
- 染色质制备
- 预清除和免疫沉淀 (IP):加能特异性识别组蛋白修饰(如 H3K4)的抗体,抓带有特殊修饰的核小体。
- 免疫复合物的收集,洗涤和洗脱
- 反向交联:去交联,去掉与 DNA 交联的组蛋白。
- DNA 清理:纯化 DNA。
- 进行 PCR 反应或 DNA 测序。

- 原代 T 细胞中六个组蛋白标记的全基因组谱

组蛋白修饰的交互作用模型
- 组蛋白修饰之间会发生相互作用,一个组蛋白的修饰可能触发另一个组蛋白修饰。

- 组蛋白的 乙酰化激活 染色质,DNA 和组蛋白的 甲基化使染色质失活。
- DNA 和组蛋白的甲基化与异染色质有关。两种类型的甲基化事件可能是相关联的。
组蛋白 K 泛素化修饰

- 泛素化 修饰的结果是给赖氨酸 K 加一个蛋白质(泛素)。
- H2A/H2B 泛素化修饰,引起结构变化。
- 一般在细胞内,蛋白质泛素化后发生降解,但组蛋白的泛素化修饰是改变结构。
组蛋白修饰的作用方式
1、修饰直接影响染色质的整体结构: 松开染色质,通常激活转录。
- K 乙酰化:降低组蛋白的正电荷
- S / T 磷酸化:降低正电荷
- K 泛素化:改变核小体的构象
2、修饰调节(正或负)效应分子的结合。 修饰染色质,激活或抑制转录。
- K/R Methylation
10.1.3 DNA 甲基化
DNA 甲基化的位点
- DNA 甲基化主要形成 5-甲基胞嘧啶 (5-mC) 和少量的 N6-甲基腺嘌呤 (N6-mA) 及 7-甲基鸟嘌呤 (7-mG),约占总核苷酸量的 1%。

- 由于 5-甲基胞嘧啶 (5-mC) 脱氨后生成胸腺嘧啶 (T),因此 DNA 甲基化提高了该位点的突变频率。

- 5-甲基胞嘧啶可自发或在酶的催化下转变为胸腺嘧啶。无论是在体外还是在体内,不需要酶促,就能自发脱氨基转变为胸腺嘧啶 (T),从而导致了部分 CG 变成 TG。
- 5-甲基胞嘧啶的这种自发脱氨基反应的速度是有酶介导的非甲基化胞嘧啶脱氨基生成尿嘧啶 (U) 的两倍以上。
- 甲基化有利于脱氨基反应。
DNA 发生甲基化的原因
- 1、原核生物中,DNA 甲基化是为了抵抗噬菌体侵害而发生碱基 C 和 A 上的化学修饰。如大肠杆菌的限制修饰系统中,自身 DNA 特定位点的甲基化可以避免限制性内切酶的切割。
- 2、真核生物中,甲基化被分为对称性甲基化 (canotical / symmetric methylation),包括 CpG 和 CpNpG) ,以及非对称甲基化 (asymmetric methylation),包括 CpHpH (H=A+T+C) 。多数细胞 5-甲基胞嘧啶主要出现在 CpG 中。DNA 甲基化能引起染色质结构、DNA 构象、组蛋白修饰及 DNA 与蛋白质相互作用方式的改变,从而控制基因表达(一般是 抑制基因表达)。
CpG island 作为甲基化调控基因转录的单位
-
多个 CpG 序列 (1-2kb) 集合成簇形成了富含甲基化位点的 CpG 岛 (
CpG island),具有很高的序列保守性。 -
真核生物约一半的 CpG 岛存在于所有组成型表达的管家基因中,但这些 CpG 岛处于组成型非甲基化状态;另外一半出现在部分 (<40%) 组织特异性调控基因的启动子中。
-
完全甲基化的位点是回文序列,其在两条 DNA 链上被甲基化。在 CpG 双峰的两条链上的胞嘧啶上发现大多数 DNA 甲基化。
-
半甲基化位点是仅在一条 DNA 链上甲基化的回文序列。复制将完全甲基化的位点转换为半甲基化位点。
-
去甲基酶是除去 DNA、RNA 或蛋白质甲基的酶。
-
甲基转移酶(甲基化酶)是向小分子、蛋白质或核酸等添加甲基的酶。
DNA 甲基转移酶

● 甲基化转移酶 (DNA methylase, DNMT):包括日常型甲基化转移酶 和从头合成型甲基化转移酶 。
-
日常型甲基化转移酶 (maintenance DNMT) 可以在甲基化母链模板的指导下甲基化新合成链的相应位点,使 DNA 迅速由半甲基化状态转变为完全甲基化状态,即参与甲基化的维持 (maintenance) ,催化速度很快,但酶的效率有限,有时不能完全把两条链上的甲基化位点都甲基化,此时会发生表观遗传特性改变。

-
从头合成型甲基化转移酶 (de novo DNMT)可以催化 CpG 成为 mCpG,此过程不需母链指导,但速度很慢。这一类甲基化酶主要调控特异基因的甲基化,在基因表达的表观遗传学调控中起十分重要的作用。

● 两种去除 DNA 甲基化的方法:
- 去甲基化酶
- DNA 复制
甲基化 CpG 结合蛋白 (
MBD: methyl-CpG binding domain proteins) ——抑制转录

● DNA 甲基化后,会招募能识别 DNA 甲基化的蛋白质结合 CpG,从而抑制转录。
MeCP1(Methyl-C binding Protein 1):结合到单个甲基化的 CpG 碱基上MeCP2(MethyI-C binding Protein 2) 和MBD1-4:多结合在 CpG 岛上
● 保守结构域:
- methyl-CpG-binding domain(
MBD) 甲基化 CpG 结合结构域,与甲基化 CpG 结合; - transcription repression domains(
TRD) 转录抑制结构域,与各种共抑制因子复合物相互作用,如 mSin3A-HDAC1, c-Ski 和组蛋白甲基转移酶。
甲基化调控基因转录的两种机制
- DNA 位点被甲基化后抑制转录因子结合到 DNA 上。
- DNA 甲基化可以使特定的转录阻遏物结合到 DNA 上:MeCP2 通过与具有组蛋白去乙酰化酶活性的 Sin3 阻遏物复合体结合,从而降低组蛋白乙酰化水平,抑制基因的转录活性。
DNA 的甲基化与组蛋白的修饰互相作用:
DNA methylation induces histone de-acetylation DNA 甲基化诱导组蛋白去乙酰化:MeCP2 结合甲基化的 DNA 后,招募去乙酰化酶 HDAC 复合体 Sin3,使得组蛋白发生去乙酰化。

组蛋白甲基化和 DNA 甲基化是相互联系的
Histone H3-K9 methylation induces DNA methylation 组蛋白 H3K9 甲基化诱导 DNA 甲基化:(Occur in heterochromatin regions) 发生在异染色质区域。组蛋白 H3K9 甲基化后,招募识别组蛋白甲基化修饰的蛋白 SP1,SP1 招募 DNMT,使得组蛋白上的 DNA 发生甲基化。

- SUVAR39H: 组蛋白甲基转移酶 (H3-K9 上的甲基化)
- HP1: Heterochromatin-associated Protein 1 异染色质相关蛋白
★ 多种组蛋白修饰(尤其是抑制性修饰) 最终均可以导致 DNA 的甲基化,从而更有效、更持久地抑制基因表达。
研究 DNA 甲基化的方法
-
通过甲基化敏感的限制酶消化分离基因组 DNA
-
Bisulfite sequencing:PCR 扩增亚硫酸氢盐处理的 DNA。
数百个序列:直接测序
全基因组序列:微阵列分析/焦磷酸测序

-
(更简单常用)用识别 mC 的抗体通过亲和纯化分离的甲基化 DNA 片段,后全基因组深度测序。
DNA 甲基化的分布
- 染色体水平上,DNA 甲基化在着丝粒附近水平最高。
- 基因水平上,DNA 甲基化高水平区域涵盖了多数转座子,假基因和小 RNA 编码区。
- 拟南芥 DNA 甲基化的代表性实例

- 启动子中甲基化的基因倾向于以低水平表达;
编码区中甲基化的基因以更高水平组成型表达。

讨论: 如何对基因组进行定点表观遗传修饰和功能研究?
10.2 转录后水平的调控 (post-transcriptional regulation)
10.2.1 前体 RNA 加工
● 真核生物 mRNA 的修饰过程:
- 加 5’帽
- 加 3’ poly-A 尾
- RNA 剪接:从 pre-mRNA 中去除内含子
● RNA 加工调控(前体 RNA→成熟 RNA)两种方式:
A) poly(A) 位点的选择:产生不同的前 mRNA 分子。
B) 剪接位点的选择(可变剪接) : 产生不同的功能性 mRNA。
➢ 选择性加 A 尾独立于可变剪接。
➢ 选择性加 A 尾或可变剪接的产物是由相同基因编码但在结构和功能上不同的蛋白质:蛋白质亚型。
➢ RNA 加工可以是组织特异性的。
由 PolyA 和剪接位点控制-人降钙素 (calcitonin) 基因 (
CALC)
- 降钙素 (
calcitonin):甲状腺和大脑的某些神经元分泌的参与钙剂骨质代谢的一种多肽类激素,对破骨组织细胞有急性抑制作用,能减少体内钙由骨向血中的迁移量。 - CALC 含有 5 个外显子和 4 个内含子
- 选择性加 A 尾在甲状腺细胞中加在第 4 外显子旁边,而在神经细胞中 PolyA 位点位于第 5 外显子旁边。
- 甲状腺细胞中内含子 1,2,3 和 4 拼接在一起,而在神经元细胞中 1,2,3 和 5 内含子选择性剪接在一起。

剪接位点选择调控——果蝇性别决定
- 性别决定:X:A ratio is 1 (2X:2A) = Female, 0.5(1X:2A) = Male
X:A 比率是生物体中 X 染色体数量和常染色体组数量之间的比率。 - X:A 比率信息被传送给性别决定基因,这些基因决定了女性和男性发育途径之间的选择,调控从主控基因 Sxl(性致死)开始。
- Sxl 基因表达:Sxl 基因含有 8 个外显子,第 3 个外显子含有一个终止密码子。
- 在 female 中,剪接除去 #3 外显子以及所有内含子 → 功能性 SXL 蛋白
- 在 male 中,选择性剪接去除内含子而不是 #3 外显子 → 没有功能性 SXL 蛋白质

- Tra 基因表达:Tra #2 外显子前有终止密码子
- 在 female 中,有 SXL 蛋白,使 #2 外显子前的终止密码子发生剪接 → 有功能的 TRA 蛋白质
- 在 male 中,没有 SXL 蛋白,不发生剪接,#2 外显子前的终止密码子使得翻译提前终止 → 无 TRA 蛋白质产生

- dsx 基因表达:发生选择性剪接和加 A 尾
- 在 female 中,有 TRA 蛋白,在 #4 外显子后加 A 尾 → DSX-F 蛋白
- 在 male 中,没有 TRA 蛋白,#4 外显子被剪接去除,并在 #6 外显子后加 A 尾 → DSX-M 蛋白

10.2.2 mRNA 转运
真核生物 mRNA 的修饰和转运出核过程
- <1min: 转录开始,5’加帽
- 6min: 3’ end of mRNA is released by cleavage
- 20min: 3’加 polyA 尾
- 25min: mRNA 转运出核至细胞质
- 大于 240min: 核糖体翻译 mRNA
RNA 转运调控
- 调节从细胞核离开到细胞质的转录物数量。
- 可能,有高达 50% 的蛋白质编码 RNA 的主要永远不会离开细胞核,被降解。
snRNPs(核小核糖核蛋白,small nuclear ribonucleoprotein) 颗粒对于保留细胞核中的 mRNA 很重要。
剪接体保留模型
- 剪接体:剪接复合物,由前体 mRNA 和 snRNPs 组成,与 mRNA 核输出竞争
- 剪接过程后,内含子在降解前与 snRNPs 结合
- 甲基化的 5’帽是 mRNA 输出至细胞质所必需的;没加 5’帽的 mRNA 在核内起调控作用。
- HIV Rev 蛋白对核出口的调控
- RNA 中的内含子阻止转运
- Rev 结合内含子,协助输出
10.2.3 翻译
翻译时间上的调控
- 被掩盖的 mRNAs(
masked mRNAs):未受精卵中储存的 mRNA 与蛋白质相关,保护它们免受 RNase 降解以及抑制翻译。 - 受精后,抑制在几分钟内即可缓解。
- 受精后蛋白质合成速率显著增加。
Poly(A) 促进翻译
- polyA 尾巴已知可促进翻译启动。
- 一般而言,储存的非活性 mRNA 比活性 mRNAs(100-300As) 具有更短的 PolyA 尾巴 (15-90As)
- 较短的尾巴是合成时就比较短还是开始较长后来被截短了?
在小鼠/青蛙的卵母细胞中,前 mRNA 具有长尾巴 (300-400As)
成熟的储存 mRNA A 尾短 (40-60 As)
积极翻译的 mRNAs 有 100-300As
细胞质中调控去腺苷酸化和多聚腺苷酸化信号
- 去腺苷酸化:在 mRNA 的 3’UTR 中,在 AAUAAA 序列的上游,存在作为去腺苷化信号 (UUUUUAU) 的富含共有序列 (AU) 的元件 (
ARE)。 - 多聚腺苷酸化:激活一个存储的 mRNA, 该信号 (ARE 元件)由聚腺苷酸化酶识别并添加 ~ 150As。
10.2.4 RNA 降解
mRNA 降解调控 (RNA turnover)
- rRNA 和 tRNA 都非常稳定,但不同 mRNA 稳定性差异大
- 调节信号改变 mRNA 的稳定性
- mRNA 二级结构和 ARE 序列(富含 AU) 也影响 mRNA 半衰期
mRNA 降解途径
● 去腺苷酸化依赖性降解途径 (没有 A 尾时)
- 酶催化过程
- 聚 (A) 尾部去腺苷酸化直到尾巴太短 (5-15As),不能与 PAB (polyA 结合蛋白)结合
- 5’帽被移除(脱帽) (DCP1)
- 5’至 3’外切核酸酶降解
● 去腺苷酸化不依赖性降解途径 (有 A 尾时)
- 酵母 dcp1 突变体能够降解 mRNA
- 脱帽而不脱腺苷
- 快速降解通过 5’至 3’外切核酸酶
- 或在内部切割后进一步降解
RNA 干扰 (RNA interference : siRNA and miRNA)
● 短干扰 RNA(Short/small interfering RNA, siRNA)

- 19-nt 双链
- 2-nt 3’末端突出
- 5’-磷酸基团
- 3’-OH: 在动物中 3’-OH 被修饰后便失去活性
- 植物 siRNAs 3’甲基化,可能保护或稳定了 siRNA
- 分为引导链与乘客链 (Guide Vs passenger strands)。引导链介导 mRNA 的降解。乘客链在 siRNA 形成有功能的复合物之前就被降解。
- 具有双链的不对称性
➢ siRNA 的发现:早在 1988 年就有 RNAi 的现象报道。当时的研究人员希望通过转基因过表达的手段使得牵牛花的颜色变深,但在他们得到的诸多转基因牵牛花中,有些花不但颜色没有更深,反而变成白色。相类似现象也在其他转基因植物中出现,被称为基因表达的共抑制现象 (co-suppression)。
- 不论引入靶基因(查尔酮合成酶)正义链 (Sense RNA) 和反义链 (antisense RNA) 都能导致内源靶基因表达量降低
- 正义链+反义链有更强的效果
- 在线虫和果蝇中,该现象被称为 RNA 干扰。
● RNAi 的重要特性是以双链 RNA 行使功能
- 线虫性腺细胞中显微注射 MEX3 的反义 mRNA 和 dsRNA,然后用原位杂交技术检测子代胚胎时期的线虫内源 MEX3 RNA
a) 正常胚胎 (Negative control),对照;
b) 未注射 RNA, MEX3 表达量很高;
c) 注射反义 RNA: MEX3 表达量明显降低;
d) 注射双链 RNA:几乎检测不到 MEX3 的表达。 - 实验表明,双链 RNA 对内源基因表达的干扰效率远高于单链的 RNA,真正起到 RNA 沉默作用的应该是双链 RNA。此外,研究还发现,引入的外源 RNA 只对同源基因有高沉默效率,暗示它们之间可能需要形成配对的基础。
- 2000 年,果蝇细胞提取物的体外 RNAi 系统,发现不论是否有靶 mRNA 存在,引入的外源 dsRNA 的正、反义链都会被切割成 21-23nt 的小片段,相对应的 mRNA 也会被降解为长度差为 21-23nt 的片段,说明这种降解很可能是由 21-23nt 小片段介导的,并且这种降解需要 ATP 提供能量。
➢ siRNA 的生物合成(来源)
- 病毒 RNA 以及环境、实验等因素引入的外源 RNA
- 基因组重复片段
- 自然存在的双链 RNA
- 转座子★

● RNA 沉默的主要调控分子
- RNaseⅢ Proteins: Dicer-1, Dicer-2, Drosha
- dsRNA-Binding Domain Proteins: R2D2, Pasha, Loquacious
- Argonaute Proteins: Argonaute-1, Argonaute-2, Argonaute-3, Piwi, Aubergine
RISC 的形成过程(三个核心步骤)

(1)经 Dicer 切割形成双链小片段:
-
Dicer 是一类RNaseⅢ蛋白,这类内切核糖核酸酶能特异识别双链,通常含有
解旋酶 Helicase 结构域、PAZ 结构域、一对RNaseⅢ结构域、双链 RNA 结合域(dsRBD)。
-
PAZ结构域(110aa domain found in Piwi, Ago, Zwille & Dicer proteins)结合双链 RNA 3’端 2 个不配对核苷酸(2nt overhang),PAZ domain 对 3’的 2nt 不具有特异性。 -
两个
RNaseⅢ结构域形成分子内二聚体结构,各催化剪切一条链,产生双链断裂。 -
dsDBD 的结合位点不特异。
-
PAZ 结构域和 RNaseⅢ催化切点约相距 6.5nm(65 埃), 与 20 多个核苷酸的长度相当,因此,Dicer 作为分子尺子,将 dsRNA 加工成 21nt 的 siRNA。
(2) R2D2 的装配:siRNA 的装载需要双链 RNA 结合蛋白 R2D2 的帮助。
- R2D2 包含两个一前一后的的双链 RNA 结合结构域。
- R2D2 与 siRNA 热稳定性较高的 3’端结合。
- siRNA 的不对称性 (
siRNA asymmetry) : 引导链的 5’端稳定性较差。 - Dicer-2 和 R2D2 形成异源二聚体,
Dicer/R2D2/siRNA三者形成 RISC 装载复合物(the RISC-Loading Complex,RLC)。 - R2D2 招募 Argonaute 蛋白,开始组装 RISC。
(3)沉默复合物 RISC(RNA-Induced Silencing Complex) 的装配和成熟:
- Argonaute 与 Dicer 可以发生蛋白质-蛋白质相互作用,Argonaute 首先与 Dicer 交换,结合到 siRNA 双链的一端,然后与 R2D2 交换,将整个双链小 RNA 都转载到 Argonaute 中。此时,Argonaute 将乘客链降解,形成有功能的沉默复合物。
- Argonaute 蛋白 (
Ago) 有 PAZ、MID、PIWI 三个结构域:

PAZ domain:结合引导 RNA 3’末端不配对的 2nt(与 Dicer 相同)。MID domain:特异性识别引导 RNA 的 5’端。MID domain 的磷酸基团和 MID 结合口袋中的氨基酸残基形成氢键,这种 RNA-蛋白质的相互作用需要二价阳离子 (Mg2+)。PIWI domain:采用 RNase H 折叠(具有 5 个β折叠并缠绕着一系列 α 螺旋),因此被认为可能是具有 RNaseH 活性的结构域,可催化剪切 passenger strand 和 mRNA。
● 活性 RISC 复合物的产生过程
- 活化的 RISC 复合物(guide strain 和 Ago 形成的复合物)含有与 Dicer 产生的 siRNA 的引导链相关的 Argonaute 。
- ① Dicer 切割;
② Dcr-2(Dicer-2)/R2D2 异源二聚体与 siRNA 形成 RISC 加载复合体 (the RISC-loadingcomplex,RLC).
③ Dcr-2 and R2D2 招募 Ago2 直接到双链 siRNA.
④ Ago2 先交换 Dcr-2, 后交换 R2D2, 与 siRNA 结合。
⑤ Ago2 催化酶切过客链(蓝色),从而从 siRNA 双链体释放指导链(红色)并产生活性 RISC。

切割催化模型
切割活性的体外证明:

- siRNA 引导链在 5’末端与 Ago2 的
MID/PIWI结构域结合,在 3’末端与PAZ结构域结合。 - 靶标 mRNA 首先被 siRNA 的种子区域(接近 5’末端)结合,配对延伸到 3’末端。
- 切割 mRNA 在 siRNA 的 5’端 10nt 位置。
- 切割位点:

siRNA 的功能——(1) 降解 mRNA

- 人类 Ago2 与 2 种 siRNAs 和 1500nt RNA 靶标混合
- 产生预期大小的产物,取决于 siRNA、靶 RNA 和 Mg2+
● 在一些生物体中,siRNA 信号被放大并扩散

-
RNA 依赖的 RNA 聚合酶 (RNA-dependent RNA polymerase,
RDRP) 使 siRNA 继续扩增,产生次级 siRNA 放大效应。- RDRP 最早克隆自被类病毒侵染的番茄中 (1998, Plant Cell)
- 与 RNA 病毒编码的 RdRP 基本没有同源性
- 以 RNA 为模版的 RNA 扩增机制可见于植物和线虫,在果蝇和哺乳动物基因组中不存在
- 是产生次级 siRNA 的放大效应的主要因子
-
以 RNA 为模版的 RNA 扩增机制(
RDRP的作用机制)

- A. 通常情况下,RNA 不会沉默,因为 RDR 蛋白质无法接近模板 RNA 序列。成熟的真核 mRNA 与各种蛋白质结合,如帽结合蛋白 (CBP) 和聚腺苷结合蛋白 (PABP) 可能限制 RDR 的结合。
- B. RNA 缺乏 5’帽或 3’多聚 A 尾,允许 RDR 蛋白接近并以 RNA 为模版,扩增出 dsRNA, 进入 siRNA 途径(经 Dicer 切割等步骤产生 siRNA, 来降解内源 RNA)。这个过程也可视作机体清除错误 mRNA 的机制。
- C. 对于结合蛋白完好的 mRNA, 如果体内有来自病毒、转座子或细胞 RNA 中的少量的初级 siRNA, 可以通过碱基配对与靶 RNA 退火,并用作 RDR 的引物以 mRNA 为模版合成双链 RNA, 产生新的 RISC, 新扩增产生的各种次级 siRNA 可以与靶 mRNA 的不同区域配对,更大范围的降解靶 mRNA。
➢ RNAi 的生物学意义
- 保护基因组免受外源核酸侵入
- 维持基因组稳定
- 参与基因表达调控
● 非细胞自主的 RNA 沉默与系统性 (systemic) RNA 沉默
RNAi 作用可以向其它细胞传递,因此受到其他细胞或组织传来的小 RNA 的抑制,称为非细胞自主 (non-cell autonomous) 的 RNA 沉默,可以分为两类:
- 细胞与细胞间的传递:包括近程与远程两种。
- 系统性传递:通过传输器官(如韧皮部)在不同组织间广泛传播。
- 移动信号可能是 siRNA 或 dsRNA

● 宿主 RNA 和 siRNA 沉默途径已被认为是植物免疫的重要组成部分。
植物响应和防御病原体感染的一种方式是通过 siRNA 沉默免疫系统。
为了处理植物防御反应,病原体已经发展成熟的机制来避免和反击这种防御策略。
VSR: Viral Suppressors of RNA silencing
siRNA 的功能——(2) RNA-Directed DNA Methylation(
RdDM) 沉默转座子或甲基化重复序列
RdDM主要通过 dsRNA 介导相同 DNA 序列发生重新甲基化 (de novo methylation) 而实现转录水平的基因沉默。- 引起相同序列 DNA 甲基化的物质是 dsRNA。
- RdDM 的生物学意义:阻抑不必要基因 (repetitive sequences) 和有害基因(尤其是 transposons) 的表达,对维护基因组的稳定至关重要。

miRNA
➢ miRNA 的发现:
- 1993 年,Lee RC 等在线虫 (C.elegans) 中意外地发现了一种定时调控胚胎后期发育的 miRNA-lin4, 它是一种非编码 RNA, 长度为 22 nt。
- 2000 年,miRNA-let7 的发现掀起了寻找 miRNA 的热潮。
- 在线虫 (C.elegans) 当中,通过功能缺失突变体的筛选,找到了 let-7 和 lin-41 基因,其中 let-7 也是长度为 22 nt 的非编码 RNA。
- 不同物种中的 let-7 基因具有序列保守性,且均可与 lin-41 基因的 3’UTR 区域互补。
➢ miRNA 的生成
● RNA PolⅡ转录 miRNA 基因(与 mRNA 的转录相似)
- 由 polⅡ 转录为 pri-miRNA(主要前体)
- Pri-miRNA 含有 7-甲基鸟苷帽和 poly (A) 尾
- PolⅡ 与 miRNA 基因启动子结合
- miRNA 基因转录对 α-amanitin(鹅膏蕈碱)敏感
- PolⅡ 依赖性转录使 miRNA 产生的时间和空间调节成为可能。
● 形成成熟 miRNA 的基本过程:
- 先由 RNA PolⅡ 产生的较长的初级转录产物 pri-miRNA, 中间有一段不完美配对的茎-环结构;
- 经过第一步切割产生 pre-miRNA;
- 再经第二步切割产生双链 miRNA;
- 双链解链形成成熟的长 21nt 左右的单链 miRNA。
- 参与整个过程的基因和切割过程在动、植物中略有不同。
- 动物中,miRNA 前体的两次切割都需要有 RNaseⅡ 核酸内切酶以及双链 RNA 结合蛋白完成。
◆Drosha是一种RNaseⅢ, 将pri-miRNA的 3’端和 5’端切割产生长约 70nt、5’端带磷酸基团、3’端为羟基的 miRNA 前体pre-miRNA, 同时保留茎-环结构区,并且切割端具有 2~3nt 的 3’不配对碱基,这是由 RNaseⅢ 的性质所决定的。
◆ Drosha 的正确切割还需要一类双链 RNA 结合蛋白的帮助,在果蝇中是Pasha蛋白,在线虫中是Pash-1蛋白,而在哺乳动物中则为DGCR8蛋白。
◆ 切割后的前体pre-miRNA经由Exportin5/RanGTP运出细胞核进入胞质基质中。胞质基质中的 RNaseⅢ 为Dicer, 切割 pre-miRNA 茎-环结构环的一端形成成熟的长约 21nt 的双链 miRNA, 即miRNA-miRNA*, 其中的 miRNA 链是之后真正行使功能的成熟 miRNA 链。有研究认为,哪条链作为 miRNA 由其 5’端热不稳定性所决定,5’端相对不稳定的链更有可能成为 miRNA 。 - 植物中 没有 Drosha 的同源基因,pri-miRNA 的两步切割由 Dicer 的同源基因 Dicer-like1(
DCL1) 完成。DCL1 在细胞核内,pri-miRNA 经 DCL1 两步切割,两端形成双链 miRNA, 由 Exportin5 的同源基因HASTY运输出细胞核。与动物中的 miRNA 不同,植物双链 miRNA 两个 3’末端的自由羟基被甲基转移酶HEN1甲基化,以羟甲基的形式存在。甲基化的 3’末端被保护不受降解,还有助于形成成熟的 RISC。

- 动物中,miRNA 前体的两次切割都需要有 RNaseⅡ 核酸内切酶以及双链 RNA 结合蛋白完成。
● miRNA 的选择性装载:
- 与 siRNA 一样,单链 miRNA 可以装载在 RISC 中介导转录后沉默的发生。
- RISC 的装载对 5’端的碱基有所选择,大部分 AGO 蛋白倾向于结合 5’端为 U 或者 A 的 miRNA。
➢ miRNA 的功能
(1)miRNA 介导的 mRNA 降解:和 siRNA 一样装载成 RISC 后使互补配对的 mRNA 降解,miRNA-RISC 的原理和 siRNA 相似,但由此介导的基因沉默还需要一些其他功能保守的元件参与。
-
果蝇的研究发现,靶 mRNA 的降解除了需要 AGO 蛋白以外,还需要
GW182、脱腺苷基因 (NOT1/CAF1)、脱帽相关基因 (DCP) 等,说明这种转录后沉默机制 需要经历“脱帽脱尾"事件。 -
GW182蛋白介导 miRNA 效应:- GW182 与 Ago 蛋白结合(AGO 招募 GW182)。
- GW182 与 polyA 结合蛋白(此处为
PABP或 PABPC1) 结合。 - GW182 中断 eIF4G 和 PABP 的结合,从而破坏 mRNA 的环化。
- 开放的 mRNA 经历去腺苷酸化然后脱落。

-
mRNA 成环→翻译活跃;mRNA 闭环→翻译抑制
(2)miRNA 可抑制 mRNA 的翻译,降低靶基因的蛋白水平但不影响其 mRNA 水平。
- miRNA 介导的翻译抑制可能发生在不同阶段。
- 可能是通过形成 miRNA:mRNA 配对分子,阻碍有功能的核糖体与 mRNA 结合、装配,发生核糖体的“drop-off"。
- 可能发生在翻译起始阶段,miRNA 可能通过抑制翻译起始因子与 5’帽子相结合的方式影响核糖体的组装。
● microRNA 靶作用位点——在动物当中的预测和验证
- 动物 miRNAs 只与它们的靶位点保持很低的互补性:仅为 miRNAs 中 2-7nt(称为种子序列
seed sequence),且决定了 miRNA 的功能。
➢ 在无脊椎动物 miRNA 的 miRNA 靶位点内,与 miRNA 的 2-7 位核苷酸配对的残基在其他物种的直系同源 mRNA 中是保守的。
➢ miRNA 的核苷酸 2-7 在同源多细胞动物 miRNA 中是最保守的。
➢实验证据还表明,siRNA 中的核苷酸 2-7 在引导切割中比其他核苷酸更重要 - 与种子序列配对是必要的
- 在 12-17 位碱基的额外配对增强了 miRNA 靶向性
- 结合站点的偏好性:富含 AU 区域

-植物 miRNAs 和靶 mRNA 具很高序列互补性,因此可以利用类似于 siRNA 介导的剪切机制去剪切靶 mRNAs
● 如何研究 miRNA 的功能?(当成基因来研究)
- miRNA 基因敲除突变体
- miRNAs 基因过表达突变体
- 在内源启动子作用下表达 miRNA-resistant targets
- 约半数预测的保守 miRNA 的靶位点都存在于转录因子的 mRNAs 中(转录因子只占基因的 6%)
➢ miRNA 的生物学功能(调控生理和发育过程)
● 植物 miRNAs
- 通过靶向转录因子 mRNAs, 许多 miRNA 在细胞分化和发育模式中发挥作用
- 其他 miRNA 靶向非转录因子 mRNA, 可能在生理过程或应激反应中发挥作用
- dcl1 无效突变体的胚胎致死表型说明 miRNA 具有非常重要的功能
● 动物 miRNAs
- 发育方式
◆ 缺乏 Dicer 的 ES 细胞是可存活的,但不能在体外和体内分化。
◆ Dicer 基因敲除斑马鱼胚胎发育有障碍 - 生理功能
- 癌症
● RNA 指导的从头甲基化 (RdDM)
双功能 MIR 基因的模型:转录后产生两种产物
① 20-22nt miRNA:结合 AGO1,抑制 mRNA 翻译;
② 23-27nt siRNA:结合 AGO4,使得靶基因 DRM1/2 发生甲基化,抑制靶基因转录。
➢ miRNA 与 siRNA 比较
- miRNA 与 siRNA 有许多共同之处。都是小分子 RNA,在真核生物中基因表达的调控中发挥重要作用。它们在正常细胞新陈代谢、发育和防御侵略者方面很重要。
- siRNA 由 Dicer 从更长的 dsRNA 片段产生,将其组装到 RISC 中,并且靶向具有完全互补性的 mRNA, 通过 RNA 的切割和降解或通过形成异染色质产生沉默。
- miRNA 是通过基因转录,然后经 Drosha 切割再由 Dicer 从发夹结构加工产生的,并且它们通常与它们的靶标 mRNA 具有不完全互补性,从而对翻译产生影响,而非 Ago 介导的 mRNA 裂解。
10.2.6 蛋白质降解
● 蛋白质降解(水解)控制
-
合成和降解之间的平衡
-
不同的蛋白质具有不同的寿命
组成型基因:蛋白质寿命可短暂
短寿命 mRNA: 其蛋白质可以有较长的寿命
受体与热休克蛋白:具有短的半衰期 -
在真核生物中,蛋白质降解需要辅因子泛素 Ubiquitin
-
Ubiquitin泛素:~ 8kd 保守的多肽,C-末端 Gly 与靶蛋白 Lys 的-NH2 相互作用。 -
泛蛋白与蛋白质结合作为识别标签,使蛋白质被蛋白水解酶降解,泛素在降解过程中完整释放。
● 泛素化系统

- E1: 泛素激活酶
E2: 泛素结合酶
E3: 泛素-蛋白质连接酶 - 被标记降解的蛋白质通常需要几个泛素分子
- 在蛋白酶体(
Proteasome)降解:泛素依赖的降解、泛素非依赖的降解
● N 端法则
-
特定的 N-末端 AA(第一个氨基酸) 直接与蛋白质的半衰期(
half life)有关。

-
N-末端 AA 指导泛素分子与蛋白质结合的速率并决定半衰期。
思考题
- 植物细胞中 siRNA 的 off-target 可能会产生什么影响?
- 如果在哺乳动物细胞中引入 RDR(RNA 依赖的 RNA 聚合酶) 会怎么样?