一、背景
关于耐药性有很多数据库可以使用,但是CARD的优势在于数据库较为准确,只有经过文章进行验证后的基因才会被记录
二、软件链接
软件github:
GitHub - arpcard/rgi: Resistance Gene Identifier (RGI). Software to predict resistomes from protein or nucleotide data, including metagenomics data, based on homology and SNP models.![]() https://github.com/arpcard/rgi#check-database-version软件文章:
https://github.com/arpcard/rgi#check-database-version软件文章:
CARD 2017: expansion and model-centric curation of the comprehensive antibiotic resistance database | Nucleic Acids Research | Oxford Academic (oup.com)![]() https://academic.oup.com/nar/article/45/D1/D566/2333912软件web版本:
https://academic.oup.com/nar/article/45/D1/D566/2333912软件web版本:
The Comprehensive Antibiotic Resistance Database (mcmaster.ca)![]() https://card.mcmaster.ca/home
https://card.mcmaster.ca/home
三、下载安装
在linux系统上安装较为简单,可以直接使用conda进行安装
conda create --name rgi
conda install --channel conda-forge --channel bioconda --channel defaults rgi可以直接下载软件后手动进行安装
########安装
wget https://card.mcmaster.ca/latest/software
tar -jxvf card-software.tar.bz2
tar -zxvf 5.2.1.tar.gz
cd rgi-5.2.1/
conda env create -f conda_env.yml
conda activate rgi
pip install . -i https://pypi.tuna.tsinghua.edu.cn/simple
四、具体使用
使用时需要先添加对应的数据库
wget https://card.mcmaster.ca/latest/data
tar -xvf data ./card.json
######local版本
rgi load --card_json /path/to/card.json --local
######system版本
rgi load --card_json /path/to/card.json添加数据库后rgi有三种使用方式,对应不同的数据
RGI main (Genomes, Genome Assemblies, Metagenomic Contigs, or Proteomes)
RGI bwt (Metagenomic Short Reads, Genomic Short Reads)
RGI kmer_query (K-mer Taxonomic Classification)
后两种目前暂未发表
rgi main -i test.fa -o test -t contig --include_loose --local --clean其中-t 为模式可选择输入contig或是蛋白序列,--include_loose输出时会有loose的结果,--local是使用前面添加的local数据库,--clean会将对应的临时文件删除只留下结果文件
如果为contig软件会自动进行ORF的查找,使用Prodigal软件进行查找,如果无法找到对应的ORF,则不会进行后续的分析,所以可能会导致假阴性的结果,并且软件默认小于30bp的不会进行分析,如果contig的质量不好以及序列较短,需要加上--low_quality参数,如果提交的是蛋白序列则会跳过ORF预测这一步,直接进行数据库的比对,使用的是blast和diamond,默认是blast,查找对应的同源基因,其中还有剩余的多个模式protein variant model、rRNA gene variant model、protein overexpression model
最终会生成两个文件txt和json文件,文件结果在github上有具体说明
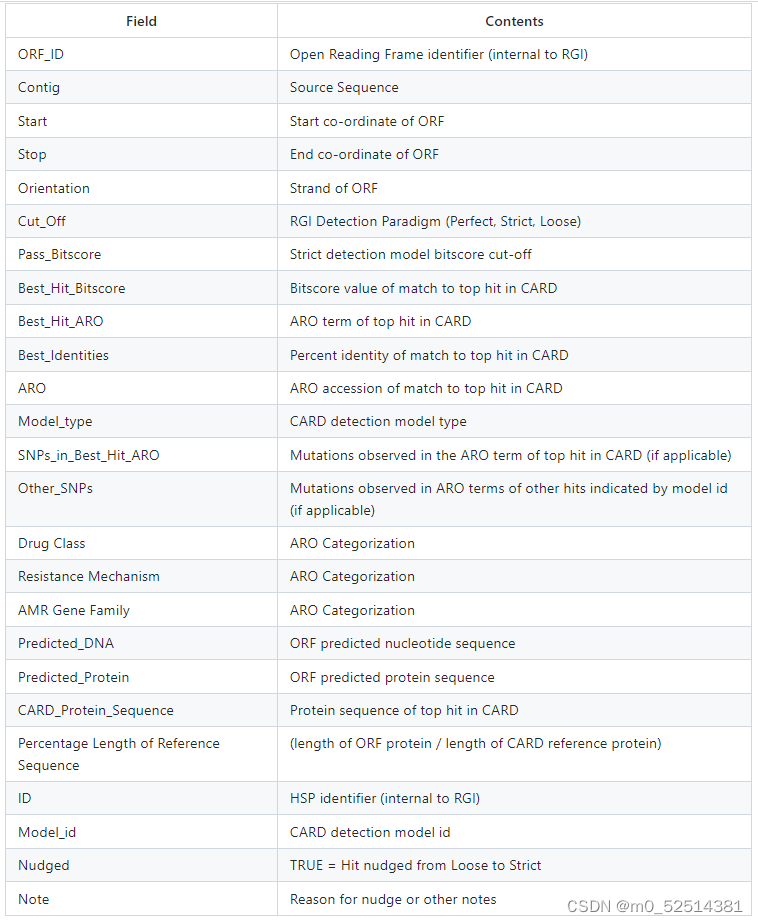
其中Best_Hit_ARO对应相关的耐药基因