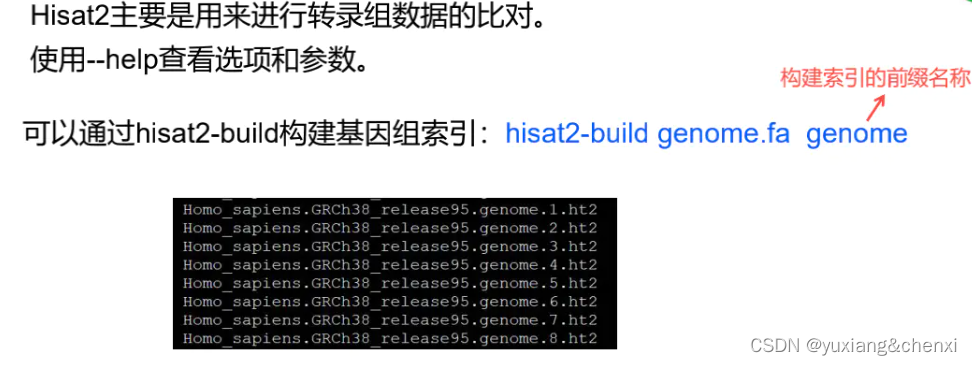
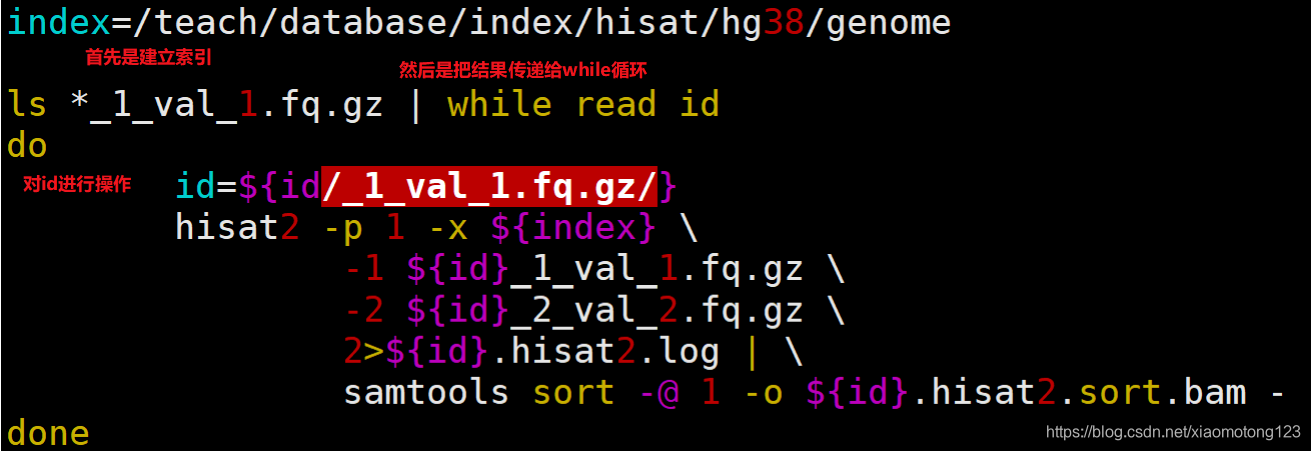
一、 拿到测序数据之后,首先选择参考基因组及比对工具进行比对
##build index
hisat2-build -p 4 Pyrus_bretschneideri_chromosome_V121121.fa genome-V1##mapping
hisat2 -x /home/xiaolong/xiaolong-data/Domestication-RNA/reference-genome/genome-V1 -p 20 --min-intronlen [int] --max-intronlen [int] -I 0 -X [int] -1 tt3_1_2.clean.fq.gz -2 tt3_1_1.clean.fq.gz -S tt1.sam >hisat2_running1_out nohup tophat -p 20 -G /home/xiaolong/xiaolong-data/Domestication-RNA/reference-genome/Pyrus_bretschneideri_Chr_gene.gff --library-type fr-unstranded -o tophat_CG1_out /home/xiaolong/xiaolong-data/Domestication-RNA/reference-genome/Pyrus_bretschneideri_chromosome_V121121 CG105_1_1.clean.fq.gz CG105_1_2.clean.fq.gz &featureCounts -T 4 -p -t exon -g gene_id -a /home/xiaolong/xiaolong-data/Domestication-RNA/reference-genome/Pyrus_bretschneideri_Chr_gene.gtf -o CG1.txt CG1.samNote: 注释文件的gff 与 gtf格式互相转换--使用gffread
通过conda安装gffread
conda install -c bioconda gffread gff to gtf
gffread anno.gff -T -o anno.gtf2. 提取每个基因上的count数,只需提取出第1列和第7列的信息
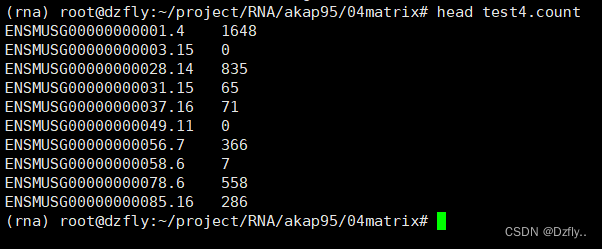
cut -f 1,6,7 CG1.txt |grep -v '^#' >CG1.count三、计算rpkm值(01.cal-RPKM.py)
#!/public/home/xiaolong/anaconda3/bin/python
# -*- coding: utf-8 -*-
"""
@File : 01.cal_RPKM.py
@Time : 2021/12/03 16:26:35
@Author : Xiaolong
@Version : 1.0
@Contact : bioinformaticc@163.com
@License : (C)Copyright 2020-2021
@Desc : None
@Usage : None
"""from __future__ import divisionimport os,sys## sys.argv[1] input count fileinf=open(sys.argv[1],"r")ouf=open(sys.argv[1]+".rpkm.xls","w")ouf.write("%s\t%s\t%s\t%s\n" % ("Geneid", "Length", "Count","RPKM"))for line in inf:breaktotal=0for line in inf:a=line.strip().split()num=int(a[2])total+=numsum=totalprint (sum)inf1=open(sys.argv[1],"r")for line1 in inf1:breakfor line1 in inf1:b=line1.strip().split()c=(int(b[2])*1000000000)/(sum*int(b[1])) ouf.write("%s\t%s\t%s\t%s\n" % (b[0],b[1],b[2],c))四、使用EdgeR做差异分析
## EdgeR 需要使用BiocManager进行安装
To install core packages, type the following in an R command window:
if (!require("BiocManager", quietly = TRUE)) install.packages("BiocManager") BiocManager::install()Install specific packages, e.g., “edgeR”, with
BiocManager::install(c("edgeR"))1. 进入R,并加载 edgeR 包载,读取read count数
library(edgeR)
data <- read.delim("CG-TTGZ.count", row.names=1, stringsAsFactors=FALSE)
head(data)
dim(data)Noter: 数据格式(CG-TTGZ.count)
GeneID CK1 CK2 CK3 T1 T2 T3
contig11g00001 3 5 5 3 10 0
contig11g00002 0 0 0 0 0 0
contig11g00003 3 0 1 2 5 2
contig11g00004 2 0 0 3 1 0
contig11g00005 54 59 43 36 35 25
contig11g00006 2 5 5 9 21 14
contig11g00008 0 0 0 0 0 0
contig11g00007 36 39 30 63 25 35
......CK对照,1 2 3 三个重复;T处理,1 2 3 三个重复。
2. 构建分组变量
targets <- data.frame(Lane = c(1:6), Treatment = c(rep("TTGZ",3),rep("CG",3)),Label = c(paste("TTGZ", 1:3, sep=""), paste("CG", 1:3, sep="")))targets3. 创建基因表达列表,进行标准化因子计算
y <- DGEList(counts=data[,1:6], group=targets$Treatment, genes=data.frame(Length=data[,6]))colnames(y) <- targets$Labeldim(y)4. 对表达量偏低的基因进行过滤
keep <- rowSums(cpm(y)>1) >= 3y <- y[keep,]dim(y)Note: 基因在至少3个样本中得count per million(cpm)要大于1
5. 重新计算库大小
y$samples$lib.size <- colSums(y$counts)6. 进行标准化因子计算 默认使用TMM方法
y <- calcNormFactors(y)yNote: 这里通过图形的方式来展示实验组与对照组样本是否能明显的分开以及同一组内样本是否能聚的比较近 即重复样本是否具有一致性
pdf("MDS.pdf")plotMDS(y)dev.off()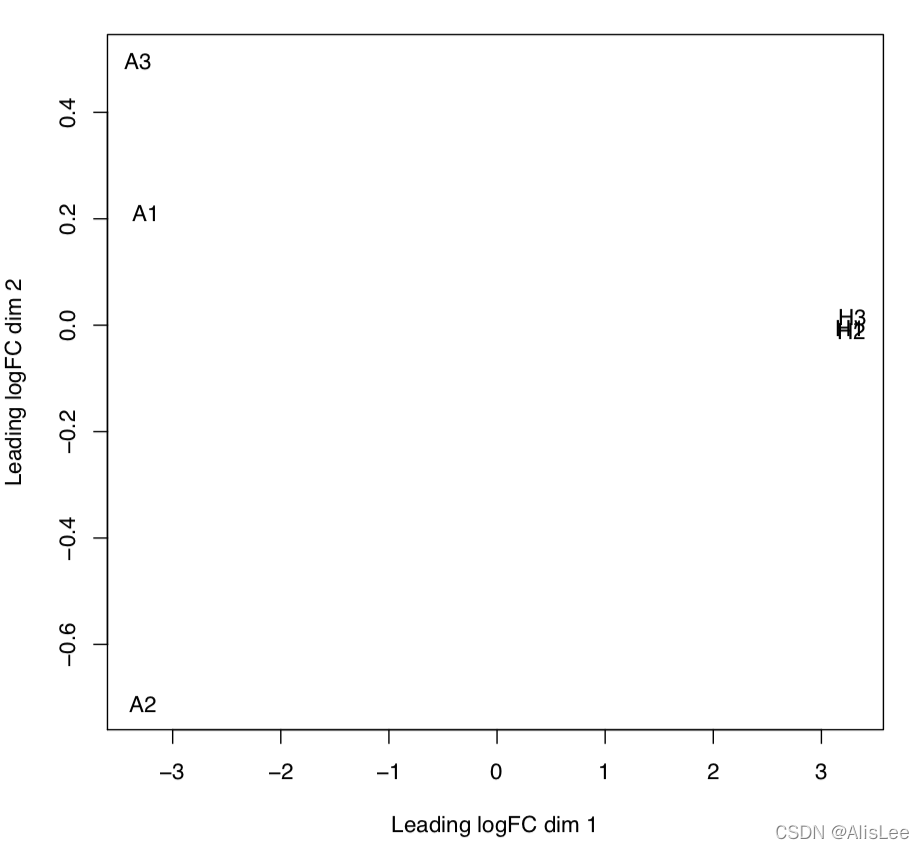
7. 估计离散度
y <- estimateCommonDisp(y, verbose=TRUE)y <- estimateTagwiseDisp(y)pdf("BCV.pdf")plotBCV(y)dev.off()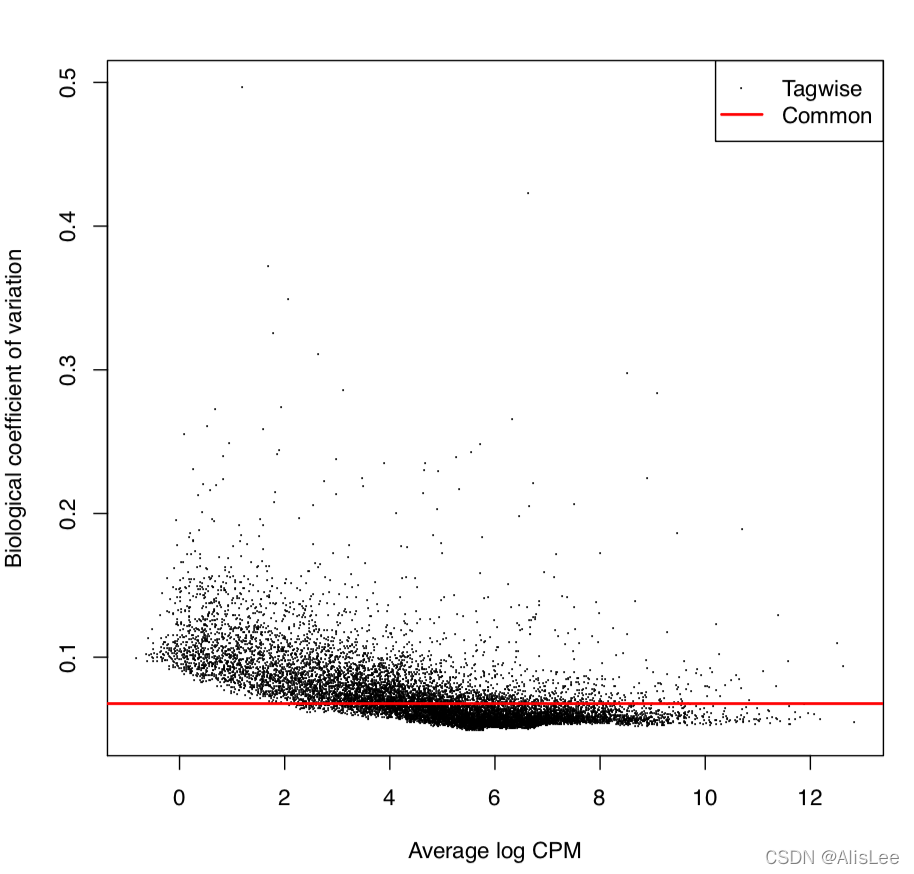
8. 通过检验来鉴别差异表达基因
et <- exactTest(y)top <- topTags(et)top9. 定义差异表达基因与基本统计
summary(de <- decideTestsDGE(et)) # 默认选取FDR = 0.05为阈值Note: 图形展示检验结果
detags <- rownames(y)[as.logical(de)]pdf("Smear.pdf")plotSmear(et, de.tags=detags)abline(h=c(-1, 1), col="blue")dev.off()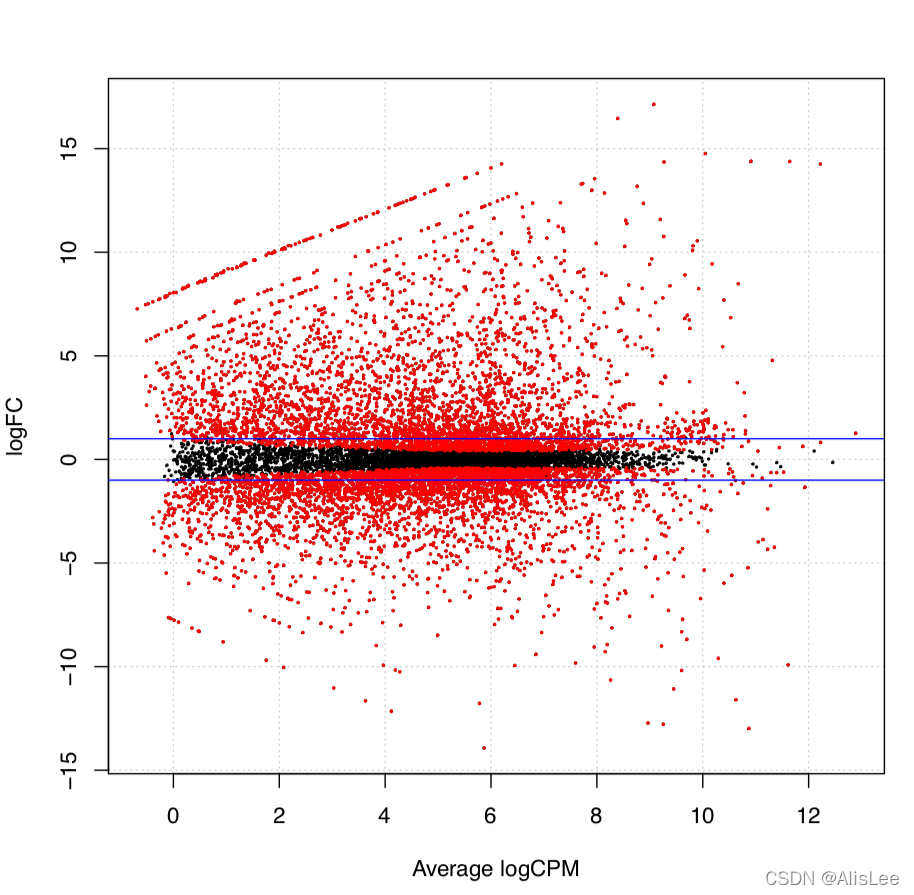
10. 差异基因结果输出
out <- topTags(et, n=Inf)write.table(out, file="DEG.txt", row.names = T, col.names = T, quote=F,sep="\t")Reference: Comparative Transcriptomic Analysis Provides Insight into the Domestication and Improvement of Pear (P. pyrifolia) Fruit | Plant Physiology | Oxford Academic



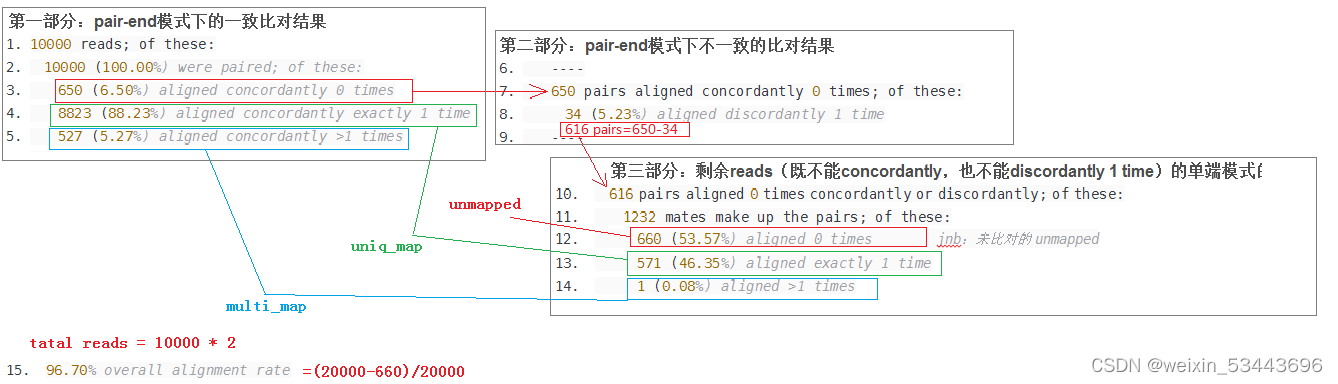
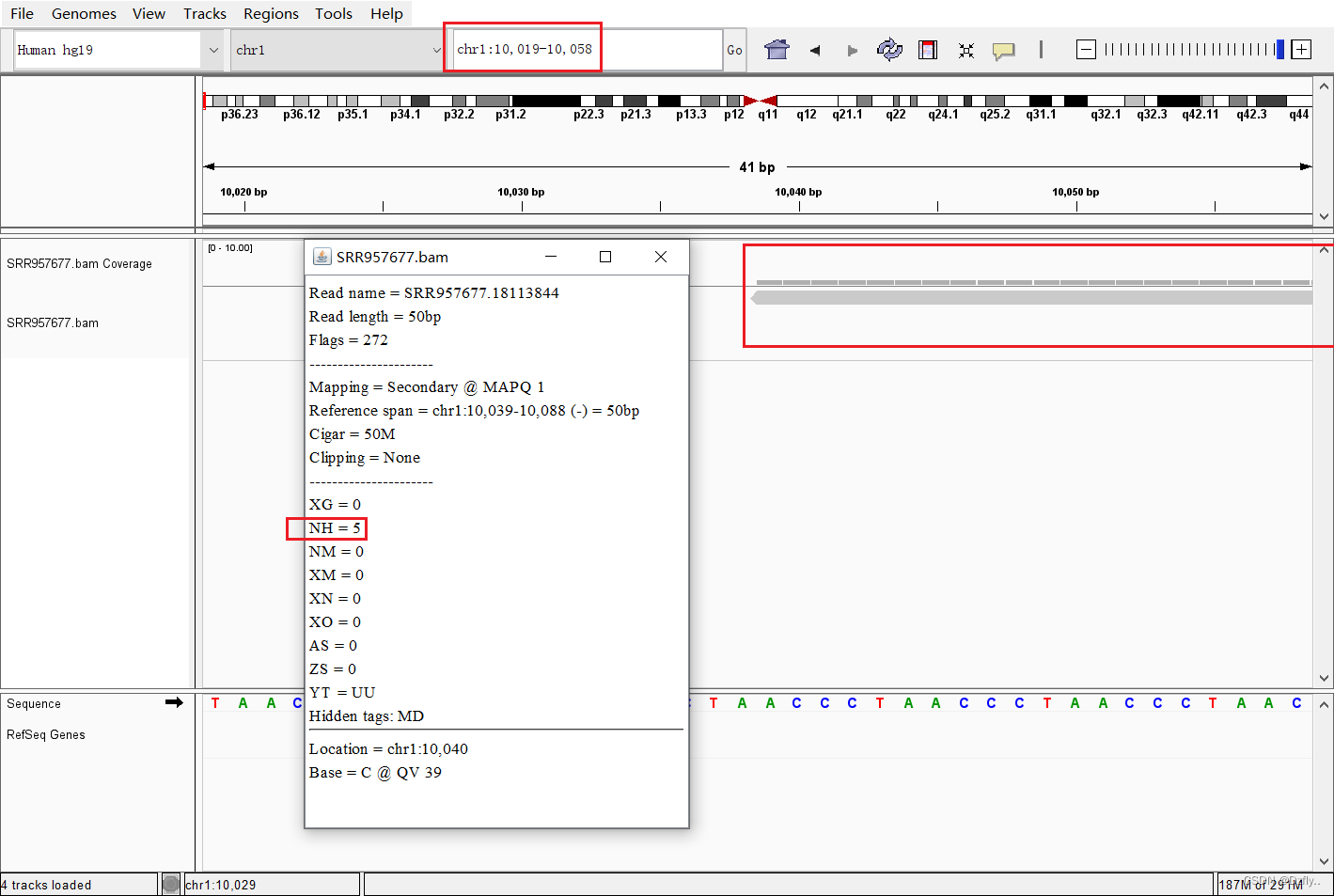

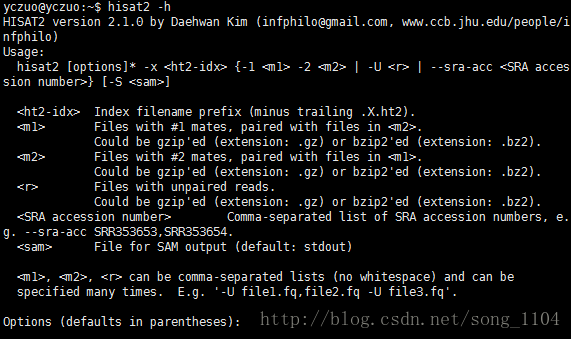

![转录组学习之序列比对(Hisat2)[学习笔记通俗易懂版]](https://img-blog.csdnimg.cn/e99c9df3c6ca436491401c75aafe334e.png)