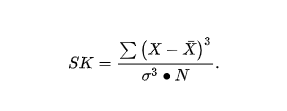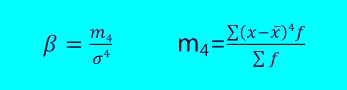机器学习算法-随机森林初探(1)
随机森林拖了这么久,终于到实战了。先分享很多套用于机器学习的多种癌症表达数据集 https://file.biolab.si/biolab/supp/bi-cancer/projections/。
机器学习算法-随机森林之理论概述
分类问题评估指标有:准确率 (Accuracy)、精准率 (Precision)、灵敏度 (Sensitivity)、ROC曲线、AUC值。
回归问题评估指标有:MAE和MSE。
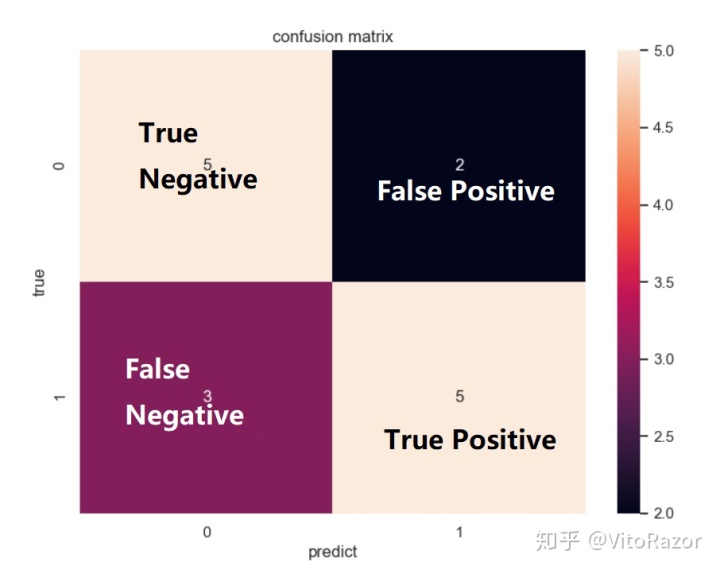
假设有下面一个分类效果评估矩阵Confusion matrix(也称混淆矩阵,总觉得这个名字奇怪),如下
行代表实际值,列代表预测值。如DLBCL组有56+2个样品,其中56个被预测为DLBCL,2个被预测为FL。
confusion <- data.frame(Predicted_as_DLBCL=c(56,8), Predicted_as_FL=c(2,11), class.error=c(0.03448, 0.4211),row.names = c("Originally_classified_as_DLBCL","Originally_classified_as_FL"))confusion## Predicted_as_DLBCL Predicted_as_FL class.error
## Originally_classified_as_DLBCL 56 2 0.03448
## Originally_classified_as_FL 8 11 0.42110准确率 (Accuracy)表示预测正确的结果占总样本的比例
计算如下:
准确率可以判断总的正确率,但在各个分组样本数目差别较大时就不能作为一个很好的评价标准。比如上面confusion matrix中,所有样品全部预测为DLBCL,则准确率可达75.3%。这一不负责任的分法跟预测模型的准确率 (87.0%)相差不是太大。
精准率 (Precision)
所有被预测为
DLBCL的样品中有多少确实是DLBCL;所有被预测为
FL的样品中有多少确实是FL;
灵敏度 (Sensitivity)
灵敏度 (Sensitivity)也称为真阳性率。
实际为
DLBCL的样品中有多少被预测为DLBCL;实际为
FL的样品中有多少被预测为FL:
不同的分组,灵敏度(Sensitivity)值差别很大,尤其是样本数目不均衡时。
假阳性率 (false-positive)
假阳性率 (false-positive)实际为1-灵敏度:
DLBCL的预测假阳性率指所有实际为FL的样本被错误的预测为DLBCL的比例;FL的预测假阳性率指所有实际为DLBCL的样本被错误的预测为FL的比例。
ROC曲线
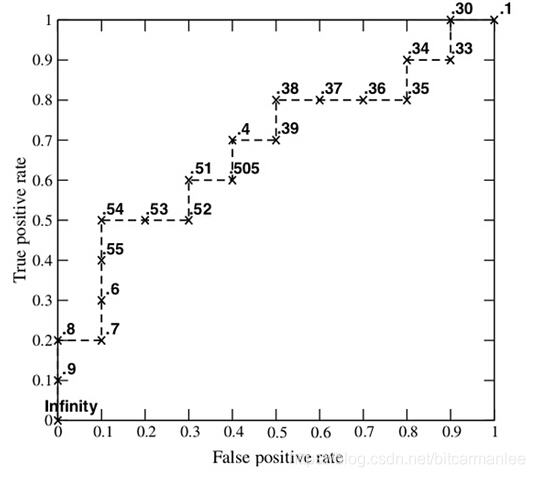
ROC (Receiver Operating Characteristic)特征曲线就是横轴为假阳性率,纵轴为真阳性率的一条曲线。这条曲线越陡越好,说明在较低的假阳性率时可以获得较高的真阳性率。
根据阈值设定的变化,模型的假阳性率和真阳性率随之变化,形成了ROC曲线。
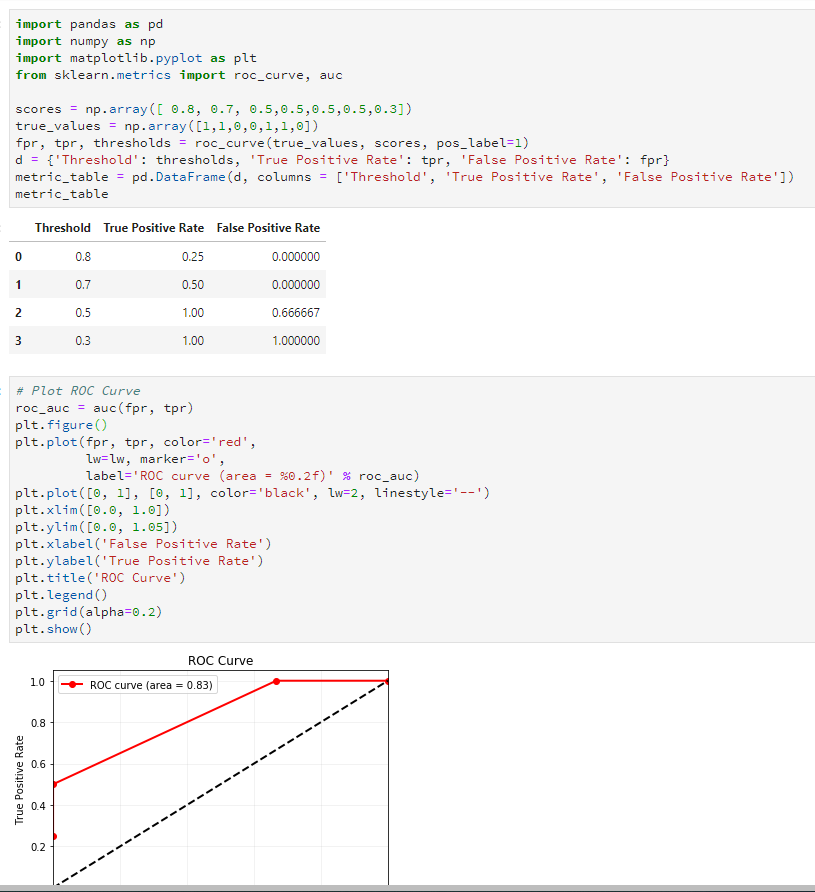
那么ROC曲线是怎么绘制的?假设有一个预测结果如下:
probability <- data.frame(Original_class= c("DLBCL", "DLBCL", "DLBCL", "DLBCL", "DLBCL", "FL", "DLBCL","FL", "FL"),Predicted_socre_for_sample_in_class_DLBCL=seq(0.9,0.1,-0.1))
probability## Original_class Predicted_socre_for_sample_in_class_DLBCL
## 1 DLBCL 0.9
## 2 DLBCL 0.8
## 3 DLBCL 0.7
## 4 DLBCL 0.6
## 5 DLBCL 0.5
## 6 FL 0.4
## 7 DLBCL 0.3
## 8 FL 0.2
## 9 FL 0.1如果设置Predicted_socre_for_sample_in_class_DLBCL:
>0.75为阈值标准,那么灵敏度=2/6=0.33,假阳性率为1-3/3=0;>0.65为阈值标准,那么灵敏度=3/6=0.50,假阳性率为1-3/3=0;>0.55为阈值标准,那么灵敏度=4/6=0.67,假阳性率为1-3/3=0;>0.45为阈值标准,那么灵敏度=5/6=0.83,假阳性率为1-3/3=0;>0.35为阈值标准,那么灵敏度=5/6=0.83,假阳性率为1-2/3=0.33;>0.25为阈值标准,那么灵敏度=6/6=1.00,假阳性率为1-2/3=0.33;>0.15为阈值标准,那么灵敏度=6/6=1.00,假阳性率为1-1/3=0.66;>0.05为阈值标准,那么灵敏度=6/6=1.00,假阳性率为1-0/3=1.00;
还是写个程序来算吧。
thresholdL = seq(1,0,-0.05)
right_class = "DLBCL"
score_column = "Predicted_socre_for_sample_in_class_DLBCL"
original_right = sum(probability$Original_class == right_class)
original_wrong = sum(probability$Original_class != right_class)tpr_fpr <- function(probability, score_column, threshold, right_class, original_right, original_wrong){pass_threshold = as.vector(probability[probability[[score_column]]>threshold,1])# print(pass_threshold)pass_threshold_true = sum(pass_threshold == right_class)pass_threshold_false = sum(pass_threshold != right_class)tpr <- pass_threshold_true/original_rightfpr <- pass_threshold_false/original_wrongreturn(c(threshold=threshold, tpr=tpr, fpr=fpr, right_class=right_class))
}ROC_data = as.data.frame(do.call(rbind, lapply(thresholdL, tpr_fpr, probability=probability, score_column = score_column, right_class=right_class, original_right=original_right, original_wrong=original_wrong)))
ROC_data$tpr <- as.numeric(ROC_data$tpr)
ROC_data$fpr <- as.numeric(ROC_data$fpr)ROC_data结果如下
## threshold tpr fpr right_class
## 1 1 0.0000000 0.0000000 DLBCL
## 2 0.95 0.0000000 0.0000000 DLBCL
## 3 0.9 0.0000000 0.0000000 DLBCL
## 4 0.85 0.1666667 0.0000000 DLBCL
## 5 0.8 0.1666667 0.0000000 DLBCL
## 6 0.75 0.3333333 0.0000000 DLBCL
## 7 0.7 0.3333333 0.0000000 DLBCL
## 8 0.65 0.5000000 0.0000000 DLBCL
## 9 0.6 0.5000000 0.0000000 DLBCL
## 10 0.55 0.6666667 0.0000000 DLBCL
## 11 0.5 0.6666667 0.0000000 DLBCL
## 12 0.45 0.8333333 0.0000000 DLBCL
## 13 0.4 0.8333333 0.3333333 DLBCL
## 14 0.35 0.8333333 0.3333333 DLBCL
## 15 0.3 0.8333333 0.3333333 DLBCL
## 16 0.25 1.0000000 0.3333333 DLBCL
## 17 0.2 1.0000000 0.3333333 DLBCL
## 18 0.15 1.0000000 0.6666667 DLBCL
## 19 0.1 1.0000000 1.0000000 DLBCL
## 20 0.0499999999999999 1.0000000 1.0000000 DLBCL
## 21 0 1.0000000 1.0000000 DLBCL简单地绘制下ROC曲线
library(ggplot2)ggplot(ROC_data, aes(x=fpr, y=tpr, group=right_class)) + geom_step(direction="vh", color='red') + geom_abline(intercept = 0, slope = 1) + theme_classic() + scale_y_continuous(expand=c(0,0)) + xlab("False positive rate") + ylab("True positive rate") + coord_fixed(1)如果right_class是FL呢?(这里只是用同一套数据,方便说明问题,实际需要对score取反)
thresholdL = seq(1,0,-0.05)
right_class = "FL"
score_column = "Predicted_socre_for_sample_in_class_DLBCL"
original_right = sum(probability$Original_class == right_class)
original_wrong = sum(probability$Original_class != right_class)tpr_fpr <- function(probability, score_column, threshold, right_class, original_right, original_wrong){pass_threshold = as.vector(probability[probability[[score_column]]>threshold,1])# print(pass_threshold)pass_threshold_true = sum(pass_threshold == right_class)pass_threshold_false = sum(pass_threshold != right_class)tpr <- pass_threshold_true/original_rightfpr <- pass_threshold_false/original_wrongreturn(c(threshold=threshold, tpr=tpr, fpr=fpr, right_class=right_class))
}ROC_data2 = as.data.frame(do.call(rbind, lapply(thresholdL, tpr_fpr, probability=probability, score_column = score_column, right_class=right_class, original_right=original_right, original_wrong=original_wrong)))
ROC_data2$tpr <- as.numeric(ROC_data2$tpr)
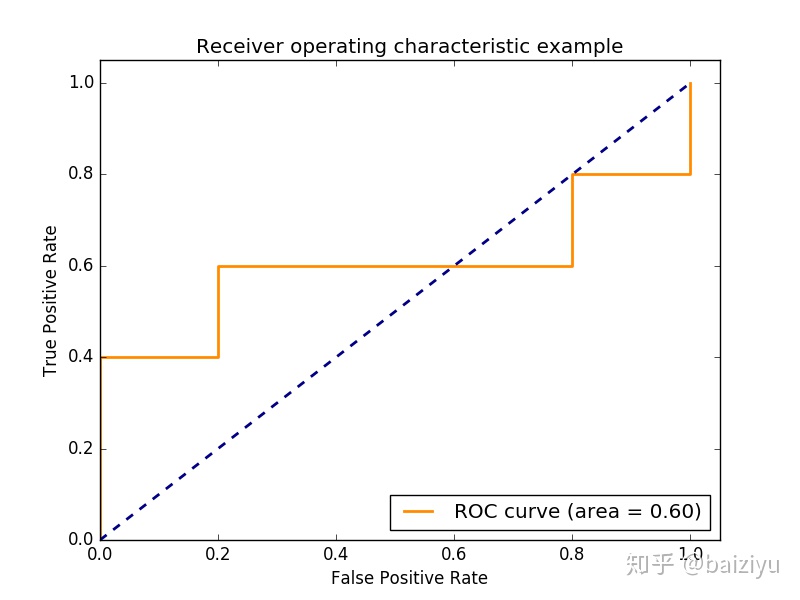
ROC_data2$fpr <- as.numeric(ROC_data2$fpr)ggplot(ROC_data2, aes(x=fpr, y=tpr, group=right_class)) + geom_step(direction="vh", color='red') + geom_abline(intercept = 0, slope = 1) + theme_classic() + scale_x_continuous(expand=c(0,0)) + xlab("False positive rate") + ylab("True positive rate") + coord_fixed(1)从这张图可以看到,不管是根据FL计算,还是DLBCL计算,ROC曲线是一致的。
ROC_data3 = rbind(ROC_data, ROC_data2)ggplot(ROC_data3, aes(x=fpr, y=tpr, color=right_class)) + geom_step(direction="vh") + geom_abline(intercept = 0, slope = 1) + theme_classic() + xlab("False positive rate") + ylab("True positive rate") + coord_fixed(1)同时ROC曲线不不因样品不均衡而受影响。
阈值的改变只是会改变真阳性率(灵敏度)和假阳性率。但是ROC曲线却不会变化。这只是我们控制假阳性率和灵敏度在合理范围时设置的过滤标准。
AUC (Area under curve)
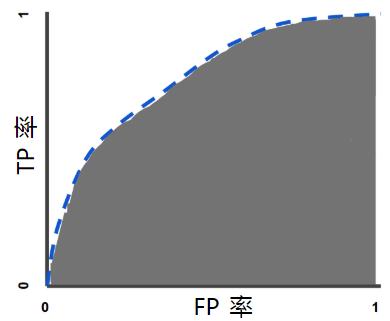
如何根据ROC曲线判断一个模型的好坏呢?ROC曲线越陡越好,说明在较低的假阳性率时可以获得较高的真阳性率。一般通过曲线下面积AUC评估一个ROC曲线的好坏。一般模型的AUC值在0.5-1之间,值越大越好。
下面是一个经验展示。AUC=1是最理想的情况。AUC=0.5就是随机模型。如果总是AUC<0.5模型就可以反过来用。
实际计算面积时并不是按几何图形进行计算的。通常根据AUC的物理意义进行计算。AUC值是一个概率值,当你随机挑选一个正样本以及负样本,当前的分类算法根据计算得到的Score值将这个正样本排在负样本前面的概率就是AUC值,AUC值越大,当前分类算法越有可能将正样本排在负样本前面,从而能够更好地分类。
假设有一套数据包含n个样本,M个正样本,N个负样本。每个样本计算一个Score值,所有样本按Score值排序。Score值最大的样本的rank为n(下面例子中是9),Score值次大的样品的rank为n-1(下面例子中是8)。正样品中rank最大的样本有rank_positive_max - 1 - (M-1)个负样本比他的score小 (M-1代表多少个正样本比);正样品中rank第二大的样本有rank_positive_second - 1 - (M-2)个负样本比他的score小;
如下面样品a是rank最大的正样品其rank值为9,有3个负样品得分比其低。3怎么算的呢?3 = 9 - 1 - (6-1) = rank1 - M(9是样品a的rank,-1是排除自身,6是正样本数,6-1是除了a之外的正样本数)
如下面样品b是rank次大的正样品其rank值为8,有3个负样品得分比其低。3怎么算的呢?3 = 8 - 1 - (6-2) = rank2 - M+1(8是样品b的rank,-1是排除自身,6是正样本数,6-2是除了a,b之外的正样本数)
单独提取出后面的值就是M, M+1, M+2, M+3, ..., M + M-1,其加和就是
这样就可以总结出一个公式:
probability_rank <- data.frame(Sample = letters[1:9],Original_class= c("DLBCL", "DLBCL", "DLBCL", "DLBCL", "DLBCL", "FL", "DLBCL","FL", "FL"),Predicted_socre_for_sample_in_class_DLBCL=seq(0.9,0.1,-0.1),rank=seq(9,1,-1))
probability_rank## Sample Original_class Predicted_socre_for_sample_in_class_DLBCL rank
## 1 a DLBCL 0.9 9
## 2 b DLBCL 0.8 8
## 3 c DLBCL 0.7 7
## 4 d DLBCL 0.6 6
## 5 e DLBCL 0.5 5
## 6 f FL 0.4 4
## 7 g DLBCL 0.3 3
## 8 h FL 0.2 2
## 9 i FL 0.1 1写个函数计算
# 测试用例
# probability <- data.frame(Original_class= c("DLBCL", "DLBCL", "DLBCL", "DLBCL", "DLBCL", "FL", "DLBCL","FL", "FL"),
# Predicted_socre_for_sample_in_class_DLBCL=sample(seq(0,1,0.1),9))
# probability
AUC <- function(probability, score_column, class_column, right_class){sample_count = nrow(probability)positive_count = nrow(probability[probability[[class_column]]==right_class,])negative_count = sample_count - positive_countprobability <- probability[rev(order(probability[[score_column]])),]probability$rank <- sample_count:1print(probability)rank_sum = sum(probability[probability[[class_column]]==right_class, "rank"])return((rank_sum - (positive_count+positive_count^2)/2)/(positive_count * negative_count))
}AUC(probability, "Predicted_socre_for_sample_in_class_DLBCL", "Original_class", "DLBCL")## Original_class Predicted_socre_for_sample_in_class_DLBCL rank
## 1 DLBCL 0.9 9
## 2 DLBCL 0.8 8
## 3 DLBCL 0.7 7
## 4 DLBCL 0.6 6
## 5 DLBCL 0.5 5
## 6 FL 0.4 4
## 7 DLBCL 0.3 3
## 8 FL 0.2 2
## 9 FL 0.1 1## [1] 0.9444444参考
https://www.spectrumnews.org/opinion/viewpoint/quest-autism-biomarkers-faces-steep-statistical-challenges/
https://www.cnblogs.com/gatherstars/p/6084696.html
https://blog.csdn.net/qq_24753293/article/details/80942650-
往期精品(点击图片直达文字对应教程)
后台回复“生信宝典福利第一波”或点击阅读原文获取教程合集
(请备注姓名-学校/企业-职务等)
![[sklearn]性能度量之AUC值](https://img-blog.csdnimg.cn/20190510134654758.png?x-oss-process=image/watermark,type_ZmFuZ3poZW5naGVpdGk,shadow_10,text_aHR0cHM6Ly9ibG9nLmNzZG4ubmV0L0t5cmllX0lydmluZw==,size_16,color_FFFFFF,t_70)