文章目录
- 前言
- 一、发现问题
- 二、分析问题
- 三、解决问题
- 四、读写速度
- 总结
- 吐槽
前言
当你使出了浑身解数,read.csv和read.table还是无法读入数据时,或许可以尝试一下readr包中的read_table,read_csv等函数。尝试一下!
我是一个很专一(懒)的人,尽管网上有人说readr包中读入数据较传统函数速度快,我都一直用的传统方法,没有尝试过readr包。
直到今天实验室师兄告诉我他r语言读不进去数据,我尝试了很久,使用了很多参数,也网上搜索了很久,但都无果(可能是我比较笨),我才尝试使用这个包,结果就是,没有传入过多的参数就读进来了,太牛了,我哭死。
所以记录一下整个过程以及比较它和传统函数读写数据速度的差别。
一、发现问题
数据我就不上传了,说不定你们也不会遇到这种问题。。主要看一下报错吧!
我一开始觉得可能是数据编码的问题,然后:
a <- read.csv(file = 'seerv.csv',header = T,fill = T,check.names = F,fileEncoding = 'GBK')
a <- read.csv(file = 'seerv.csv',header = T,fill = T,check.names = F,fileEncoding = 'UTF-8')
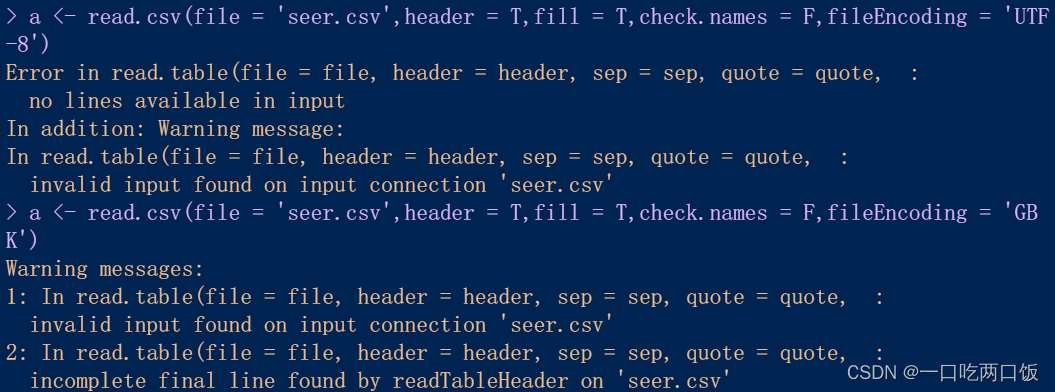
关于里面的header,fill,check.names,encoding,fileEncoding等参数我都进行了尝试(很多情况下,这些参数的更改可以顺利的读入数据),无果。(想进一步尝试并且交流的,我可以私发部分数据)
二、分析问题
除了更改部分参数,我还想着欣赏一下read.table的源代码,结果发现里面部分可能是引用的C语言,然而当初仅仅学习了C语言基础,所以企图通过源码解决问题的路子又断了。
通过read.table,不要加括号。或者edit(read.table) 可以查看代码。
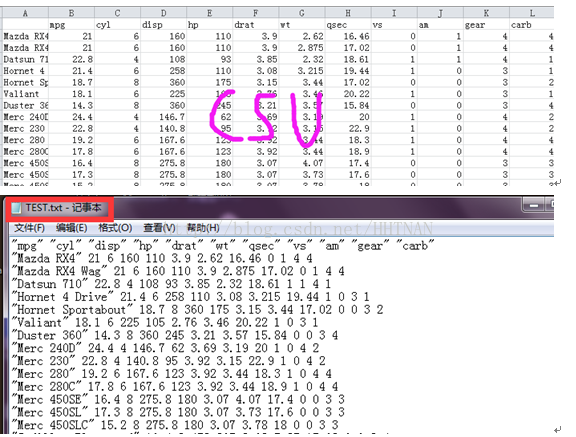
这是部分源码的图:
报错信息“no lines available in input”赫然出现,我知道在哪儿可能有问题,可是我却没有办法进一步分析原因。
三、解决问题
最后我脑抽在网上搜了一下,R语言读入数据,发现可以通过readr包读写数据,然后就成功读入了。
然后我,我懒,真的不想搞懂为什么传统方法读那个数据的时候会报错,就此作罢。
library(readr)
a <- read_csv(file = 'seer.csv')

我*,以前的时候不使用check.names=F参数的话,传统方法会将某些特殊符号编码为.(点),而read_csv根本不需要用什么check.names参数。
四、读写速度
最后,很多人都说这个包读写数据快,我就测试一下它和传统函数在读写速度上的区别,这个区别到底有多大。
d <- matrix(rnorm(25000000),nrow = 5000,ncol = 5000)
d <- as.data.frame(d)
t1 <- Sys.time()
write.csv(d,file = 'write.csv.csv')
Sys.time()-t1
t1 <- Sys.time()
write_csv(d,file = 'write_csv.csv')
Sys.time()-t1t1 <- Sys.time()
a <- read.csv(file = 'write.csv.csv')
Sys.time()-t1t1 <- Sys.time()
b <- read_csv(file = 'write_csv.csv')
Sys.time()-t1
写入数据:
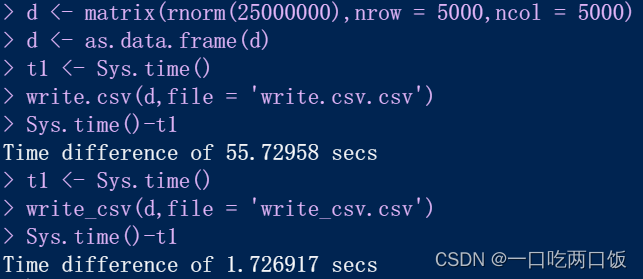
55.7秒 vs 1.7秒
write_csv: 的速度是write.csv: 的近30 倍。
读入数据:
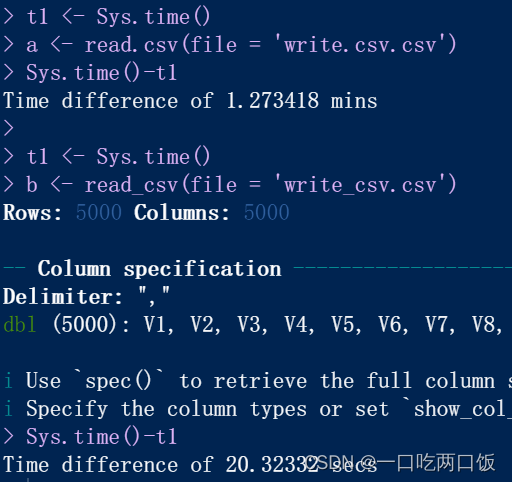
1.3分 vs 20.3秒
read_csv: 读取数据的速度是read.csv的近3倍。
仅用一个数据集和一对函数进行比较,虽然结果速度差距可能不准确,但readr包读写速度真的快,我总不能每个数据都测试吧。
这里只是单纯进行一对函数读写速度的比较
函数里面的参数就不多提了。
总结
我太不想搞懂为什么数据读不进去的原因了,只要能读出来就行,不然师兄折磨我。哈哈哈!
吐槽
说个今天遇到的无语事,某某对于购买的代码,代码跑不下去了,报错了,发给我代码和数据,我跑了他的代码发现,基因矩阵里面没有预设的那个基因名字,换一个基因去研究就行了(可能说明书不够详尽)。然后就没有然后了,一句谢谢都没有。
小白一枚,欢迎大家批评指正。
如果大家能解决前面的read.csv问题的,私聊我,我给你部分数据。