一、环境要求
目前只支持在Linux和MacOS系统,虽然可以用 -parallel 进行多线程处理,但基本消耗的是CPU内存。
二、FreeSurfer的下载与安装
1. 下载安装包
官网链接:rel7downloads - Free Surfer Wiki
根据自己系统型号下载对应安装包,我的电脑是Ubantu18.2的,下载的是freesurfer-linux-centos6_x86_64-7.2.0.tar.gz,记得科学上网,不然下载要二十多个小时。

或者直接在官网复制下载链接后,在自己想要安装的路径下打开终端,使用如下命令下载安装包
wget https://surfer.nmr.mgh.harvard.edu/pub/dist/freesurfer/7.2.0/freesurfer-linux-centos6_x86_64-7.2.0.tar.gz2. FreeSurfer的安装
FreeSurfer没有繁琐的安装过程,对下载的压缩包进行解压即可,解压后目录下会生成名为freesurfer的文件夹,记住这个路径,之后配置环境需要用到
解压命令:
tar -zxv -f freesurfer-linux-centos6_x86_64-7.2.0.tar.gz
3. 注册
软件需要官方授权,只需要在网站进行注册,官方会在发送一封邮件,把附件的license.txt放在刚才生成的freesurfer文件夹下即可
注册网站:FreeSurfer Registration form
4. 安装tcsh
在终端输入:
sudo apt-get install tcsh5. 配置环境
根据解压的路径设置文件夹权限,命令:
sudo chmod -R 777 ./freesurfer添加环境变量,这里需要修改为自己电脑freesurfer解压的路径:
使用 vi ~/.bashrc 打开系统环境文件,在~/.bashrc文件中添加如下两句:
export FREESURFER_HOME=/home/LYL/freesurfer #freesurfer解压路径
source $FREESURFER_HOME/SetUpFreeSurfer.sh
export SUBJECTS_DIR=/home/LYL/data/Brain/MRI #处理结果保存路径
添加结束后后,按Esc,输入:wq 即可退出并保存,在终端输入
source ~/.bashrc #环境修改立即生效输入recon-all --help,未报错则安装成功。
三、FreeSurfer的使用
官方教程:recon-all - Free Surfer Wiki,ReconAllDevTable - Free Surfer Wiki
注意:数据需要全部放在同一文件夹下,且并列排布,不要文件夹下再包含文件。
1. 启动
一般选择在MRI数据所在目录或freesurfer安装目录打开终端,每次重新打开时都要输入命令:
export FREESURFER_HOME=/home/LYL/freesurfer #freesurfer解压路径
source $FREESURFER_HOME/SetUpFreeSurfer.sh
2. 基础语句介绍
freesurfer一共有31个处理步骤,使用recon-all可完成全部处理步骤,如只需部分可应用其他语句,这里稍作介绍。
recon-all -i 1.nii -s AD1 -sd /home/LYL/data/AD/MRI/ -all
# 1.nii为需处理的nii图
# AD为处理后的存放的文件夹名字(处理结束后自动生成)
# /home/LYL/data/AD/MRI/为处理结果存放位置2.1 recon-all全步骤
01. 运动校正与确认
02. NU (非均匀强度标准化处理)
03. Talairach 变换计算
04. 强度标准化1
05. 去除脑壳
06. EM登记 (线性体积登记)
07. CA 强度标准化
08. CA 非线性体积登记
09. 去除颈部
10. 脑壳LTA
11. CA标记 (体积标记, 如Aseg)和统计
12. 强度标准化2 (控制点开始)
13. 白质分割
14. 白质ASeg编辑
15. 填充(开始白质编辑)
16. Tessellation细分曲面技术 (开始每个脑半球的操作)
17. 平滑1
18. 胀平1
19. QSphere
20. 自动化局部解剖修复
21. 最终的曲面 (从这里开始编辑软脑膜)
22. 平滑2
23. 胀平2
24. 球形映射
25. 球形登记
26. 球形登记,对侧半球
27. Subject平均曲率映射
28. 皮质划分 - Desikan_Killian和Christophe 分类
29. 皮质划分统计
30. 皮质色带覆盖
31. 皮质划分到aseg映射
每一步的输入输出和参数可参考:FreeSurfer核心命令之recon-all - FreeSurfer使用手册 - 秋月斋人 | 豆瓣阅读
2.2 分步骤处理
| 语句 | 对应操作 |
| -autorecon1 | 完成步骤 1-5 |
| -autorecon2 | 完成步骤 6-23 |
| 完成 autorecon2后, 检查最终曲面: | |
| a. 如果需要编辑白质,执行 -autorecon2-wm,完成步骤15-23 | |
| b. 如果需要增加控制点,执行 -autorecon2-cp,完成步骤12-23 | |
| c. 如果需要修正软脑膜,执行 -autorecon2-pial,完成步骤21-23 | |
| d. 继续执行 -autorecon3,完成步骤24-31 | |
| -hemi ?h | 用参数声明仅处理lh还是rh(默认均处理) |
3. 颅骨剥离+仿射对齐
如果是需要做颅骨剥离和对齐,可以用下面这段代码批量处理,将如下代码存为preprocessing.py文件,在终端输入 python preprocessing.py ,回车即可
#!/media/milab-2080ti/LabData/LYL/freesurfer/python/bin/python3.6
import os
import globpath = r"/media/milab-2080ti/LabData/LYL/1-DATA-ORIGINAL/EMCI/MRI" #数据读取目录
new_path = r"/media/milab-2080ti/LabData/LYL/2-DATA-pre/EMCI/MRI" #结果保存目录images = glob.glob(os.path.join(path,"*.nii")) #读取path下的nii文件
# 下面为freesurfer的环境配置命令
a = "export FREESURFER_HOME=/media/milab-2080ti/LabData/LYL/freesurfer;"
b = "source $FREESURFER_HOME/SetUpFreeSurfer.sh;"
c = "export SUBJECTS_DIR="+new_path+";"for image in images:
# 将文件路径和文件名分离filename = os.path.split(image)[1] # 将路径名和文件名分开filename = os.path.splitext(filename)[0] #将文件名和扩展名分开,如果为.nii.gz,则认为扩展名是.gzfilename = filename[:15] #根据扩展名的不同,只保留文件名即可,这里需要做更改#cur_path=os.path.join(path,filename) #存原始目录#cur_path = os.path.join(path, filename)cur_path = os.path.join(new_path, filename)# print("file name: ", cur_path)# freesurfer环境配置、颅骨去除、未仿射对齐mpz转nii、仿射对齐、仿射对齐mpz转nii.gz格式# recon-all是数据开始处理的命令, -parallels指并行处理,-autorecon1 -subjid指只使用recon-all的前五步:运动校正、非均匀强度归一化、Talairach变换、强度归一化,去颅骨,具体见recon-all介绍# mri_convert是进行格式转换,从mgz转到nii.gz,只是为了方便查看# --apply_transform:仿射对齐操作cmd = a + b + c \+ "recon-all -parallel -i " + image + " -autorecon1 -subjid " + cur_path + "&&" \+ "mri_convert " + cur_path + "/mri/brainmask.mgz " \+ cur_path + "/mri/" + filename + "_brainmask.nii.gz;"\+ "mri_convert " + cur_path + "/mri/brainmask.mgz --apply_transform " \+ cur_path + "/mri/transforms/talairach.xfm -o " \+ cur_path + "/mri/brainmask_affine.mgz&&" \+ "mri_convert " + cur_path + "/mri/brainmask_affine.mgz " \+ cur_path + "/mri/"+filename+"_affine.nii.gz;"# print("cmd:\n",cmd)os.system(cmd)完成一个后,会在SUBJECTS_DIR目录下生成一个以原图名称命名的文件夹
文件夹的子目录mri中存放的是完成预处理的结果
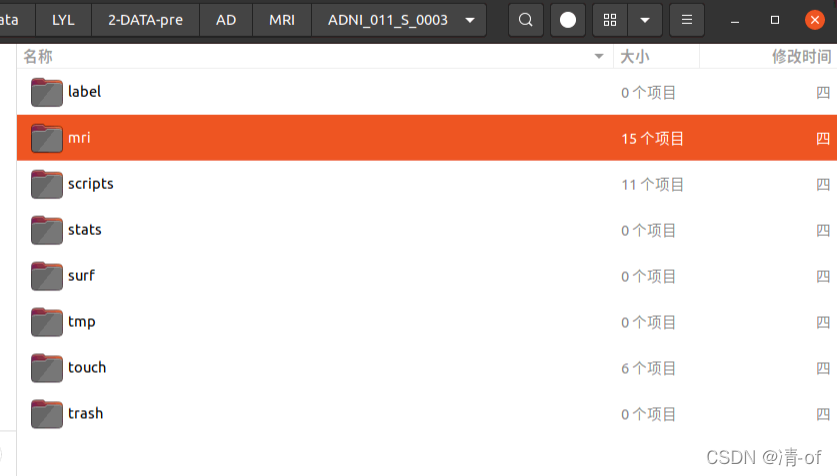
剥离颅骨结果:brainmask.mgz
仿射校正结果:brainmask_affine.mgz
剥离颅骨转成nii结果:ADNI_011_S_0003_brainmask.nii.gz
仿射校正转成nii结果:ADNI_011_S_0003_brainmask_affine.nii.gz

官方安装教程:FS7_linux - Free Surfer Wiki