在线通路注释,一般使用DAVID、KASS、KOBAS等工具。
Kobas : KOBAS(基于KEGG Orthology Based Annotation System)是用于基因/蛋白质功能注释(注释模块)和功能集富集(Enrichment module)的Web服务器。给定一组基因或蛋白质,它可以确定通路,疾病和基因本体论(GO)术语是否显示统计学显着性。
KOBAS 3.0由两个功能组成,注释(Annotation)和富集(Enrichment)
KOBAS 3.0的输入不支持gene symbol,一般操作将Symbol ID转换成Entrez Gene ID(或者)ensembl格式的ID。
推荐进行基因ID转换的网站:gprofiler : http://biit.cs.ut.ee/gprofiler/gconvert.cgi
注释(Annotation):
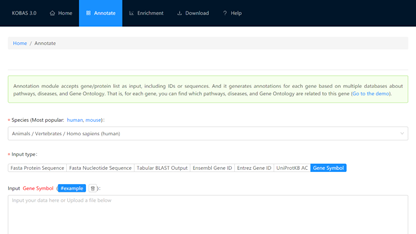
对于Annotation模块,它接受基因/蛋白质列表作为输入,包括ID或序列。 对于每个基因,您可以找到与该基因相关的途径,疾病和基因本体(GO).
富集(Enrichment)
富集模块为您提供答案,了解哪些途径,疾病和GO术语与您刚输入的基因/蛋白质具有统计学意义。
该模块在KOBAS 2.0中称为“识别”。它接受与Annotation模块相同的输入格式,并且还允许Annotation模块的结果作为输入(请参阅3.1中的详细信息)。它基于第一代基因集富集方法,一种称为过表达分析(ORA)的基因水平统计,这是一种基于超几何分布的简单且经常使用的测试。
注意事项:
输入数据:
1、Gene/protein ID、Sequences(FASTA format)、Tabular BLAST output
2、Gene expression matrix
输出结果:
- Term KEGG的注释类
- Database 数据库类型
- ID Term对应的ID
- Input 输入的基因数目
- input number 富集到这个Term的输入基因个数
- Background number 数据库中富集到这个通路的总有基因数量
- P-value P值
- Corrected P-Value 校正后P值
KOBAS 本地操作:
下载 应用: http://kobas.cbi.pku.edu.cn/kobas-2.1.1/kobas-3.0.3.tar.gz
下载 数据库: http://kobas.cbi.pku.edu.cn/download_file.php?type=seq_pep&filename=ko.pep.fasta.gz
### 1、KO数据库建索引
$ diamond makedb --in ko.pep.fasta --db ko -p 24### 2、diamond blast
$ diamond blastx -e 1e-5 --db ko -q genes.nucleotide.catalog.fa -p 24 -f 6 qseqid qlen qstart qend qcovhsp slen sstart send score evalue positive length ppos sseqid stitle nident mismatch gaps gapopen bitscore pident -o kobas.annotation### 转成Tabular BLAST output format
$ awk -F "\t" '{print $1"\t"$14"\t"$21"\t"$12"\t"$17"\t"$19"\t"$3"\t"$4"\t"$7"\t"$8"\t"$10"\t"$20}' kobas.annotation > kobas.annotation.m8### 注释
$ annotate.py -i kobas.annotation.m8 -t blastout:tab -s ko -o kegg.annotate.tmp### 检测
$ identify.py -f kegg.annotate.tmp -o kegg.identify.tmp -p K -m h -b tmp### 富集
$ cluster.py -i kegg.identify.tmp -m k -o kegg.cluster.txt ### 查看注释结果
$less kegg.annotate.tmp
##ko KEGG Orthology
##Method: BLAST Options: evalue <= 1e-05; rank <= 5
##Summary: 3431 succeed, 2654 fail#Query KO ID|KO name|Hyperlink
### 序列 K号|酶号 基因|ko pathway路径
gene_3301|GeneMark.hmm|1482_nt|+|3899966|3901447 K00045|E1.1.1.67, mtlK|http://www.genome.jp/dbget-bin/www_bget?ko:K00045
gene_3075|GeneMark.hmm|774_nt|+|3605726|3606499 K01692|paaF, echA|http://www.genome.jp/dbget-bin/www_bget?ko:K01692
gene_1278|GeneMark.hmm|1332_nt|+|1517095|1518426 None
......Query: gene_3301|GeneMark.hmm|1482_nt|+|3899966|3901447
KO: K00045 E1.1.1.67, mtlK
Pathway: Fructose and mannose metabolism KEGG PATHWAY ko00051Query: gene_3075|GeneMark.hmm|774_nt|+|3605726|3606499
KO: K01692 paaF, echA
Pathway: Fatty acid degradation KEGG PATHWAY ko00071Tryptophan metabolism KEGG PATHWAY ko00380
....功能富集分析概述 : https://www.jianshu.com/p/5a4bda169247















](https://img-blog.csdnimg.cn/b7d5f168deff4064a604be2dfbb5d4fb.gif#pic_center)