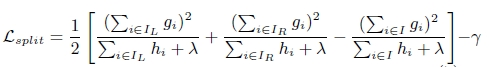首先刚接手这种关于信号的分类问题,以下可能会有不对的地方,接下去通过学习会对不正确的地方进行更正或者补充。
心电信号分类参考文献:Cardiologist-LevelArrhythmiaDetectionwithConvolutionalNeuralNetworks
目标:对传感器采集的某一维信号数据(time-amplitude)进行分类,比如通过一段一维心音信号进行正常/异常的分类。
方法:这里采用端到端通过卷积神经网络的方式对一段时域的一维心音信号得到正常/异常的类别诊断(本质是分类模型的搭建),卷积神经网络使用参考文献中的,结构如下所示:
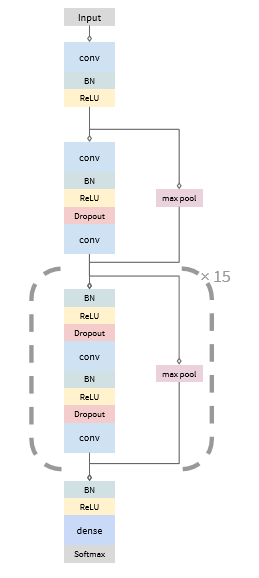
网络结构参数:
输入shape:(batch_size,steps,1),steps必须是256的倍数,对应的label shape:(batch_size,steps/256,class_number),由于网络下采样256倍,这样最后feature map的输出shape(无Flatten层,所以输出有3个维度):(batch_size,steps/下采样倍数256,softmax_class_number)与label shape才能匹配上。从这里可以看出,对应输入的steps个采样点,每256个采样点有一个label,那么steps个采样点就有steps/256个label
网络从input->output共下采样256倍(每两个resnet block进行2倍下采样)
共16个resnet block,每四个resnet block增加2倍滤波器的数量,分别为32,64,128,256
卷积核:1*1 kernel size
Maxpooling中设置padding=‘same’
具体方法:
1、心音数据预处理:
原因:不同数据集的音频数据制作标准会有差异,这种差异包括:音频采样率,通道数,长短,降噪法,尺度等,所以在对所有数据集进行融合之前需要最大可能地统一这些标准。

2、特征提取:
心音信号有时域特征和频域特征,由于这里借鉴的是心电信号的分类模型,所以这里只采用一维的时域特征进行分类模型的搭建,即直接拿一维心音信号的采样点强度作为输入(输入的steps必须是256的倍数,且每256个采样点有一个label)。
代码实现:
(1)分类模型的搭建:
from keras import backend as Kdef _bn_relu(layer, dropout=0, **params):from keras.layers import BatchNormalizationfrom keras.layers import Activationlayer = BatchNormalization()(layer)layer = Activation(params["conv_activation"])(layer)if dropout > 0:from keras.layers import Dropoutlayer = Dropout(params["conv_dropout"])(layer)return layerdef add_conv_weight(layer,filter_length,num_filters,subsample_length=1,**params):from keras.layers import Conv1D layer = Conv1D(filters=num_filters,kernel_size=filter_length,strides=subsample_length,padding='valid',kernel_initializer=params["conv_init"])(layer)return layerdef add_conv_layers(layer, **params):for subsample_length in params["conv_subsample_lengths"]:layer = add_conv_weight(layer,params["conv_filter_length"],params["conv_num_filters_start"],subsample_length=subsample_length,**params)layer = _bn_relu(layer, **params)return layerdef resnet_block(layer,num_filters,subsample_length,block_index,**params):from keras.layers import Add from keras.layers import MaxPooling1Dfrom keras.layers.core import Lambdadef zeropad(x):y = K.zeros_like(x)return K.concatenate([x, y], axis=2)def zeropad_output_shape(input_shape):shape = list(input_shape)assert len(shape) == 3shape[2] *= 2return tuple(shape)shortcut = MaxPooling1D(pool_size=subsample_length, padding='same')(layer)zero_pad = (block_index % params["conv_increase_channels_at"]) == 0 \and block_index > 0if zero_pad is True:shortcut = Lambda(zeropad, output_shape=zeropad_output_shape)(shortcut)for i in range(params["conv_num_skip"]):if not (block_index == 0 and i == 0):layer = _bn_relu(layer,dropout=params["conv_dropout"] if i > 0 else 0,**params)layer = add_conv_weight(layer,params["conv_filter_length"],num_filters,subsample_length if i == 0 else 1,**params)layer = Add()([shortcut, layer])return layerdef get_num_filters_at_index(index, num_start_filters, **params):return 2**int(index / params["conv_increase_channels_at"]) \* num_start_filtersdef add_resnet_layers(layer, **params):layer = add_conv_weight(layer,params["conv_filter_length"],params["conv_num_filters_start"],subsample_length=1,**params)layer = _bn_relu(layer, **params)for index, subsample_length in enumerate(params["conv_subsample_lengths"]):num_filters = get_num_filters_at_index(index, params["conv_num_filters_start"], **params)layer = resnet_block(layer,num_filters,subsample_length,index,**params)layer = _bn_relu(layer, **params)return layerdef add_output_layer(layer, **params):from keras.layers.core import Dense, Activationfrom keras.layers.wrappers import TimeDistributedlayer = TimeDistributed(Dense(params["num_categories"]))(layer)return Activation('softmax')(layer)def add_compile(model, **params):from keras.optimizers import Adamoptimizer = Adam(lr=params["learning_rate"],clipnorm=params.get("clipnorm", 1))model.compile(loss='categorical_crossentropy',optimizer=optimizer,metrics=['accuracy'])def build_network(**params):from keras.models import Modelfrom keras.layers import Inputinputs = Input(shape=params['input_shape'],dtype='float32',name='inputs')if params.get('is_regular_conv', False):layer = add_conv_layers(inputs, **params)else:layer = add_resnet_layers(inputs, **params)output = add_output_layer(layer, **params)model = Model(inputs=[inputs], outputs=[output])if params.get("compile", True):add_compile(model, **params)return model
(2)训练脚本
from __future__ import print_function
from __future__ import division
from __future__ import absolute_importimport argparse
import json
import keras
import numpy as np
import os
import random
import timeimport network
import load
import utilMAX_EPOCHS = 100def make_save_dir(dirname, experiment_name):start_time = str(int(time.time())) + '-' + str(random.randrange(1000))save_dir = os.path.join(dirname, experiment_name, start_time)if not os.path.exists(save_dir):os.makedirs(save_dir)return save_dirdef get_filename_for_saving(save_dir):return os.path.join(save_dir,"{val_loss:.3f}-{val_acc:.3f}-{epoch:03d}-{loss:.3f}-{acc:.3f}.hdf5")def train(args, params):print("Loading training set...")train = load.load_dataset(params['train'])print("Loading dev set...")dev = load.load_dataset(params['dev'])print("Building preprocessor...")preproc = load.Preproc(*train)print("Training size: " + str(len(train[0])) + " examples.")print("Dev size: " + str(len(dev[0])) + " examples.")save_dir = make_save_dir(params['save_dir'], args.experiment)util.save(preproc, save_dir)params.update({"input_shape": [None, 1],"num_categories": len(preproc.classes)})model = network.build_network(**params)stopping = keras.callbacks.EarlyStopping(patience=8)reduce_lr = keras.callbacks.ReduceLROnPlateau(factor=0.1,patience=2,min_lr=params["learning_rate"] * 0.001)checkpointer = keras.callbacks.ModelCheckpoint(filepath=get_filename_for_saving(save_dir),save_best_only=False)batch_size = params.get("batch_size", 32)if params.get("generator", False):train_gen = load.data_generator(batch_size, preproc, *train)dev_gen = load.data_generator(batch_size, preproc, *dev)model.fit_generator(train_gen,steps_per_epoch=int(len(train[0]) / batch_size),epochs=MAX_EPOCHS,validation_data=dev_gen,validation_steps=int(len(dev[0]) / batch_size),callbacks=[checkpointer, reduce_lr, stopping])else:train_x, train_y = preproc.process(*train)dev_x, dev_y = preproc.process(*dev)model.fit(train_x, train_y,batch_size=batch_size,epochs=MAX_EPOCHS,validation_data=(dev_x, dev_y),callbacks=[checkpointer, reduce_lr, stopping])if __name__ == '__main__':parser = argparse.ArgumentParser()parser.add_argument("config_file", help="path to config file")#config_file为神经网络的参数,保存成.json文件parser.add_argument("--experiment", "-e", help="tag with experiment name",default="default")args = parser.parse_args()params = json.load(open(args.config_file, 'r'))train(args, params)
(3)batch_data,batch_label的生成
from __future__ import print_function
from __future__ import division
from __future__ import absolute_importimport json
import keras
import numpy as np
import os
import random
import scipy.io as sio
import tqdmSTEP = 256def data_generator(batch_size, preproc, x, y):num_examples = len(x)examples = zip(x, y)examples = sorted(examples, key = lambda x: x[0].shape[0])end = num_examples - batch_size + 1batches = [examples[i:i+batch_size]for i in range(0, end, batch_size)]random.shuffle(batches)while True:for batch in batches:x, y = zip(*batch)yield preproc.process(x, y)class Preproc:def __init__(self, ecg, labels):self.mean, self.std = compute_mean_std(ecg)self.classes = sorted(set(l for label in labels for l in label))self.int_to_class = dict( zip(range(len(self.classes)), self.classes))self.class_to_int = {c : i for i, c in self.int_to_class.items()}def process(self, x, y):return self.process_x(x), self.process_y(y)def process_x(self, x):x = pad(x)x = (x - self.mean) / self.stdx = x[:, :, None]return xdef process_y(self, y):# TODO, awni, fix hack pad with noise for cincy = pad([[self.class_to_int[c] for c in s] for s in y], val=3, dtype=np.int32) y = keras.utils.np_utils.to_categorical(y, num_classes=len(self.classes))return ydef pad(x, val=0, dtype=np.float32):max_len = max(len(i) for i in x)padded = np.full((len(x), max_len), val, dtype=dtype)for e, i in enumerate(x):padded[e, :len(i)] = ireturn paddeddef compute_mean_std(x):x = np.hstack(x)return (np.mean(x).astype(np.float32),np.std(x).astype(np.float32))def load_dataset(data_json):with open(data_json, 'r') as fid:data = [json.loads(l) for l in fid]labels = []; ecgs = []for d in tqdm.tqdm(data):labels.append(d['labels'])ecgs.append(load_ecg(d['ecg']))return ecgs, labelsdef load_ecg(record):if os.path.splitext(record)[1] == ".npy":ecg = np.load(record)elif os.path.splitext(record)[1] == ".mat":ecg = sio.loadmat(record)['val'].squeeze()else: # Assumes binary 16 bit integerswith open(record, 'r') as fid:ecg = np.fromfile(fid, dtype=np.int16)trunc_samp = STEP * int(len(ecg) / STEP)return ecg[:trunc_samp]if __name__ == "__main__":data_json = "examples/cinc17/train.json"train = load_dataset(data_json)preproc = Preproc(*train)gen = data_generator(32, preproc, *train)for x, y in gen:print(x.shape, y.shape)break
(4)预测的时候用到(归一化),保存训练数据集中一维心音信号的均值,标准差,类别等信息
import os import cPickle as pickledef load(dirname):preproc_f = os.path.join(dirname, "preproc.bin")with open(preproc_f, 'r') as fid:preproc = pickle.load(fid)return preprocdef save(preproc, dirname):preproc_f = os.path.join(dirname, "preproc.bin")with open(preproc_f, 'w') as fid:pickle.dump(preproc, fid)
(5)模型预测(batch_data的预测)
from __future__ import print_functionimport argparse
import numpy as np
import keras
import osimport load
import utildef predict(data_json, model_path):preproc = util.load(os.path.dirname(model_path))dataset = load.load_dataset(data_json)x, y = preproc.process(*dataset)model = keras.models.load_model(model_path)probs = model.predict(x, verbose=1)return probsif __name__ == '__main__':parser = argparse.ArgumentParser()parser.add_argument("data_json", help="path to data json")parser.add_argument("model_path", help="path to model")args = parser.parse_args()probs = predict(args.data_json, args.model_path)
小结:
心音信号的分类不能像心电信号一样处理,心电信号采样频率一般是200Hz,输入拿256个采样点,那么也有1.28s,且每1.28s有一个label,这对于通过1.28s心电信号的特征进行疾病诊断是合理的,而网络输入更加是同一个人连续30s的心电信号,那么就有200Hz*30=6000个采样点,由于256个采样点一个label,网络下采样倍数为256倍,所以输入为int(6000/256)*256个采样点。而心音信号的采样频率一般是4000Hz,数据预处理的时候一般下采样到1000Hz或2000Hz,如果也像心电一样拿256个采样点,那么时间只有0.256s或0.128s,而通过这不到1s的时间就要进行疾病诊断是不合理的,所以如果是做心音信号分类的话就不能采取如上的网络结构,可以修改的方法有如下:
(1)改变以上网络的输出结构,采用Flatten层,输出class_number个节点的softmax层,由于心音信号输入网络一般是3s左右, 是原始音频经过slice(切割)处理的,那么根据固定采样频率2000Hz来算的话,输入网络的采样点个数就为2000Hz*3s=6000个采样点
(2)采用频域特征,将一维时域信号转变成二维的时频信号再输入神经网络
(3)采用循环神经网络(这个具体没尝试过,接下去研究一下)