本文同步发布于个人博客https://www.horosama.com
相关文章链接:
单细胞测序系列之二:单细胞基因组测序
单细胞测序系列之三:单细胞转录组测序
文章目录
- 1 引言
- 2 第一代测序技术
- 3 第二代测序技术
- 4 第三代测序技术
1 引言
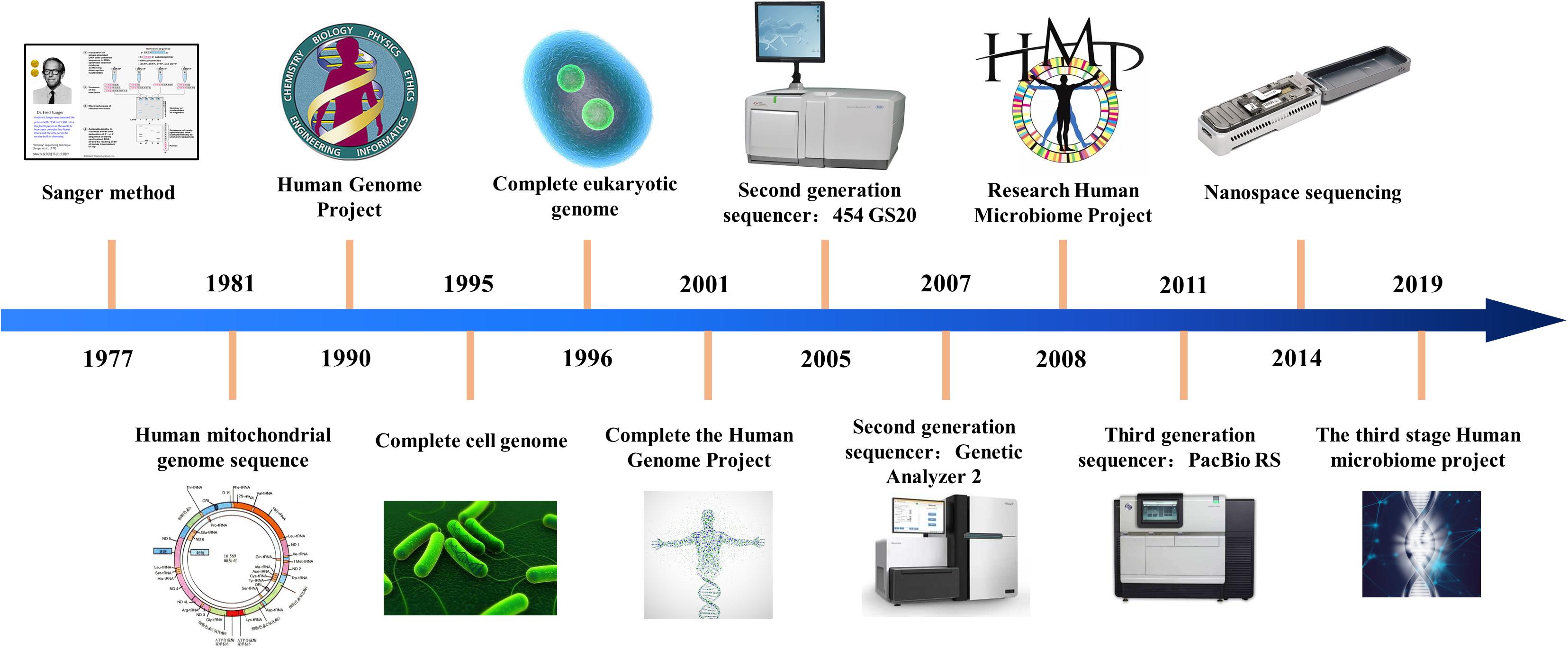
生命的秘密藏在DNA的A、C、G、T四种碱基的排列组合之中,而解密生命的首要任务,就是要确定DNA的序列。
核酸测序技术是20世纪生物学研究中最重要的发明之一,极大的推动了生物科学的发展1。基于核酸测序技术之上的人类基因组计划(Human Genome Project,HGP),和曼哈顿原子弹计划和阿波罗登月计划一起,被称为人类科技史上彻底改变世界的三大工程2。人类基因组测序计划对生命科学和医学等相关的研究有着直接的促进作用,也为人类的健康和生存提供了重要的基础。同时,在HGP的基础上,后续的各种基因组学、转录组学、表观基因组学、生物信息学等各个领域的研究也得到了极大的发展。
2 第一代测序技术
1968年,吴瑞先生提出了第一个DNA组成的测定方法3,1970年,吴瑞先生将引物延伸(primer extension)用于DNA测序,开了DNA测序的先河,并成为了后续的Sanger测序法的重要基础4。可以说,吴瑞先生是当之无愧的DNA测序之父。饶毅老师的博文《君子爱“生” 得之有道》5中更是写道:“1971年吴瑞的引物延伸,是测序的一个关键步骤,给奖是可以的”(这里指诺贝尔化学奖)。
1975年,Sanger在吴瑞的引物延伸测序技术的基础上,发表了自己的DNA测序方法6,1977年Gilbert等人建立了化学裂解法7,同一年,Sanger也发表文章改进了自己之前的测序方法,正式确立了目前第一代测序的主流方法-双脱氧链终止法(Sanger测序法)8。这项工作也使Sanger和Gilbert等获得了1980年的诺贝尔化学奖,值得一提的是,1958年Sanger就因完成了胰岛素化学结构的测定获得了当年的诺贝尔化学奖,是目前为止唯一一位两次获得化学奖的科学家。
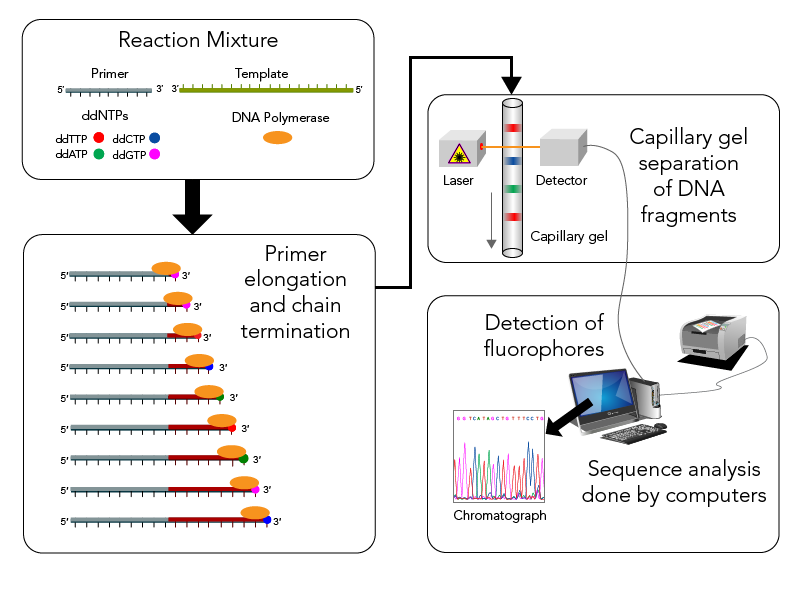
Sanger测序的原理参见图二:
- 使用DNA聚合酶延伸结合在待定序列模板上的引物,直到掺入一种链终止核苷酸为止。
- 每一次序列的测定由四管单独的反应构成,每个反应中含有所有的四种脱氧核糖核苷三磷酸(deoxy-ribonucleoside triphosphate,dNTP),并混入了一定量的双脱氧核苷三磷酸(dideoxy-ribonucleoside triphosphate,ddNTP)。
- 由于ddNTP缺少3’-OH基团,不能被DNA聚合酶延伸,会使延长的oligo选择性地在A、C、G或者T处终止,终止的位置由反应中相应的ddNTP决定。其中每一种的dNTPs和ddNTPs的浓度比例是可以调整的,使反应中可以得到不同长度的链终止产物。
- 通过高分辨率变性凝胶电泳分离长度不同的产物片段,就可以得出测序结果。
在Sanger时期,人们使用同位素标记和放射性显影来区分ddNTP,后来随着技术的发展,荧光标记代替了同位素标记,激光激发后使用自动成像系统进行检测,同时结合毛细管电泳技术的使用,大大提高了一代测序的准确性和速度。ABI的3730/3730XL系列自动测序仪就是目前主流的一代测序平台,在人类基因组计划中发挥了重要作用。
3 第二代测序技术
经过工业界数十年的改进,第一代测序的测定成本已经大幅下降,测定速度相对于最初的方法也有了极大的提高,但是因为对于电泳分离技术的依赖,第一代测序技术的通量和成本基本上已经达到了极限,所以人们开始寻找新的测序技术。在这个背景下,第二代测序技术(next generation sequencing,NGS)应运而生。
NGS,也称为高通量测序,可以一次性的对数十万到数百万的DNA分子进行序列测定。
2000年,Lynx Therapeutics公司推出了第一个高通量测序技术MPSS(Massively Parallel Signature Sequencing),后来该公司被Illumina公司收购9。
2005年,454 Life Sciences公司(后被Roche收购)推出了基于焦磷酸测序法的测序平台-Genome Sequencher 20 System,这是第一个商用的NGS测序平台。
2005年,Solexa公司公布了独特的边合成边测序技术(Sequencing by Synthesis,SBS),并测序了φ174噬菌体的基因组序列。2006年,Solexa公司推出了他们的1G Genetic Analyzer测序仪。该公司于2007年被Illumina公司收购,在不断的改良后,Illumina推出了GA IIx测序仪,并于2009年推出了HiSeq 2000测序仪。华大基因在2010年购入了128台HiSeq 2000测序仪。
2014年,Illumina公司推出了Hiseq X Ten机型,将10台Hiseq X连接起来,并宣称达成了1000美元基因组的目标。到了2016年,Illumina公司已经占据了70%的NGS市场份额,并且全球90%以上的测序数据都是基于Illumina的测序仪生成的。
Illumina测序的原理参见图三,以全基因组测序为:
- 先将DNA链打断成200-500bp的片段,末修加A后在两端连上特异性的接头P5和P7,扩增得到上机文库
- 带有接头的DNA片段流过flowcell,与其上固定的P5’接头通过互补配对结合,固定接头充当引物,在聚合酶的作用下进行合成反应
- DNA合成后,变性使得未与flowcell共价连接的DNA链解离并洗掉,反向链则得以保留。因为DNA单链另一端含有P7’接头,能够和临近的P7接头互补,DNA链形成桥式结构,同样的这个相邻的P7接头充当引物,在DNA聚合酶的作用下,合成双链。重复此过程,形成序列相同的DNA簇,使得在测序的过程中产生足够强的信号。变性打开,各自形成单链的DNA链固定在表面。将与P5’相连的反向链切除,留下与flowcell上P7相连的模板链,同时对游离的3’端进行封闭,防止不必要的反应。
- 加入测序引物,DNA聚合酶,4种带不同荧光标记的dNTP,且这些dNTP的3’端羟基被封闭,无法继续下一个反应。计算机检测到荧光的信号后,将不同的信号转化为对应的碱基。加入化学试剂淬灭信号,并去除3’的保护基团,并进行下一个碱基的反应。循环的次数取决于测序的长度,最终得到的是一条完整的DNA序列。
随着测序循环的增加,在扩增过程中,碱基添加出现错误的概率也随之增加、且基因簇复制的协同性降低,将导致测序质量下降,这使得一般第二代测序技术的读长都比较短,普遍不超过500bp。因此,测序完毕后需要经由生物信息技术作拼接,靠软件和算法来弥补技术上的不足,这使得NGS技术对于重复序列区域不能够很好的进行测定。

4 第三代测序技术
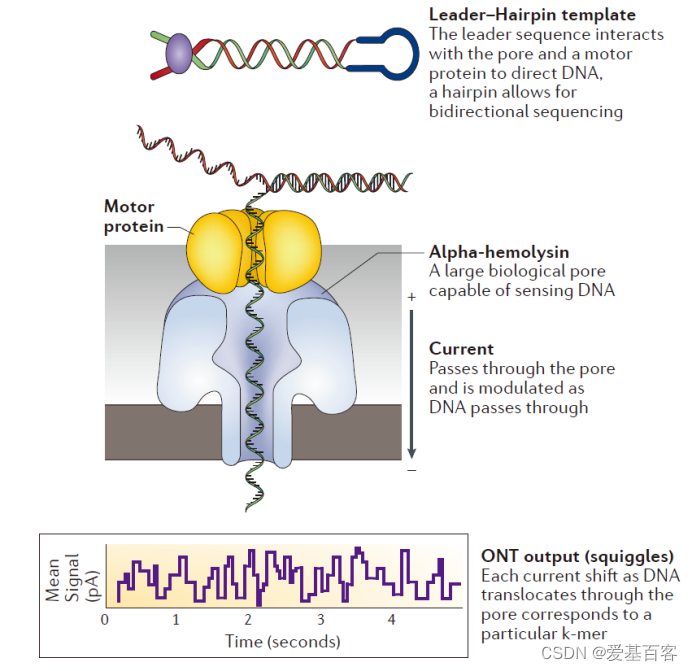
第三代测序技术以PacBio公司的SMRT技术和Oxford Nanopore Technologies纳米孔单分子测序技术为代表10。这两种技术的测序读长都非常的长,目前Nanopore测序的最长读长可达Mb级,解决了NGS技术因为读长短而带来的一系列基因组拼接问题。同时Nanopore测序还可以直接对RNA和核酸修饰等进行检测。
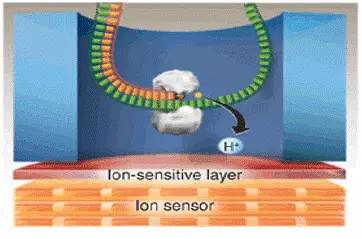
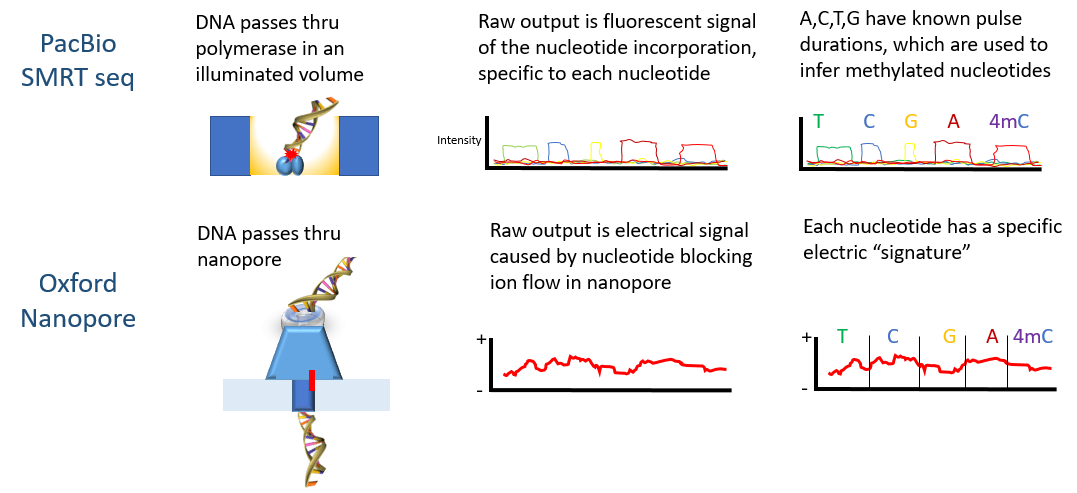
第三代测序的原理参见图四:
- PacBio SMRT技术应用了边合成边测序的思想,并以SMRT芯片为测序载体,芯片上有很多小孔,每个孔中均有DNA聚合酶。DNA聚合酶和模板结合,4色荧光标记4 种碱基(即是dNTP),在碱基配对阶段,不同碱基的加入,会发出不同光,根据光的波长与峰值可判断进入的碱基类型。
- Nanopore测序技术基于电信号而不是光信号。他们设计了一种只能容纳单分子通过的特殊纳米孔,孔内共价结合有分子接头。当DNA碱基通过纳米孔时,电荷发生变化,从而短暂地影响流过纳米孔的电流强度(每种碱基所影响的电流变化幅度是不同的),灵敏的电子设备检测到这些变化从而鉴定所通过的碱基。
参考文献:
wiki DNA sequencing ↩︎
人类科技史上彻底改变世界的三大工程 ↩︎
Wu R,Kaiser AD. Structure andbase sequence in the cohesive ends of bacteriophage lambda DNA. J Mol Biol.1968 Aug 14;35(3):523-37.https://doi.org/10.1016/s0022-2836(68)80012-9 ↩︎
Wu R. (1970). Nucleotide sequence analysis of DNA. I. Partial sequence of the cohesive ends of bacteriophage lambda and 186 DNA. Journal of molecular biology, 51(3), 501–521. https://doi.org/10.1016/0022-2836(70)90004-5 ↩︎
饶毅:君子爱“生” 得之有道 ↩︎
Sanger, F., & Coulson, A. R. (1975). A rapid method for determining sequences in DNA by primed synthesis with DNA polymerase. Journal of molecular biology, 94(3), 441–448. https://doi.org/10.1016/0022-2836(75)90213-2 ↩︎
Maxam, A. M., & Gilbert, W. (1977). A new method for sequencing DNA. Proceedings of the National Academy of Sciences of the United States of America, 74(2), 560–564. https://doi.org/10.1073/pnas.74.2.560 ↩︎
Sanger, F., Nicklen, S., & Coulson, A. R. (1977). DNA sequencing with chain-terminating inhibitors. Proceedings of the National Academy of Sciences of the United States of America, 74(12), 5463–5467. https://doi.org/10.1073/pnas.74.12.5463] ↩︎
A brief history of Next Generation Sequencing (NGS) ↩︎
Third-generation_sequencing ↩︎