Reactome 下载所有通路基因集
- Reactome 下载所有通路基因集
- Reactome 介绍
- ReactomePA
- 官网下载处理
Reactome 下载所有通路基因集
目前,网上有许多下载 KEGG、GO 数据库中所有富集通路的基因集方法,但是相对于 Reactome 来说却几乎没有。所以,我自己总结了下 Reactome 的下载方法。
比较方便主要有两种方法,一是使用 R 包 ReactomePA 可以很快速方便的得到我们想要的结果,但是有一个问题就是得到的可能与官网上会有点区别,毕竟不是最新的数据。二是从官网下载后自己处理,这个可能会稍微慢一些,但理解后结合自己的目标也很方便,并且数据跟官网是一样的。两种方法各有千秋,看自己选择就好。
Reactome 介绍
做通路分析的数据库目前有许多,但大家最熟悉的肯定还是 KEGG,Reactome 数据库与 KEGG 数据库类似,是研究 pathway 的数据库,汇集了人类各项反应及生物学通路,Reactome 目前在各大文章中也经常能够看到。关于其使用和界面网上一搜就非常之多,这里就不展示了。Reactome 我觉得可以与 KEGG 互相补充,毕竟 KEGG 目前也只覆盖了 8000 多个基因,还有半数以上没有注释到 KEGG。而且数据库还引用了 100 多个不同的在线生物信息学资源库,包括 NCBI、Ensembl、UniProt、UCSC 基因组浏览器、ChEBI 小分子数据库和 PubMed 文献数据库等。最重要的是,除了人类之外,Reactome 数据库还增加了许多其他物种,也就是说农学的部分物种可以使用该数据库进行代谢通路研究和富集分析了。


ReactomePA
现在先介绍第一种方法,使用 Y 叔的 R 包 ReactomePA 进行各通路所有基因集的整理:
```r
rm(list = ls())
# 安装 ReactomePA
BiocManager::install("ReactomePA")
library(ReactomePA)
library(reactome.db)
ls("package:reactome.db")[1] "reactome" "reactome.db" [3] "reactome_dbconn" "reactome_dbfile" [5] "reactome_dbInfo" "reactome_dbschema" [7] "reactomeEXTID2PATHID" "reactomeGO2REACTOMEID"[9] "reactomeMAPCOUNTS" "reactomePATHID2EXTID"
[11] "reactomePATHID2NAME" "reactomePATHNAME2ID"
[13] "reactomeREACTOMEID2GO"
keytypes(reactome.db)
[1] "ENTREZID" "GO" "PATHID" "PATHNAME" "REACTOMEID"# 将 Reactome 的通路信息和所含基因等信息提取作为 list
library(dplyr)
PathwayId_Genes <- as.list(reactomePATHID2EXTID) %>% .[grep("HSA",names(.))]
PathwayId_PathwayName <- as.list(reactomePATHID2NAME) %>% .[grep("HSA",names(.))]
PathwayName_PathwayId <- as.list(reactomePATHNAME2ID) %>% .[grep("HSA",.)]
Genes_PathwayId <- as.list(reactomeEXTID2PATHID) %>% .[grep("HSA",.)]
name_id <- unlist(lapply(PathwayName_PathwayId, function(x) x[[1]]))
unpair <- PathwayId_PathwayName[!names(PathwayId_PathwayName) %in% name_id]
names(unpair)[1] "R-HSA-9694614" "R-HSA-5683678" "R-HSA-4793953" "R-HSA-5579022"[5] "R-HSA-5663020" "R-HSA-5619114" "R-HSA-5619079" "R-HSA-5619044"[9] "R-HSA-5660724" "R-HSA-9694631" "R-HSA-9694719" "R-HSA-9694493"
[13] "R-HSA-9694594" "R-HSA-9694301" "R-HSA-9694548" "R-HSA-9694676"
[17] "R-HSA-9694635" "R-HSA-5660686" "R-HSA-9679509"
## PathwayId_PathwayName 和 PathwayName_PathwayId 其实是差不多的,就是两列数据刚好相反,但是我看了长度并不一样,前一个比后一个多了 19 条记录,目前我也还没明白为什么会这样,有知道的大佬请告知一下,谢谢。但是我查了这 19 条记录,基本是在 Disease 这一大块通路下面。
length(PathwayId_Genes)
[1] 2422
length(PathwayId_PathwayName)
[1] 2441
length(PathwayName_PathwayId)
[1] 2422
table(names(PathwayId_Genes) %in% name_id)
FALSE TRUE 18 2422
table(names(PathwayId_PathwayName) %in% name_id)
FALSE TRUE 19 2422
table(names(PathwayId_Genes) %in% names(PathwayId_PathwayName))
TRUE
2422
## 经过简单的探索,发现 PathwayId_Genes 的 pathway id 都能在 PathwayId_PathwayName 中找到,而在 PathwayName_PathwayId 中有 18 个 id 不匹配,虽然这两个文件长度相同。所以,根据这些情况我打算后续以 PathwayId_PathwayName 为主进行整理。# 可以看到我们已经把所有的通路 Id、Name 和所含基因都已经包含在上述列表中,下一步就是整理成表
PathwayId_PathwayName <- PathwayId_PathwayName[names(PathwayId_Genes)] #保持相同顺序
hsa_list <- mapply(c, PathwayId_Genes, PathwayId_PathwayName, SIMPLIFY=FALSE) # 合并list# 提取 pathway name
names = unlist(lapply(hsa_list , function(x) {paste(unlist(strsplit(x[length(x)],":"))[2])
}))
# 提取 pathway 所有 genes list
genes = unlist(lapply(hsa_list , function(x) {paste(unlist(x)[1:(length(x)-1)],collapse =';')
}))
# 合并
hsa_pathway = data.frame(Pathway_Id = names(hsa_list),Pathway_Name = names, Genes = genes)
length(hsa_pathway$Pathway_Id)
[1] 2422
## 至此,我们已经把基因 ENTREZID 提取出来了,下面就是 ID 转换的事情了,ID 转换的方法目前网上也一大堆,编程和网站方法都很丰富,比如这篇使用 R 和 python 进行 id 转换,https://zhuanlan.zhihu.com/p/338834196。最终结果如下,有 2422 条通路。按照开始那个表,智人相关通路是有 2580 条,这里只有 2422 条,其余的不知道哪去了,有知道的大佬请私信我,学习学习。
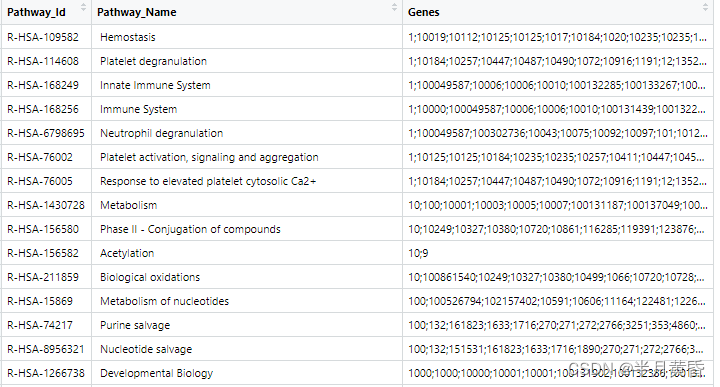
官网下载处理
官网下载链接: Reactome Download.
因为 Reactome 是开源数据库,所有数据和软件均可免费下载。进入网站可以看到有许多数据和资料,比如我们需要的通路信息、组织素材等。

在这里面根据自己的需求选择文件,右击选择复制链接,然后使用下载器进行下载。为什么不直接点击打开查看后再右击下载呢,是因为我的网访问很慢甚至出错,可能国内都有这种该情况。所以为了避免这一情况,还是用下载器下载比较快速,我用的是 Free Download Manager 下载,速度还是可以的。
下载后的数据是所有物种的,所以第一步就是选择自己的物种数据,这里使用 shell 处理比较方便,当然其他像 R 和 python 也是可以的。下面可以看到所有物种通路和单独属于人类相关通路的行数。
# 示例:Ensembl2Reactome.txt
grep "HSA" Ensembl2Reactome.txt > hsa_Ensembl2Reactome.txt # 选择智人物种
wc -l Ensembl2Reactome.txt hsa_Ensembl2Reactome.txt # 统计两个文件的行数
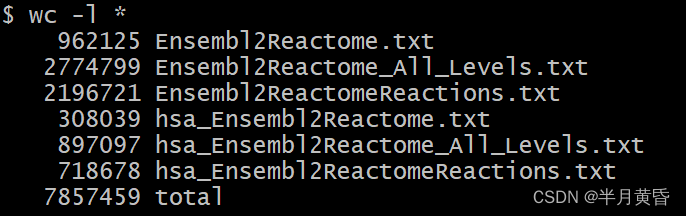
处理好后再用 R 语言进行汇总整理:
rm(list = ls())
pathway <- read.delim("hsa_Ensembl2Reactome.txt",header = F)
head(pathway)
ID <- pathway$V2[!duplicated(pathway$V2)] # pathway 第二列去重
all <- matrix(NA,nrow=length(ID),ncol=3) # 提前算好合并成的数据是几行几列,length(ID)行,3列
for (i in 1:length(ID)) {a <- pathway[pathway$V2==ID[i],]all[i,1] <- a[1,2]all[i,2] <- a[1,4]all[i,3] <- paste(a[,1],collapse = ",")
}
hsa_pathway <- as.data.frame(all)
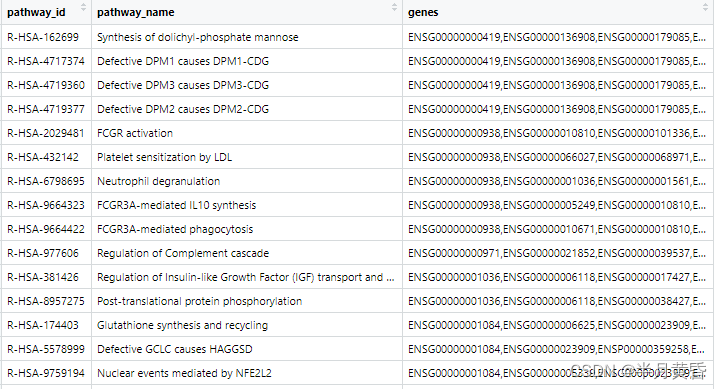
colnames(hsa_pathway) <- c("pathway_id","pathway_name","genes")
length(hsa_pathway2$pathway_id)
[1] 2066
最终结果如下图,有 2066 条通路,第一种方法得到的结果有 2422 条,这里可能是选择的文件的问题,我只是随意选了一个,以后有时间再试试其他的文件。但是无论如何,按照开始那个表,智人相关通路是有 2580 条的,但两种方法都达不到这个数据量,其余的不知道哪去了,有知道的大佬请私信我,学习学习,共勉。
在写之前,我是已经搜了很多文章也看了许多帖子,但现在一时也找不着了,下面列的参考文章只是一部分,请各位大佬见谅,如有侵权,请联系我删除。
参考文章:
[1]: https://wsa.jianshu.io/p/3ed19bd6e0ea
[2]: https://www.bioconductor.org/packages/release/bioc/html/ReactomePA.html
[3]: https://github.com/YuLab-SMU/ReactomePA
[4]: https://guangchuangyu.github.io/software/ReactomePA/
[5]: https://cloud.tencent.com/developer/article/1695223