**1、线性回归

例子
import numpy as np
import pylab
def compute_error(b,m,data):totalError = 0#Two ways to implement this#first way# for i in range(0,len(data)):# x = data[i,0]# y = data[i,1]## totalError += (y-(m*x+b))**2#second wayx = data[:,0]y = data[:,1]totalError = (y-m*x-b)**2totalError = np.sum(totalError,axis=0)return totalError/float(len(data))
def optimizer(data,starting_b,starting_m,learning_rate,num_iter):b = starting_bm = starting_m#gradient descentfor i in range(num_iter):#update b and m with the new more accurate b and m by performing# thie gradient stepb,m =compute_gradient(b,m,data,learning_rate)if i%100==0:print 'iter {0}:error={1}'.format(i,compute_error(b,m,data))return [b,m]def compute_gradient(b_current,m_current,data ,learning_rate):b_gradient = 0m_gradient = 0N = float(len(data))#Two ways to implement this#first way# for i in range(0,len(data)):# x = data[i,0]# y = data[i,1]## #computing partial derivations of our error function# #b_gradient = -(2/N)*sum((y-(m*x+b))^2)# #m_gradient = -(2/N)*sum(x*(y-(m*x+b))^2)# b_gradient += -(2/N)*(y-((m_current*x)+b_current))# m_gradient += -(2/N) * x * (y-((m_current*x)+b_current))#Vectorization implementationx = data[:,0]y = data[:,1]b_gradient = -(2/N)*(y-m_current*x-b_current)b_gradient = np.sum(b_gradient,axis=0)m_gradient = -(2/N)*x*(y-m_current*x-b_current)m_gradient = np.sum(m_gradient,axis=0)#update our b and m values using out partial derivationsnew_b = b_current - (learning_rate * b_gradient)new_m = m_current - (learning_rate * m_gradient)return [new_b,new_m]def plot_data(data,b,m):#plotttingx = data[:,0]y = data[:,1]y_predict = m*x+bpylab.plot(x,y,'o')pylab.plot(x,y_predict,'k-')pylab.show()def Linear_regression():# get train datadata =np.loadtxt('data.csv',delimiter=',')#define hyperparamters#learning_rate is used for update gradient#defint the number that will iteration# define y =mx+blearning_rate = 0.001initial_b =0.0initial_m = 0.0num_iter = 1000#train model#print b m errorprint 'initial variables:\n initial_b = {0}\n intial_m = {1}\n error of begin = {2} \n'\.format(initial_b,initial_m,compute_error(initial_b,initial_m,data))#optimizing b and m[b ,m] = optimizer(data,initial_b,initial_m,learning_rate,num_iter)#print final b m errorprint 'final formula parmaters:\n b = {1}\n m={2}\n error of end = {3} \n'.format(num_iter,b,m,compute_error(b,m,data))#plot resultplot_data(data,b,m)if __name__ =='__main__':Linear_regression()
2、卡方分布**
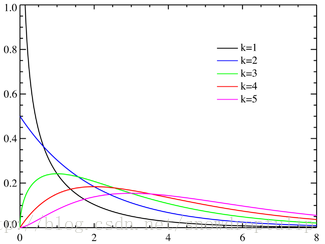
若n个相互独立的随机变量ξ₁、ξ₂、……、ξn ,均服从标准正态分布N(0,1)(也称独立同分布于标准正态分布),则这n个服从标准正态分布的随机变量的平方和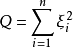 构成一新的随机变量,其分布规律称为分布(chi-squaredistribution)。其中参数n称为自由度(通俗讲,样本中独立或能自由变化的自变量的个数,称为自由度),正如正态分布中均值或方差不同就是另一个正态分布一样,自由度不同就是另一个
构成一新的随机变量,其分布规律称为分布(chi-squaredistribution)。其中参数n称为自由度(通俗讲,样本中独立或能自由变化的自变量的个数,称为自由度),正如正态分布中均值或方差不同就是另一个正态分布一样,自由度不同就是另一个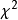 分布。记为
分布。记为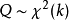 。 分布的均值为自由度 n,记为若n个相互独立的随机变量ξ₁、ξ₂、……、ξn ,均服从标准正态分布N(0,1)(也称独立同分布于标准正态分布),则这n个服从标准正态分布的随机变量的平方和构成一新的随机变量,其分布规律称为分布(chi-squaredistribution)。其中参数n称为自由度(通俗讲,样本中独立或能自由变化的自变量的个数,称为自由度),正如正态分布中均值或方差不同就是另一个正态分布一样,自由度不同就是另一个分布。记为。 分布的均值为自由度 n,记为 E( x^2) = n; 分布的方差为2倍的自由度(2n),记为 D( ) = 2n。; 分布的方差为2倍的自由度(2n),记为 D( x^2) = 2n。
。 分布的均值为自由度 n,记为若n个相互独立的随机变量ξ₁、ξ₂、……、ξn ,均服从标准正态分布N(0,1)(也称独立同分布于标准正态分布),则这n个服从标准正态分布的随机变量的平方和构成一新的随机变量,其分布规律称为分布(chi-squaredistribution)。其中参数n称为自由度(通俗讲,样本中独立或能自由变化的自变量的个数,称为自由度),正如正态分布中均值或方差不同就是另一个正态分布一样,自由度不同就是另一个分布。记为。 分布的均值为自由度 n,记为 E( x^2) = n; 分布的方差为2倍的自由度(2n),记为 D( ) = 2n。; 分布的方差为2倍的自由度(2n),记为 D( x^2) = 2n。

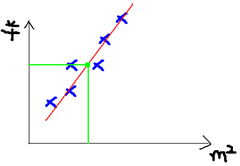
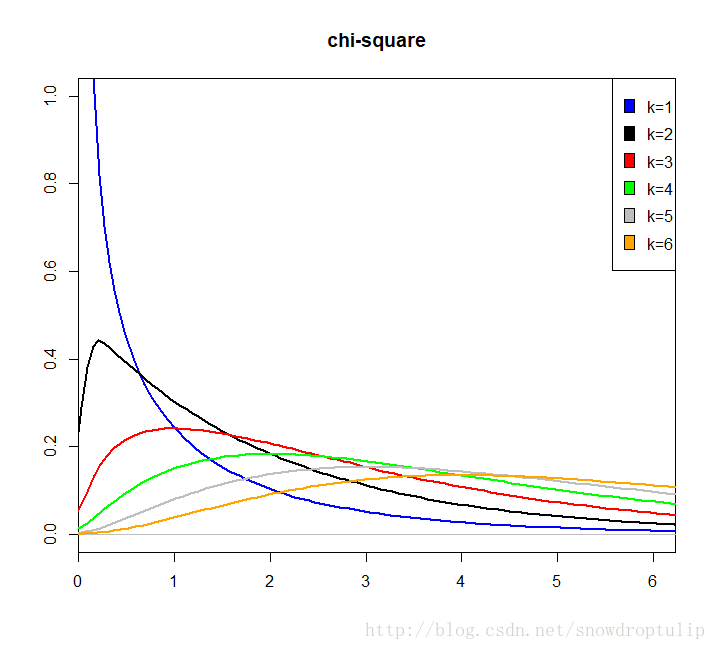
从卡方分布图可以看出:卡方分布在第一象限内,卡方值都是正值,呈正偏态(右偏态),随着参数 n 的增大;卡方分布趋近于正态分布;随着自由度n的增大,卡方分布向正无穷方向延伸(因为均值n越来越大),分布曲线也越来越低阔(因为方差2n越来越大)。
3.方差分析
方差分析----单因素方差分析/双因素方差分析
ANOVA 由英国统计学家R.A.Fisher首创,为纪念Fisher,以F命名,故方差分析
又称 F 检验 (F test)。
单因素方差分析:众多因素中仅有一个因素的的水平有多个,其余因素只有一个水平。
多因素方差分析:多个因素有多个水平。
方差分析的主要功能就是验证两组样本,或者两组以上的样本均值是否有显著性差异,即均值是否一样。
这里有两个大点需要注意:①方差分析的原假设是:样本不存在显著性差异(即,均值完全相等);②两样本数据无交互作用(即,样本数据独立)这一点在双因素方差分析中判断两因素是否独立时用。
原理:
方差分析的原理就一个方程:SST=SS组间+SSR组内 (全部平方和=组间平方和+组内平方和)
说明:方差分析本质上对总变异的解释。
组间平方和=每一组的均值减去样本均值
组内平方和=个体减去每组平方和
方差分析看的最终结果看的统计量是:F统计量、R2。

其中:g为组别个数,n为每个组内数据长度。
from scipy import stats
from statsmodels.formula.api import ols
from statsmodels.stats.anova import anova_lm
from statsmodels.stats.multicomp import pairwise_tukeyhsd
import warnings
warnings.filterwarnings("ignore")import itertoolsdf2=pd.DataFrame()
df2['group']=list(itertools.repeat(-1.,9))+ list(itertools.repeat(0.,9))+list(itertools.repeat(1.,9))df2['noise_A']=0.0
for i in data['A'].unique():df2.loc[df2['group']==i,'noise_A']=data.loc[data['A']==i,['1','2','3']].values.flatten()df2['noise_B']=0.0
for i in data['B'].unique():df2.loc[df2['group']==i,'noise_B']=data.loc[data['B']==i,['1','2','3']].values.flatten() df2['noise_C']=0.0
for i in data['C'].unique():df2.loc[df2['group']==i,'noise_C']=data.loc[data['C']==i,['1','2','3']].values.flatten()
# for A
anova_reA= anova_lm(ols('noise_A~C(group)',data=df2[['group','noise_A']]).fit())
print(anova_reA)
#B
anova_reB= anova_lm(ols('noise_B~C(group)',data=df2[['group','noise_B']]).fit())
print(anova_reB)
#C
anova_reC= anova_lm(ols('noise_C~C(group)',data=df2[['group','noise_C']]).fit())
print(anova_reC)
参考:https://github.com/Shicoder/DeepLearning_Demo/tree/master/linear_regression_use_gradient_decent
http://www.shujuren.org/article/104.html
https://www.cnblogs.com/jin-liang/p/9852321.html
http://blog.sciencenet.cn/blog-2577109-1143281.html
https://www.cnblogs.com/jin-liang/p/9852321.html






![[考研数学]概率论难点总结:样本标准差,样本均值,均值的期望和方差,与t分布、卡方分布和F分布的关系及推导](https://img-blog.csdnimg.cn/20201016120115461.png?x-oss-process=image/watermark,type_ZmFuZ3poZW5naGVpdGk,shadow_10,text_aHR0cHM6Ly9ibG9nLmNzZG4ubmV0L3F3ZTkwMA==,size_16,color_FFFFFF,t_70#pic_center)