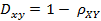期刊名:Bioinformatics
分区:Q1
发表:2022年1月8号
代码数据集:GitHub - CSUBioGroup/BACPI
一、摘要
二、数据与方法
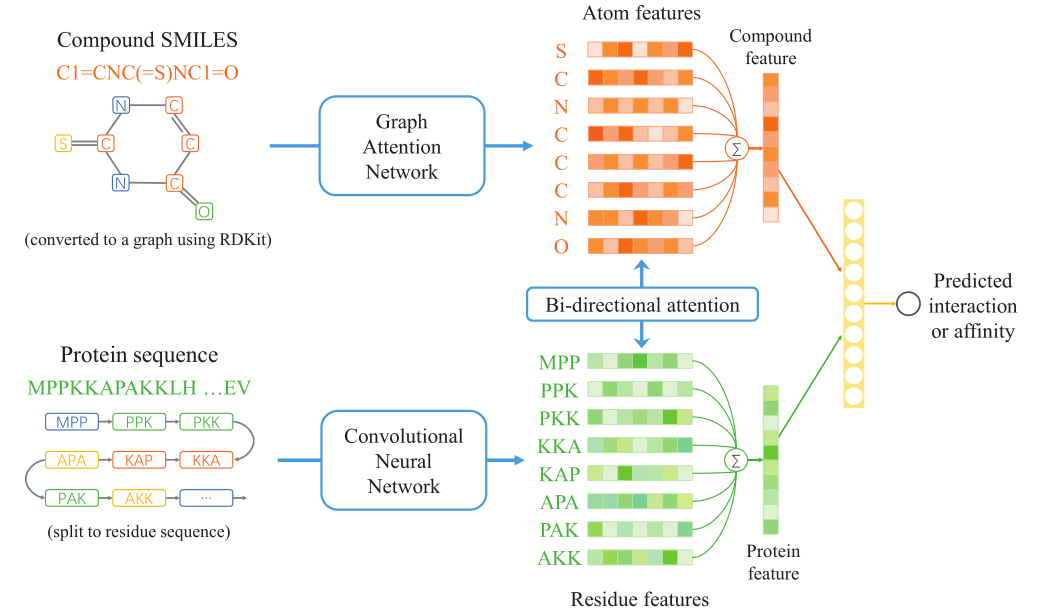
通过GAT和CNN的端到端学习获得的特征来表示化合物和蛋白质。对于化合物,我们使用
RDKit将化合物的SMILES格式转换为图形表示,并使用GAT学习化合物的特征表示,可以提取图
形的各种信息,如原子类型、芳香度、化学键类型等。对于蛋白质,我们的CNN以该蛋白质的氨
基酸序列作为输入,学习该蛋白质的特征表示。最后,利用我们的双向注意神经网络整合化合物和
蛋白质的表征,预测输入的化合物和蛋白质对的相互作用和结合亲和力。给定一组复合蛋白对标签
(交互或亲和力),培训目标是最小化损失函数(叉CPI损失预测任务和亲和力的预测均方误差),使用反
向传播来优化权重矩阵和偏差向量,CNN与双向注意神经网络
由于真实的CPI数据集通常是不平衡的,我们设置了不同的正负样本比率(如1:1、1:3和1:5)来评估
预测模型的稳健性。
三、结果


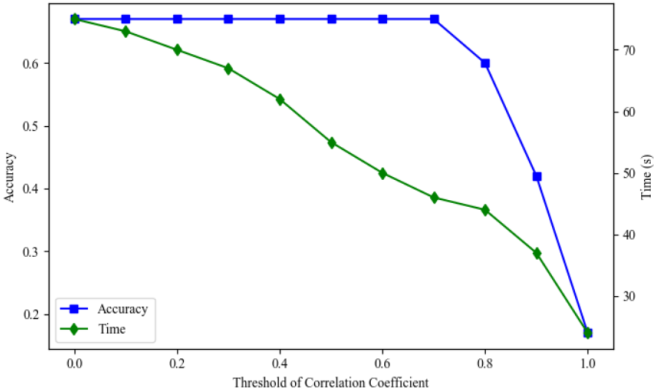
解释:图2和图3显示了人类和秀丽隐杆线虫数据集上的AUC和AUPR得分。可以看出,在
两个数据集上,与这些方法相比,提出的BACPI方法取得了最好的性能。这表明,端到端表示学习
方法可以探索有用的化合物和蛋白质的性质,以进行下游预测。我们还发现,当阳性样本和阴性样
本比例增加时,大多数方法的AUC得分保持稳定或略有上升,而所有方法的AUPR得分均有所下
降。这主要是因为召回和准确性都集中在少数类,这导致AUPR比AUC更惩罚假阳性。因此,在处
理不平衡数据集时,AUPR对方法的性能给出了更准确的评估,而AUC可能提供了对性能的乐观看
法。可以看到,在不平衡数据集(正、负样本比为1:3或1:5)上,我们的方法在AUPR方面明显优于
其他方法在人类和线虫数据集上的AUPR。实验结果表明,BACPI在不平衡数据集上具有良好的稳
定性和可靠性。

评价表2、3:深度学习方法比机器学习方法(脊回归、套索回归和RF)具有更高的准确率。这主要
是因为深度学习具有强大的特征学习能力。
深度学习方法比机器学习方法(脊回归、套索回归和RF)具有更高的准确率。这主要是因为深度学习
具有强大的特征学习能力。
表3显示了不同预测方法对4个数据集的皮尔逊相关系数(PCC)结果。BACPI方法在IC50和Ki数据集
上的效果最好,在EC50数据集上的效果与MONN和deepurpose相同,在Kd数据集上的效果次之。
所有的比较结果表明BACPI可以有效地预测化合物和蛋白质的结合亲和力。

表4显示了前10名候选药物和3种无关药物的预测结果。这些结果表明,在10种推荐药物中,有7种已经被许多研究证明了它们对SARS-CoV2的复制抑制作用。我们发现Darunavir、Cobicistat、Ritonavir和Ivermectin是目前正在接受2019年治疗冠状病毒病临床试验的四种候选药物。相比之下,三种无关药物(阿莫西林、青霉素和阿司匹林)对3CLPro靶标的吸引力和结合亲和力较弱,分别在87个药物中排名第78、80和82位。这些实验结果证实了BACPI在筛选再利用候选者方面的可靠性。