【数据库】SRA数据库介绍及数据下载
生信技术 2021-10-06 11:00
以下文章来源于生信Alpha ,作者BioinfoPenn
生信Alpha.
生物信息、生物统计、Linux系统、shell、R、Python等日常学习记录分享~欢迎交流指正~
【数据库】SRA数据库介绍及数据下载 - 目录
-
1. SRA数据库介绍
-
(1) SRP开头的ID:PRJNA = SRP
-
(2) PRJNA开头的ID:SAMN = SRS
-
(3) SRX开头的ID
-
(4) SRR开头的ID
-
-
2. SRA数据的下载
-
(1) 使用Aspera Connect 软件下载
-
(2) 使用sratoolkit中的prefetch命令下载
-
(3) 使用SRA Explorer下载
-
(4) 使用wget直接下载
-
(5) 使用SRAdbV2 R包下载
-
(6) 批量创建链接,迅雷下载
-
1. SRA数据库介绍
NCBI - SRA(Sequence ReadArchive)数据库是NCBI用于存储二代测序的原始数据,包括 454,Illumina,SOLiD,IonTorrent等,这个数据库是可以免费无限制下载的。当然,也可以到EBI - ENA数据库直接下载fastq .gz文件。
根据SRA数据产生的特点,将SRA数据分为四类,这四种分类有一个层次关系,SRA数据库用不同的前缀加以区分。
数据的层级结构是:SRP(项目研究/Studies) → SRX(实验设计/Experiments) → SRS(样本信息/Samples) → SRR(测序结果集/Runs)
伴随数据库是project,层级是PRJNA → SAMN
注:runs表示测序仪运行所产生的reads。一个Study可能包含多个Experiment,Experiments包含了Sample、DNA source、测序平台、数据处理等信息。一个Sample也可能对应多个Experiments。
一般文章里面会给出数据地址,例如,根据文章的GSE号进入GEO数据库里面,就可以看到其对应的SRA数据库ID号。https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE84498
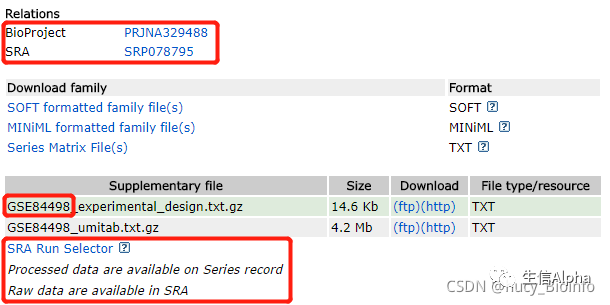
测试数据
后续讲解采用以下文章的数据:
文章:Liu H, Murphy CJ, Karreth FA, Emdal KB, White FM, Elemento O, Toker A, Wulf GM, Cantley LC.Identifying and Targeting Sporadic Oncogenic Genetic Aberrations in Mouse Models of Triple-Negative Breast Cancer. Cancer Discov. 2018 Mar;8(3):354-369. doi: 10.1158/2159-8290.CD-17-0679.
文章下载地址:http://cancerdiscovery.aacrjournals.org/content/candisc/early/2018/02/14/2159-8290.CD-17-0679.full.pdf
数据地址:
https://www.ncbi.nlm.nih.gov/sra/?term=SRP115453
https://www.ebi.ac.uk/ena/browser/view/PRJNA398328?show=reads
(1) SRP开头的ID:PRJNA = SRP
文章如果提到其数据上传到了SRA数据库,那么就会给出SRP开头的ID。如:The sequencing data have been deposited in the NCBI Sequence Read Archive (SRA) database under the accession code SRP115453.
① 查看样本列表:https://www.ncbi.nlm.nih.gov/sra?term=SRP115453,可以看到该数据集共有182个数据。
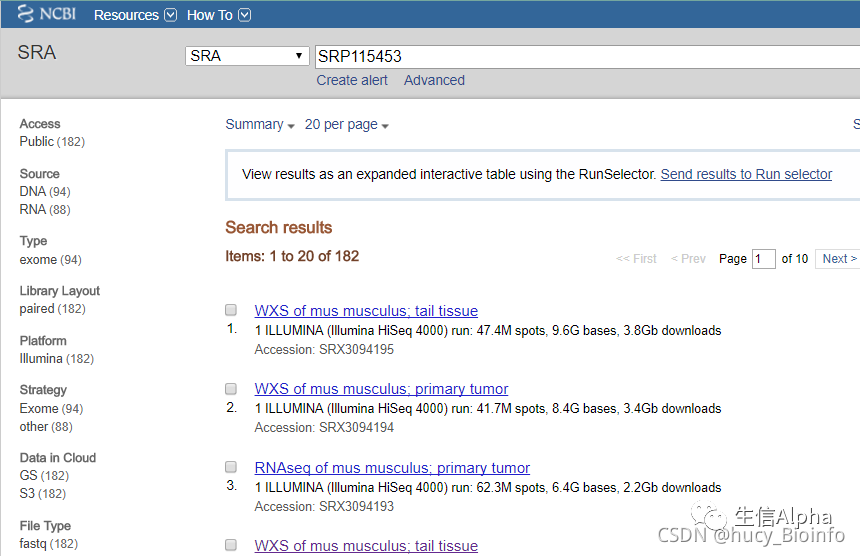
② 查看该project有哪些数据:https://www.ncbi.nlm.nih.gov/Traces/study/?acc=SRP115453;数据共1.35 T,有点大。可以根据研究目的进行样本筛选,挑选样本后,点击"Accession List"键, 下载得到SRR List 储存在SRR_Acc_List.txt 文件中。点击Metadata即可下载测序数据的相应信息,存储在SraRunTable.txt文件中。
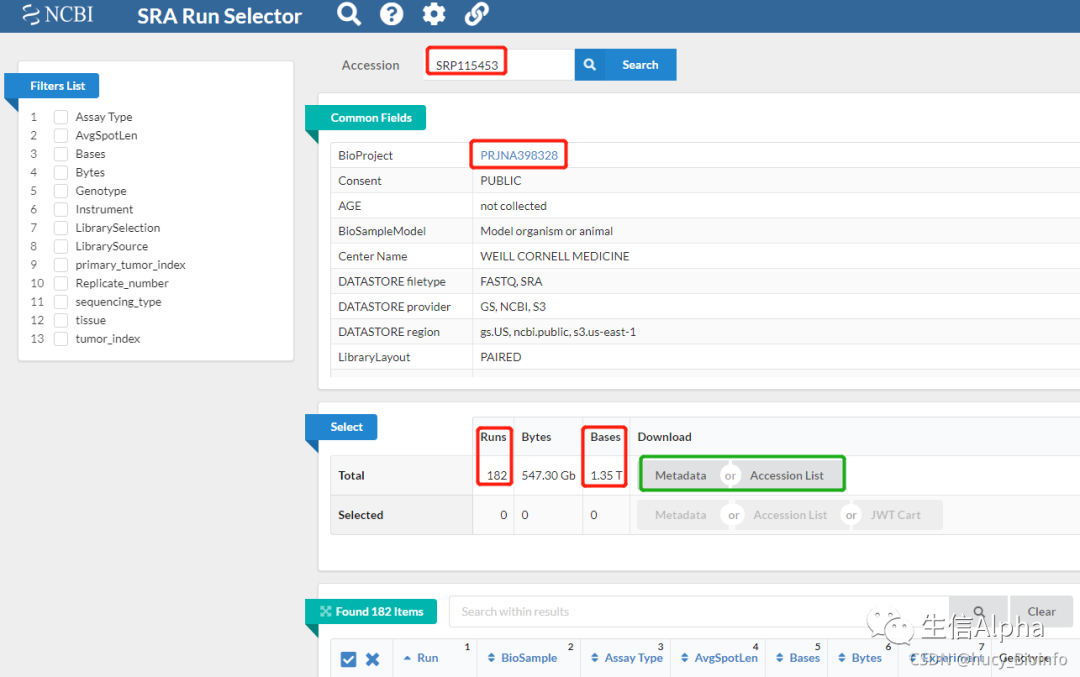
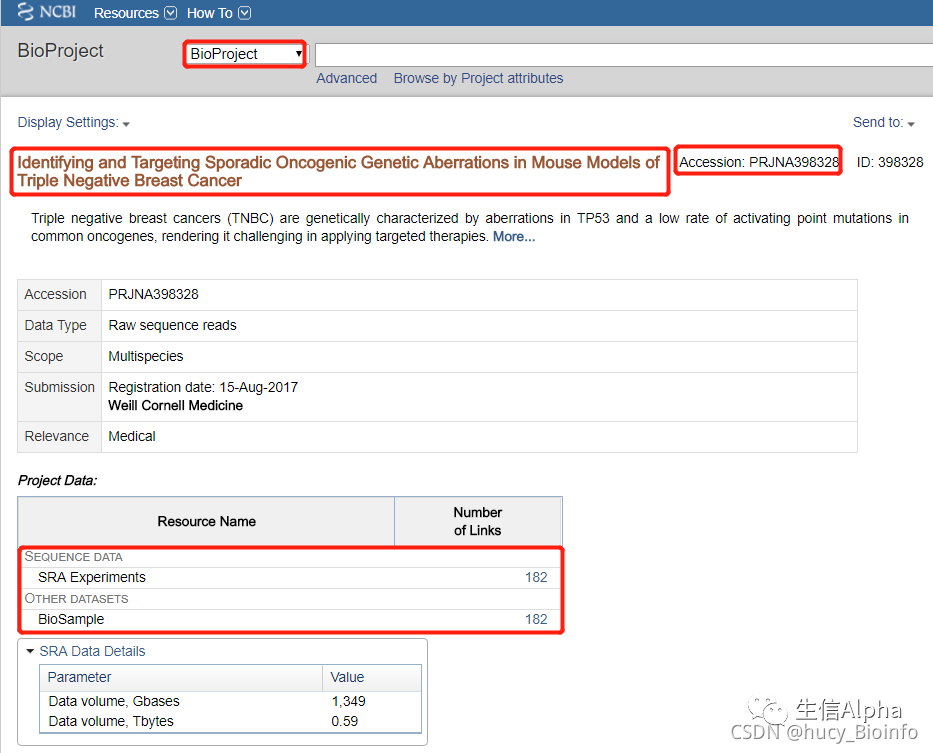
(2) PRJNA开头的ID:SAMN = SRS
PRJNA开头的ID等同于 SRP开头的ID。进入链接,可以看到发表的文章,以及涉及到的样本:https://www.ncbi.nlm.nih.gov/bioproject/PRJNA398328;该项目一共182个样本。
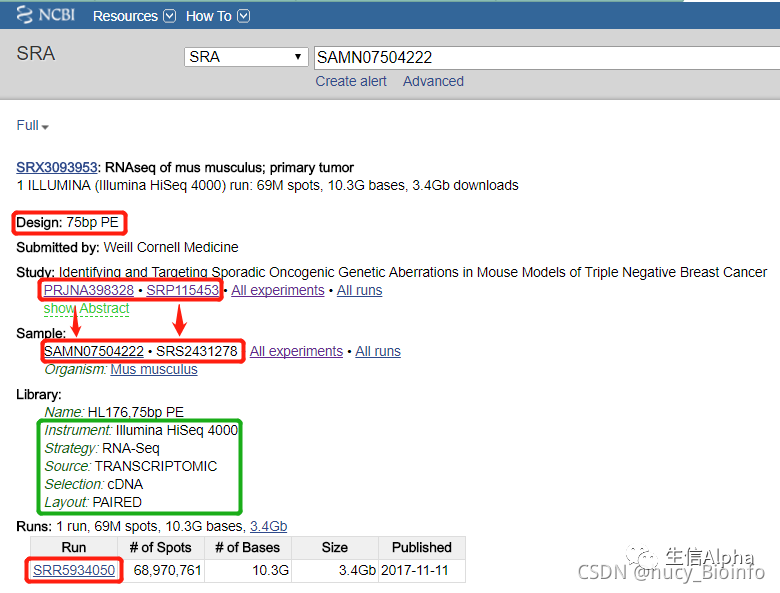
查看每个样本的情况:
链接:https://www.ncbi.nlm.nih.gov/sra?term=SAMN07504222;可以看到每个样本对应1个测序数据。SAMN开头的ID等同于SRS开头的ID。
当然,也有同一样本存在多种测序数据的情况,如:https://www.ncbi.nlm.nih.gov/sra?term=SAMN05341212 ;该样本存在6个数据。
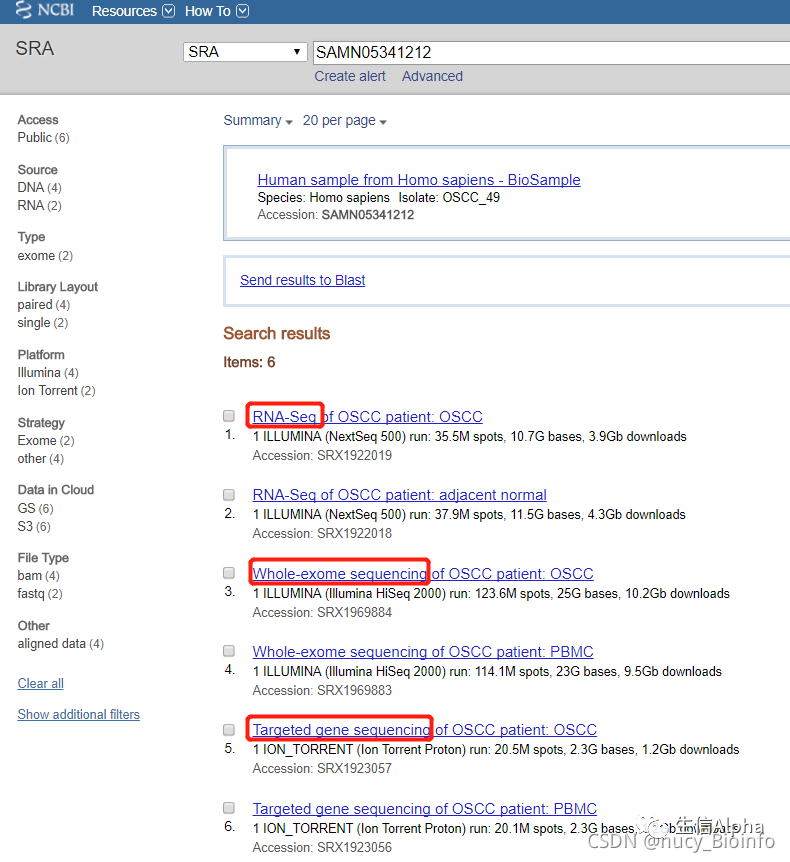
(3) SRX开头的ID
对于上例,每个样本6个数据的情况,还可以进入每个测序数据,如,https://www.ncbi.nlm.nih.gov/sra/SRX1922019
(4) SRR开头的ID
最后是,查看测序数据本身的信息:
https://trace.ncbi.nlm.nih.gov/Traces/sra/?run=SRR5934050
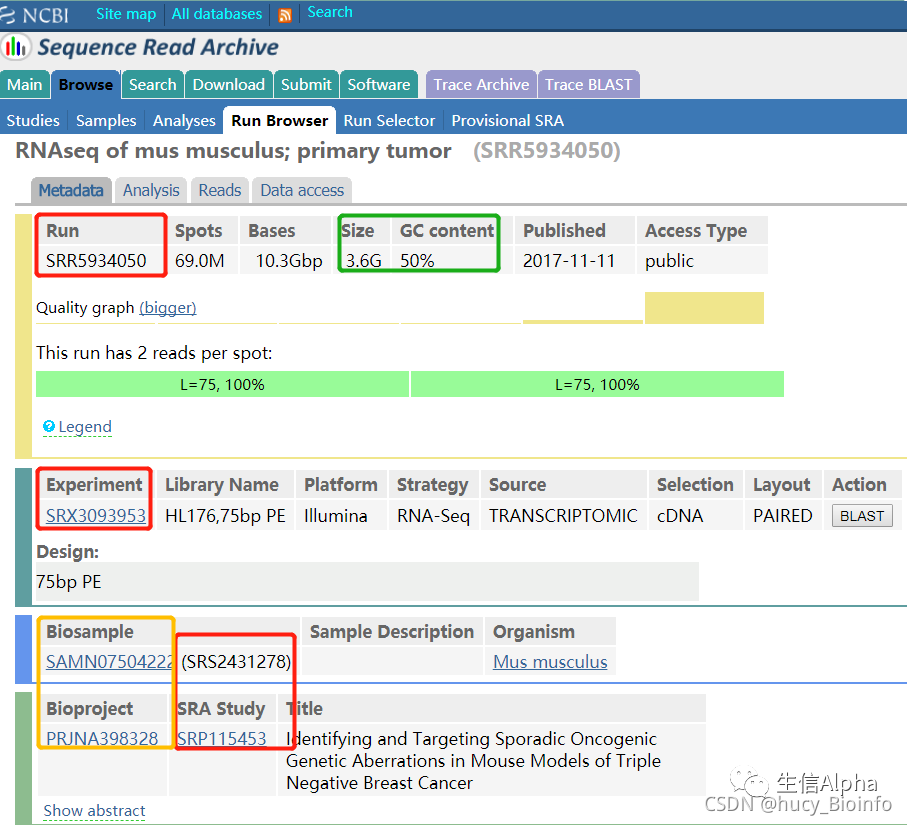
注:网页相应的位置均有链接可直接跳转到对应的数据页面。
2. SRA数据的下载
sra数据的下载可以通过网页端下载,但是比较不方便。可借助以下软件工具来进行下载。
(1) 使用Aspera Connect 软件下载
Aspera Connect (http://downloads.asperasoft.com/en/downloads/8?list) 软件,是IBM旗下的商业高速文件传输软件,与NCBI和EBI有协作合同,可以免费使用其下载高通量测序文件。下载完成后,本地用fastq-dump提取fastq文件,用sam-dump提取SAM 文件。
1) 下载并解压安装aspera
wget http://download.asperasoft.com/download/sw/connect/3.7.4/aspera-connect-3.7.4.147727-linux-64.tar.gz
tar -xvzf aspera-connect-3.7.4.147727-linux-64.tar.gz
bash aspera-connect-3.7.4.147727-linux-64.sh
2) 设置环境变量
echo 'export PATH=~/.aspera/connect/bin:$PATH' >> ~/.bashrc
source ~/.bashrc
# EBI-ENA数据的存放地址为 fasp.sra.ebi.ac.uk,ENA在Aspera的用户名是era-fasp。
3) 获取sra下载链接
EBI - ENA下载链接获取:https://www.ebi.ac.uk/ena/browser/view/PRJNA398328?show=reads;点击下图红框中的TSV,即可下载得到182个测序数据的下载链接。
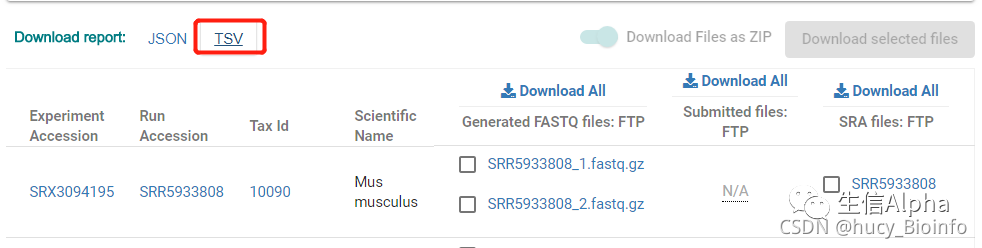
原始数据:ftp://ftp.sra.ebi.ac.uk/vol1/srr/SRR593/008/
fastq数据:ftp://ftp.sra.ebi.ac.uk/vol1/fastq/SRR593/008/
如:
-
study_accession:PRJNA398328
-
sample_accession:SAMN07504110
-
experiment_accession:SRX3094195
-
run_accession:SRR5933808
-
tax_id:10090
-
scientific_name:Mus musculus
-
fastq_ftp:ftp.sra.ebi.ac.uk/vol1/fastq/SRR593/008/SRR5933808/SRR5933808_1.fastq.gz;ftp.sra.ebi.ac.uk/vol1/fastq/SRR593/008/SRR5933808/SRR5933808_2.fastq.gz
-
sra_ftp:ftp.sra.ebi.ac.uk/vol1/srr/SRR593/008/SRR5933808
也可批量自行构建网址:
SRR593/008/SRR5933808/SRR5933808_1.fastq.gz为替换的路径掌握规律后可批量构建。
fastq
链接:ftp://ftp.sra.ebi.ac.uk/vol1/fastq/SRR前3位/00最后1位/SRR号/SRR号_1或者2.fastq.gz
例如:ftp://ftp.sra.ebi.ac.uk/vol1/fastq/SRR593/008/SRR5933808/SRR5933808_1.fastq.gz
sra
链接:ftp://ftp.sra.ebi.ac.uk/vol1/srr/SRR前3位/00最后1位/SRR号
例如:ftp://ftp.sra.ebi.ac.uk/vol1/srr/SRR593/008/SRR5933808
4) 使用ascp命令下载
Aspera用法如下:
ascp --help
# ascp [参数] 目标文件 保存路径
# -v verbose mode 实时知道程序在干什么
# -T 取消加密,否则有时候数据下载不了
# -i 提供私钥文件的地址
# -l 设置最大传输速度,一般200m到500m,如果不设置,反而速度会比较低,可能有个较低的默认值
# -k 断点续传,一般设置为值1
# -Q 一般加上它
# -P 提供SSH port,端口一般是33001
# export ASPERA_SCP_PASS=~/.aspera/connect/etc/asperaweb_id_dsa.openssh
ascp -vQT -k1 -l 400m -P33001 -i ~/.aspera/connect/etc/asperaweb_id_dsa.openssh era-fasp@ftp.sra.ebi.ac.uk:vol1/srr/SRR593/008/SRR5933808 ./ &ascp -vQT -k1 -l 400m -P33001 -i ~/.aspera/connect/etc/asperaweb_id_dsa.openssh era-fasp@fasp.sra.ebi.ac.uk:vol1/fastq/SRR593/008/SRR5933808 ./ &#era-fasp是aspera在ENA的用户名,fasp.sra.ebi.ac.uk是固定下载地址,需要记住。
#数据默认存储在当前路径的SRR5933808目录下
(2) 使用sratoolkit中的prefetch命令下载
SRA Tookit 是NCBI 提供的下载软件,需要选择合适的版本下载安装。
下载地址:https://trace.ncbi.nlm.nih.gov/Traces/sra/sra.cgi?view=software
SRA Toolkit Documentation:https://trace.ncbi.nlm.nih.gov/Traces/sra/sra.cgi?view=toolkit_doc
Frequently Used Tools:
fastq-dump: Convert SRA data into fastq format
prefetch: Allows command-line downloading of SRA, dbGaP, and ADSP data
sam-dump: Convert SRA data to sam format
sra-pileup: Generate pileup statistics on aligned SRA data
vdb-config: Display and modify VDB configuration information
vdb-decrypt: Decrypt non-SRA dbGaP data (“phenotype data”)
1) 下载
wget https://ftp-trace.ncbi.nlm.nih.gov/sra/sdk/2.11.0/sratoolkit.2.11.0-ubuntu64.tar.gz
tar xzvf sratoolkit.2.11.0-ubuntu64.tar.gz -C ~/softwares# 如有su权限,可直接下载:
su
apt install sra-toolkit# 也可使用conda安装
conda install sra-tools
2) 配置环境变量
echo "export PATH=$PATH:YOUR_PATH/sratoolkit.2.11.0-ubuntu64.tar.gz/bin" >> ~/.bashrc
source ~/.bashrc
prefetch -h
3) prefectch命令下载数据
# prefetch下载的数据在home目录下的ncbi目录里。
prefetch SRR5933808 & #下载单个SRA文件
prefetch --option-file SRR_Acc_List.txt #下载多个SRA文件默认下载的是sra格式数据,可以使用fastq-dump将sra转换为fastq了。
fastq-dump --gzip --split-3 SRR5933808.sra# 也可以直接使用fastq-dump下载数据,下载之后直接即使fastq格式。
# 不过还是选择prefetch比较好,因为sra数据格式比fastq格式占用空间较小,下载速度快;另一方面,sra也方便断点续传。
# 直接利用fastq-dump下载数据
fastq-dump --split-files SRR5933808 SRR5933808/
4) 文件拆分及格式转换
fastq-dump --split-files SRR5933808.sra
fastq-dump --split-files SRR5933808
fastq-dump --gzip --split-3 SRR5933808
# --split-spot: 将双端测序分为两份,但是都放在同一个文件中
# --split-files: 将双端测序分为两份,放在不同的文件,但是对于一方有而一方没有的reads直接丢弃
# --split-3: 将双端测序分为两份,放在不同的文件,但是对于一方有而一方没有的reads会单独放在一个文件夹里
# --gzip: 输出文件为gzip压缩格式
注:当不确定到底是单端还是双端的SRA文件,一律用--split-3。
其他工具使用示例:
# ftp://ftp.ncbi.nlm.nih.gov/sra/data/smoke_test.sh#!/bin/bash
echo "Public data dump started SRR4065643"
./fastq-dump SRR4065643
./fastq-dump --split-files SRR4065643
./sam-dump SRR4065643 > SRR4065643.sam
rm -rf SRR4065643*
./fasterq-dump SRR4065643
./prefetch --type any SRR4065643
rm -rf SRR4065643*
echo "Public data dump ended"echo "Starting dbGaP with ngc"
./fastq-dump --ngc ~/prj_phs710EA_test.ngc SRR1219879
rm -rf SRR1219879*
./fasterq-dump --ngc ~/prj_phs710EA_test.ngc SRR1219879
./sam-dump --ngc ~/prj_phs710EA_test.ngc SRR1219879
./prefetch --ngc ~/prj_phs710EA_test.ngc SRR1219879
rm -rf SRR1219879*
echo "NGC test done"
(3) 使用SRA Explorer下载
由瑞典斯德哥尔摩的Phil Ewels开发的网页下载工具:https://sra-explorer.info/#
支持输入:GSE、SRA、SRP、PRJ、EPR,甚至直接通过物种+组织+测序技术搜索
即使一个SRX包括超过一个数据集的时候(多个SRR),也会返回结果,例如:SRX1067547包含两个run。
(4) 使用wget直接下载
找到数据下载链接后直接使用如下命令下载sra或者fastq数据。
wget -c -t 0 -O SRR5933809 ftp.sra.ebi.ac.uk/vol1/srr/SRR593/009/SRR5933809
wget -c -t 0 -O SRR5933809_1.fastq.gz ftp.sra.ebi.ac.uk/vol1/fastq/SRR593/009/SRR5933809/SRR5933809_1.fastq.gz &
# -c -t 配合使用可以防止下载数据的过程中链接中断的问题,-O则可以指定下载路径和文件名。
(5) 使用SRAdbV2 R包下载
可以使用SRAdbV2 R包获取SRA数据:https://seandavi.github.io/SRAdbV2/articles/SRAdbv2.html
BiocManager::install('seandavi/SRAdbV2')
library(SRAdbV2)# 使用SRAdbV2 首先需要创建一个 R6类-Omicidx
oidx = Omicidx$new()# 创建好Omicidx实例后,就可以使用oidx$search()来进行数据检索
query=paste(paste0('sample_taxon_id:', 10116),'AND experiment_library_strategy:"rna seq"','AND experiment_library_source:transcriptomic','AND experiment_platform:illumina')
z = oidx$search(q=query,entity='full',size=100L)
# z = oidx$search(q="PRJNA398328",entity='full',size=100L)
# 其中,entity 参数是指可以通过API获得的SRA实体类型, size 参数指查询结果返回的记录数
(6) 批量创建链接,迅雷下载
获取SRR ID后,批量创建下载链接,粘贴到迅雷即可下载。
# 根据SRR_Acc_List.txt文件一键自动创建下载链接
cat SRR_Acc_List.txt|while read a ;do t1=${a:0:6};t2=${a:9:9};echo "ftp://ftp.sra.ebi.ac.uk/vol1/srr/$t1/00$t2/$a/$a.sra";done
# ftp.sra.ebi.ac.uk/vol1/srr/SRR593/008/SRR5933808
# ftp.sra.ebi.ac.uk/vol1/srr/SRR593/009/SRR5933809
# ftp.sra.ebi.ac.uk/vol1/srr/SRR593/000/SRR5933810
# ftp.sra.ebi.ac.uk/vol1/fastq/SRR593/008/SRR5933808/SRR5933808_1.fastq.gz
注:构造ftp链接后使用wget下载数据的缺点:依赖网络情况,速度较慢,容易出错。
参考阅读:
生信技能树 - 解读SRA数据库规律一文就够
生信技能树 - SRA数据库的数据并不一定要在SRA数据库下载
百迈客医学 - SRA数据下载的几种常见方法
生信星球 - 也许是最方便的SRA数据下载方式
基因学苑如 - 何下载生物数据(四):SRA数据下载