一、前期准备
Linux系统安装Freesurfer、MATLAB插件spm12、fieldtrip,Windows下载mricron、Slicer3D(需要插件SlicerFreeSurfer)软件
文件准备
- ct 和mri t1 文件,格式为dicom,需要转换为 nii 格式(可在spm中转换)转换nii详细介绍
- edf 文件,包含脑电的所有数据
- 电极位置图,手术计划
二、Ubantu下进行脑区分割
准备工作
1、安装好matlab和freesurfer
2、在目标位置创建文件夹
3、开放权限

4、将需要计算的nii文件放入文件夹
使用matlab进行计算
需要提前下载好fieldtrip并解压,设置路径(包括全部子文件夹)
1、打开mri_cut.m文件,修改位置subject00x,和ct文件的名字(代码可参考fieldtrip官网)
clc; clear; close all;subjID = 'subject006';
ct_fname = 's0001195183-0005-00001-000384-01.nii';
mri =ft_read_mri(fullfile('/media/huang/data2/zhuqianyun/sEEG',subjID,ct_fname));ft_determine_coordsys(mri);
cfg = [ ];
cfg.method = 'interactive';
cfg.coordsys = 'acpc';mri_acpc = ft_volumerealign(cfg,mri);
cfg = [ ];
cfg.filename = ['/media/huang/data2/zhuqianyun/sEEG/',subjID, '/',subjID, '_MR_acpc'];
cfg.filetype = 'nifti';
cfg.parameter = 'anatomy';
ft_volumewrite(cfg, mri_acpc)fshome = '/home/zhuqianyun/freesurfer/';
subdir = ['/media/huang/data2/zhuqianyun/sEEG/',subjID, '/'];
mrfile = [cfg.filename,'.nii'];
system(['export FREESURFER_HOME=' fshome '; ' ...
'source $FREESURFER_HOME/SetUpFreeSurfer.sh; ' ...
'mri_convert -c -oc 0 0 0 ' mrfile ' ' [subdir '/tmp.nii'] '; ' ...
'recon-all -i ' [subdir '/tmp.nii'] ' -s ' 'freesurfer' ' -sd ' subdir ' -all'])2、运行程序
3、定位(需要自己确认)
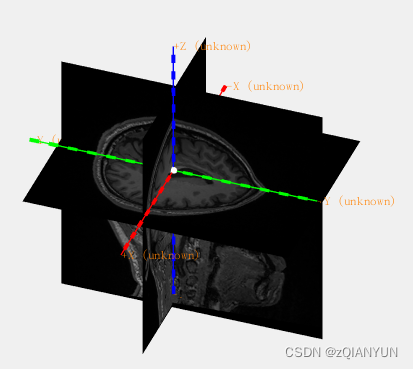
采用的方向是RAS,右前上(具体方向介绍见fieldtrip官网)
RAS表示第一维指向右,第二维指向前,第三维指向上。
ACPC 坐标系中使用的界标是前连合和后连合(AC 和 PC)
- TT坐标系原点在AC
- y 轴沿着连接 PC 和 AC 的线朝向大脑前部(p 到 a)
- z 轴朝向大脑的顶部(s 到 i)
- x轴朝向大脑的右侧(l 到 r)
代码运行后,先弹出坐标轴方向,确认为RAS(一般都正确),在命令行输入n,不用修改
然后进行acpc定位
1. To change the slice viewed in one plane, either:a. click (left mouse) in the image on a different plane. Eg, to view a moresuperior slice in the horizontal plane, click on a superior position in thecoronal plane, orb. use the arrow keys to increase or decrease the slice number by one
2. To mark a fiducial position or anatomical landmark, do BOTH:a. select the position by clicking on it in any slice with the left mouse buttonb. identify it by pressing the letter corresponding to the fiducial/landmark:press a for ac, p for pc, z for xzpointpress r for an extra control point that should be on the right sideYou can mark the fiducials multiple times, until you are satisfied with the positions.
3. To change the display:a. press c on keyboard to toggle crosshair visibilityb. press f on keyboard to toggle fiducial visibilityc. press + or - on (numeric) keyboard to change the color range's upper limit
4. To finalize markers and quit interactive mode, press q on keyboard
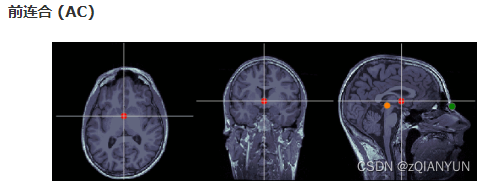
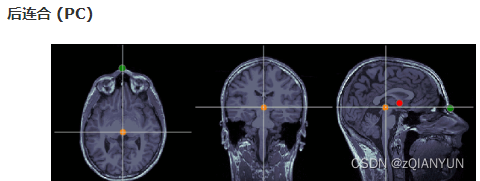
首先定位ac和pc,选好后键盘分别按a和p。
接下来是头顶,键盘按z
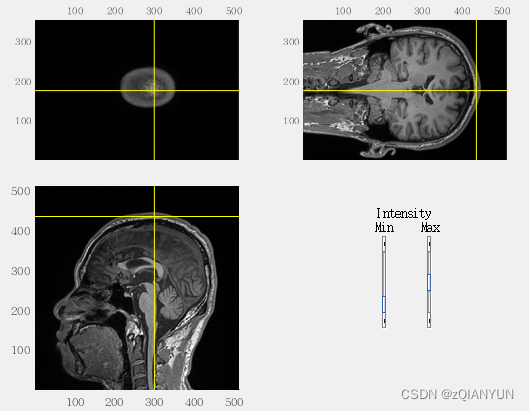
最后是右耳,键盘按r,注意是切线位置

最后输入q完成定位

需要等待比较久的时间进行计算。
生成文件
最终生成freesurfer文件夹和两个另外的nii文件
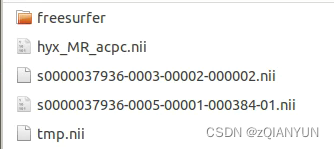
将这些文件拷贝下来就可以到Windows系统里进行配准和定点了
三、配准(Windows操作)
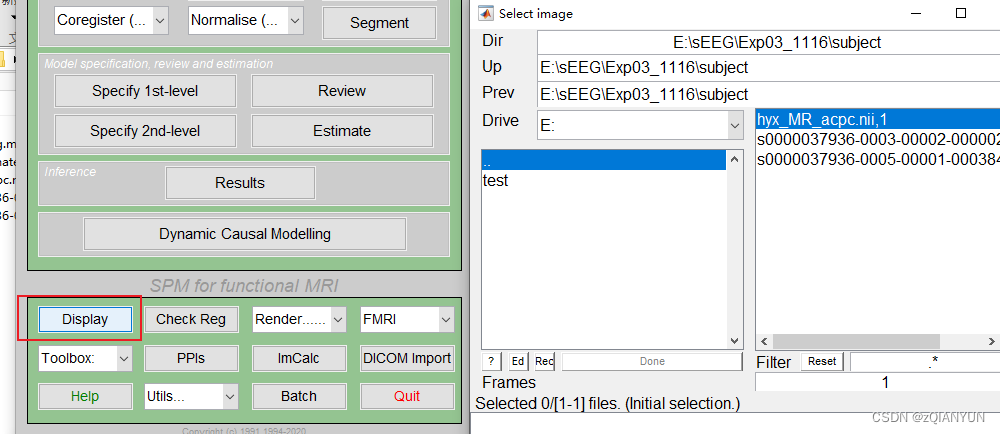
在matlab中打开spm,选择fMRI后界面如下
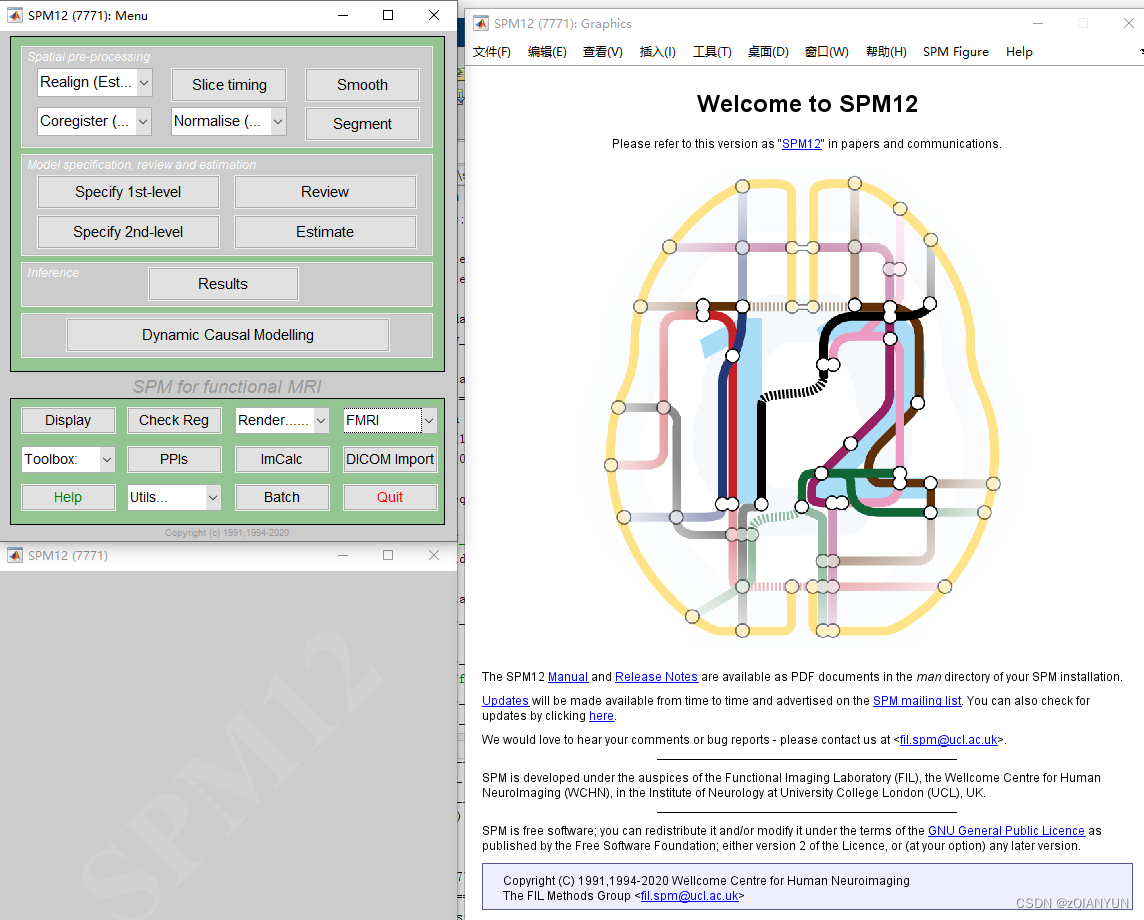
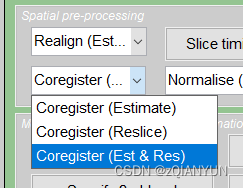
配准选择(Est & Res)
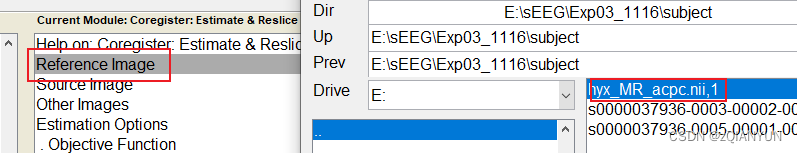
导入两个数据(nii),ref选择MRI(MR-acpc),source选择CT
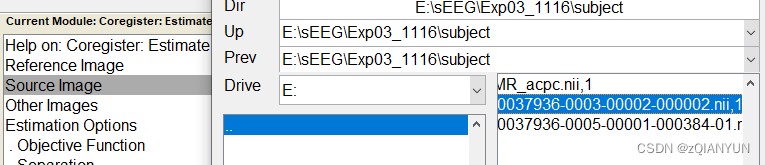
完成后点击运行
完成后会在文件夹里生成一个rs的nii文件
在mricron软件中打开rs文件看是否配准,这样就是完成啦
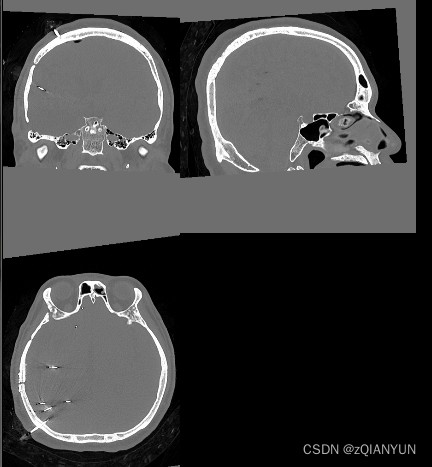
如果在配准过程出现如下情况,就是距离原点太远

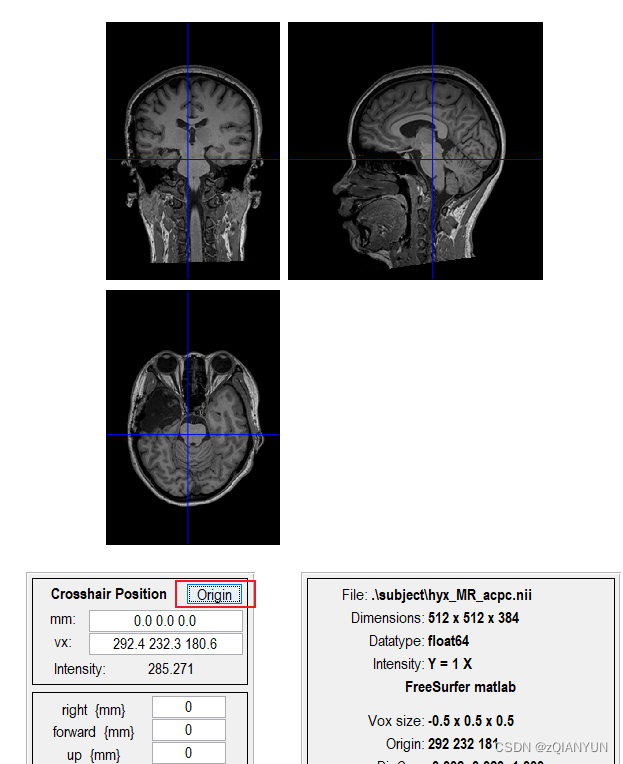
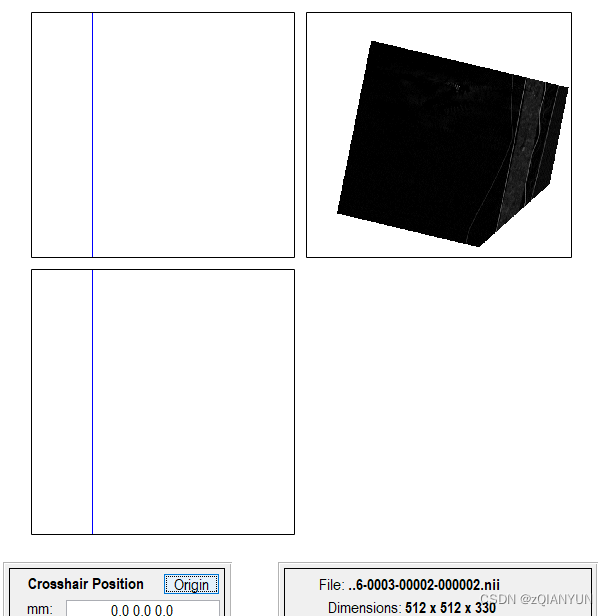
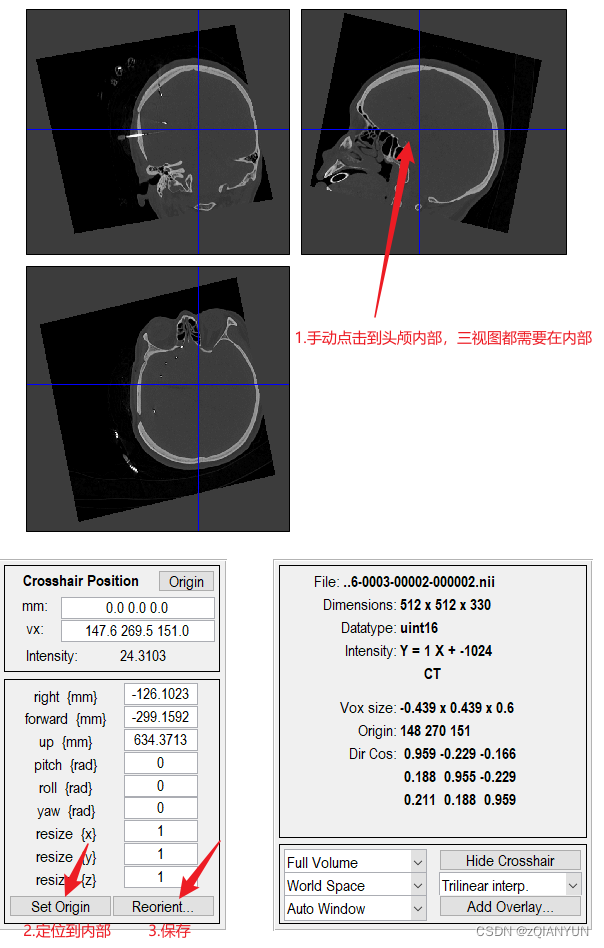
需要手动选择original,一个一个看
这个图像的原点在头颅内部
这个不在内部,需要手动调整
三、电极定点(Windows操作)
软件:3D slicer,matlab
模型生成
使用slicer4.11的软件,记得要添加SlicerFreeSurfer插件
- 文件导入slicer中
(1)分割的freesurfer文件夹下的 aparc+aseg.mgz
(2)生成的MR_acpc文件
(3)配准的rs 文件

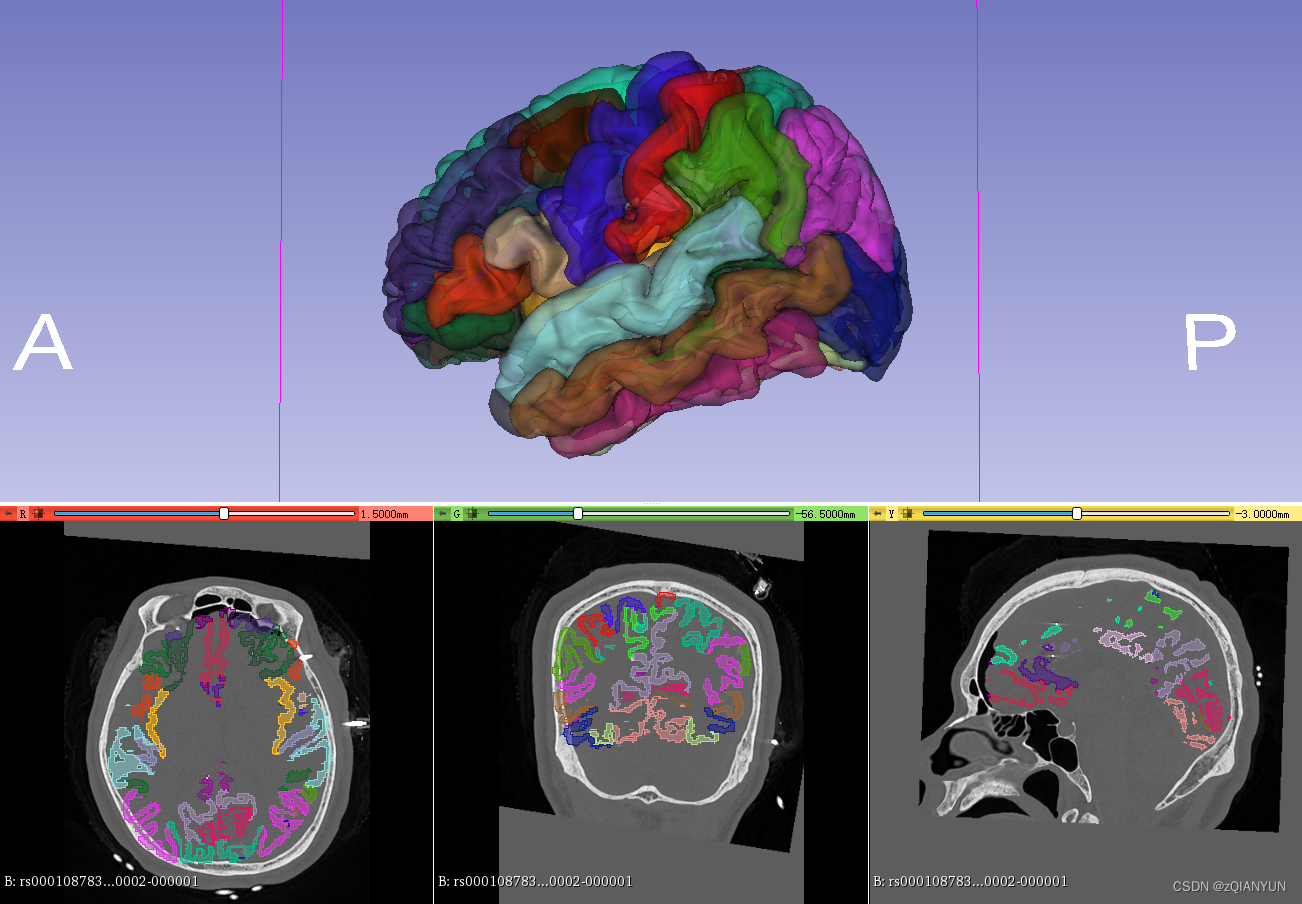
导入后生成可视化图

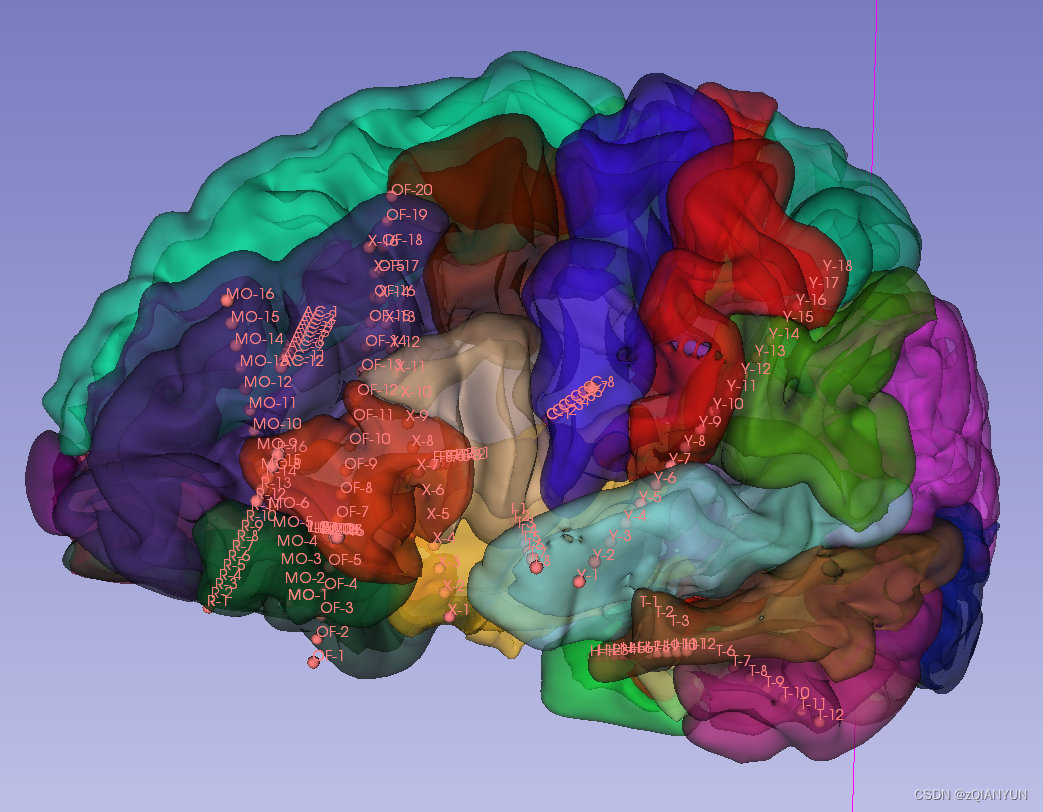
调整透明度和需要显示的区域,这里显示的是左脑皮层部分

电极定位
三视图放大,按照电极位置进行定位,需要手动定两个点,一个最里面的一个最外面的
调整一下rs文件的阈值,电极点可以显示出黑色圆圈,方便定点

最里面的电极点需要三个视图都在中心,最外面的电极点可以定在颅骨上,按照电极名称修改电极点名称。勾选菜单栏下的persistent可以连续点基准点。


全部电极标记完成后,保存整个文件夹为slicer0后关闭软件。

计算电极位置(自动定点)
打开matlab,运行cclt_coordinate_v2.m程序(需要提前设置路径,jsonlab-master。注意把程序放在与slicer0同一个文件夹下)
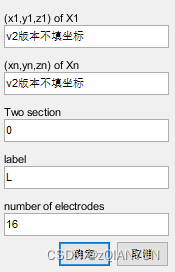
cood_info = inputdlg({'Two section','label','number of electrodes'},...,'Calculate coordinates of electrode V2',1,{'0','',''},'on');
d = 3.5;
path = [pwd,'\'];
labeln = cell2mat(cood_info(4));
jsfile = loadjson([path,'slicer0\',labeln,'.mrk.json']);
jsfile.markups.controlPoints(3:str2num(cell2mat(cood_info(5)))) = jsfile.markups.controlPoints(2);
x1 = jsfile.markups.controlPoints(1).position;
xn = jsfile.markups.controlPoints(2).position;
for n = 2:8jsfile.markups.controlPoints(n).id = num2str(n);jsfile.markups.controlPoints(n).label = [labeln,'-',num2str(n)];k = ((xn(1)-x1(1))^2+(xn(2)-x1(2))^2+(xn(3)-x1(3))^2)^0.5/3.5/(n-1);jsfile.markups.controlPoints(n).position = x1+(xn-x1)/k;
end
if ~str2num(cell2mat(cood_info(3)))for n = 9:str2num(cell2mat(cood_info(5))) %% 16jsfile.markups.controlPoints(n).id = num2str(n);jsfile.markups.controlPoints(n).label = [labeln,'-',num2str(n)];k = ((xn(1)-x1(1))^2+(xn(2)-x1(2))^2+(xn(3)-x1(3))^2)^0.5/3.5/(n-1);jsfile.markups.controlPoints(n).position = x1+(xn-x1)/k;end
elsefor n = 9:str2num(cell2mat(cood_info(5)))jsfile.markups.controlPoints(n).id = num2str(n);jsfile.markups.controlPoints(n).label = [labeln,'-',num2str(n)];k = ((xn(1)-x1(1))^2+(xn(2)-x1(2))^2+(xn(3)-x1(3))^2)^0.5/(7*3.5+10+2*(n-8)+(n-9)*1.5);jsfile.markups.controlPoints(n).position = x1+(xn-x1)/k;end
endsavepath = [path,'chanloc\'];
if exist(savepath)==0mkdir(savepath);
end
savejson('',jsfile,'filename',[savepath,labeln,'.mrk.json'],'IntFormat','%8.1f');
按照手术计划表写电极名称和电极点数量
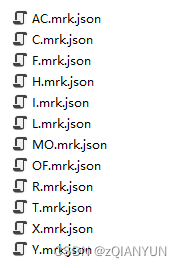
生成了.mrk.json文件
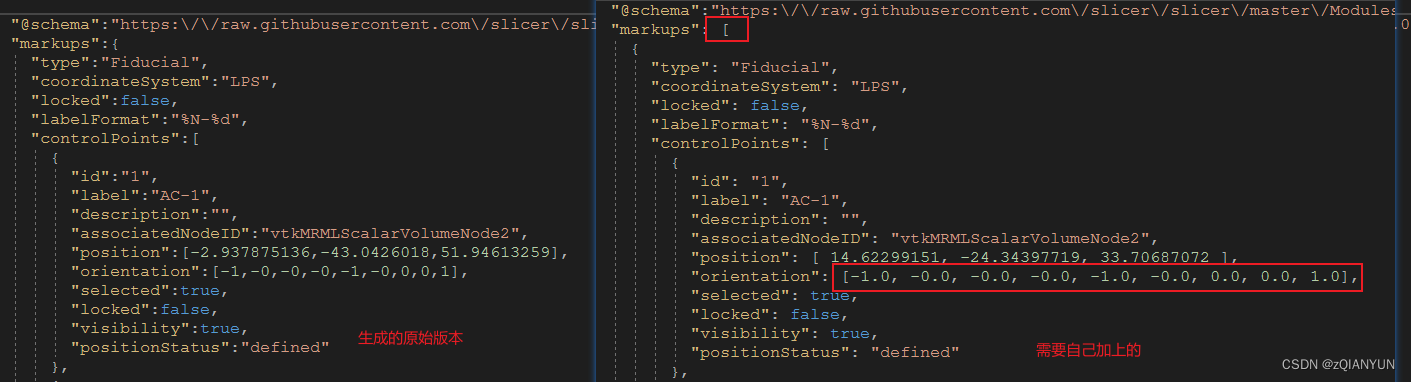
打开修改里面的括号和crtl+F 替换数字(1→1.0)
替换slicer文件夹里相应的json文件
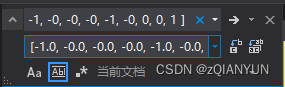
再打开3Dslicer即完成了电极定点