#自己一些问题:里面有sparcc?
#学习网站 https://github.com/stefpeschel/NetCoMi
#Github本地安装 在上面网站找到下载
#devtools::install_local("C:/Users/xxx/Documents/NetCoMi-1.0.2.tar.gz")
#加载包 安装在了R-3.6版本
library(NetCoMi)
#Control组
otu1=read.csv("L6绝对丰度filter10_Control.csv",row.names = 1,header=T)
otu1=t(otu1)
#Case组
otu2=read.csv("L6绝对丰度filter10_OSCC.csv",row.names = 1,header=T)
otu2=t(otu2)
#data=【rows are samples】, 【columns are OTUs/taxa】
net_single2 <- netConstruct(data=otu1, data2=otu2,
measure = "sparcc", # 可选:spearman spieceasi sparcc
#使用 clr 变换作为归一化方法
normMethod = "clr",
zeroMethod = "none",
sparsMethod = "threshold",adjust = "adaptBH",
#采用0.3的阈值作为稀疏化方法,只连接绝对相关性大于或等于0.3的OTU
thresh = 0.3, seed=123456,
filtTax = "none",
verbose = 3)
summary(net_single2)
#??netConstruct
#网络分析和绘图 ??netAnalyze
props_single2 <- netAnalyze(net_single2,
centrLCC = TRUE,
clustMethod = "cluster_fast_greedy",
hubPar = c("degree", "closeness","eigenvector"))
#hubPar = c("degree", "between", "closeness","eigenvector")) 也可以用其中一个筛选出核心物种
summary(props_single2)
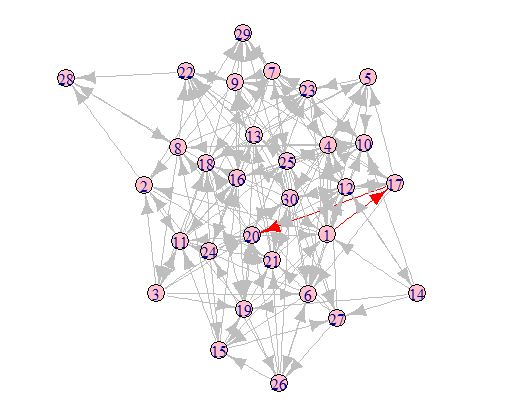
p1=plot(props_single2,
nodeColor = "cluster",
nodeSize = "clr",
repulsion = 0.8,
rmSingles = TRUE,
#所有标签大小相等=false
labelScale = FALSE,
#改变圆圈的字体大小
cexLabels = 0.6,
#如果该值减小,则节点大小更相似
#(与cexNodes结合)对于扩大小节点同时保持大节点的大小很有用
nodeSizeSpread = 3,
#改变圆圈的大小
cexNodes = 4,
#显示圆圈里面标签的大小
shortenLabels= "none",
title1 = "Network on OTU level with spearman correlations",
showTitle = TRUE,
cexTitle = 1.5)
#加载 门信息,这个如下 主要要门水平信息
| ID | genus | phyla |
| Absconditabacteria_(SR1)_[G-1] | Absconditabacteria_(SR1)_[G-1] | Absconditabacteria_(SR1) |
| Actinomyces | Actinomyces | Actinobacteria |
| Schaalia | Schaalia | Actinobacteria |
| Rothia | Rothia | Actinobacteria |
| Corynebacterium | Corynebacterium | Actinobacteria |
| Atopobium | Atopobium | Actinobacteria |
taxtab=read.csv("taxtab.csv",row.names = 1,header=T)
phyla <- as.factor(taxtab[, 2]) #Genus.1
phyla
names(phyla) <- taxtab[, "genus"]
phylcol <- c("blue","yellow", "orangered", "#009E73","#A2CD5A",
"peru" ,"#0072B2" ,"dodgerblue")
p2=plot(props_single2,
nodeColor = 'feature',
featVecCol = phyla,
colorVec = phylcol,
nodeSize = "clr",
repulsion = 0.8,
#所有标签大小相等=false
labelScale = TRUE,
#分组设定
layout = "spring",
sameLayout = TRUE,
layoutGroup = 1,
rmSingles = "none",#inboth两组没有才去掉
nodeFilter = "none",
nodeFilterPar = NULL,
#改变圆圈的字体大小
cexLabels = 1,
#如果该值减小,则节点大小更相似
#(与cexNodes结合)对于扩大小节点同时保持大节点的大小很有用
nodeSizeSpread = 4,
#改变圆圈的大小
cexNodes = 4,
title1 = "Network on OTU level with Pearson correlations",
showTitle = TRUE,
cexTitle = 1,
posCol = "#009900",
negCol = "#BF0000",
hubBorderCol = "red",
#使用相同的布局,则只能删除两个组中都未连接的节点。
groupNames = c("Control","OSCC")) #圈上面的分组名称
#简单图
p1=plot(props_single2,
nodeColor = "feature",
featVecCol = phyla,
colorVec = phylcol,
nodeSize = "clr",
repulsion = 0.6,
rmSingles = TRUE,
#所有标签大小相等=false
labelScale = FALSE,
#改变圆圈的字体大小
cexLabels = 0.7,
#如果该值减小,则节点大小更相似
#(与cexNodes结合)对于扩大小节点同时保持大节点的大小很有用
nodeSizeSpread = 3,
#改变圆圈的大小
cexNodes = 6,
#显示圆圈里面标签的长度
shortenLabels= "none",
title1 = "Network on OTU level with spearman correlations",
showTitle = TRUE,
posCol = "#009900",
negCol = "#BF0000",
hubBorderCol = "red",
groupNames = c("Control","OSCC"),#圈上面的分组名称
cexTitle = 1)
zoom=1 # 控制图片缩放比例
#导出的参数设置png 1000 500 1500 750
#导出的参数设置pdf 7.5 15
#检查最大节点是否是那些在矩阵中列和最高的节点,这些节点返回了标准化的计数
sort(colSums(net_single2$normCounts1), decreasing = TRUE)[1:10]
plot(2,6)
p3=legend('topleft', cex = 1, pt.cex = 1, title = "Phylum:",
legend=levels(phyla), col = phylcol, bty = "n", pch = 16)
p4=legend(2,5, cex = 1, title = "estimated correlation:",
legend = c("+","-"), lty = 1, lwd = 3, col = c("darkgreen",
"#BF0000"),
bty = "n", horiz = TRUE)
#核心节点
props_single2[["hubs"]][["hubs1"]]
props_single2[["hubs"]][["hubs2"]]
#比较网络
#第一步网络构建一样 net_single2 <- netConstruct
#第二步网络分析,参数看官网,先按他推荐的
props_season <- netAnalyze(net_single2,
centrLCC = FALSE,
avDissIgnoreInf = FALSE,
sPathNorm = FALSE,
clustMethod = "cluster_fast_greedy",
hubPar = c("degree", "closeness","eigenvector"),
hubQuant = 0.95,
lnormFit = FALSE,
normDeg = FALSE,
normBetw = FALSE,
normClose = FALSE,
normEigen = FALSE)
#??netAnalyze
summary(props_season)
#Quantitative network comparison ??netCompare
comp_season <- netCompare(props_season, permTest = TRUE, nPerm = 2,
adjust = "adaptBH" ,
seed=123456, verbose = FALSE)
#??netCompare
summary(comp_season)
summary(comp_season,
groupNames = c("Control", "OSCC"),
showCentr = c("degree", "closeness","eigenvector"),
numbNodes = 20)
props_single2[["hubs"]][["hubs1"]]
props_single2[["hubs"]][["hubs2"]]