基于相似性的k近邻分类
KNN(K-Nearest Neighbors)算法是一种基本的分类和回归算法,它是一种非参数算法,可以用于分类和回归问题。它的基本思想是利用已有的数据集对新的数据进行分类或回归。
在分类问题中,KNN算法通过测量不同特征之间的距离来确定新数据点与训练数据集中最相似的K个数据点,然后将新数据点分类为这K个数据点中出现最频繁的类别。
在回归问题中,KNN算法使用与分类问题相同的基本思想,但是它计算的是K个最近邻点的平均值或加权平均值,然后将该平均值作为新数据点的回归结果。
KNN算法的优点是简单易懂,易于实现,对特征空间的维数没有要求,适用于多分类问题,且对于异常值和噪声的容忍度较高。缺点是计算复杂度高,需要存储所有的训练数据,对于大数据集来说计算和存储代价较高,同时需要对特征空间进行预处理,以便有效地处理数据。
建立第一个KNN模型
#install.packages("mlr",dependencies = TRUE)
library(mlr)
library(tidyverse)
加载数据糖尿病数据集
data(diabetes,package = "mclust") #加载mclust程序包内置的数据-diabetes
diabetesTib <- as_tibble(diabetes)
summary(diabetesTib) # 145个样本,4个变量,class(因变量),其余为自变量
View(diabetesTib)
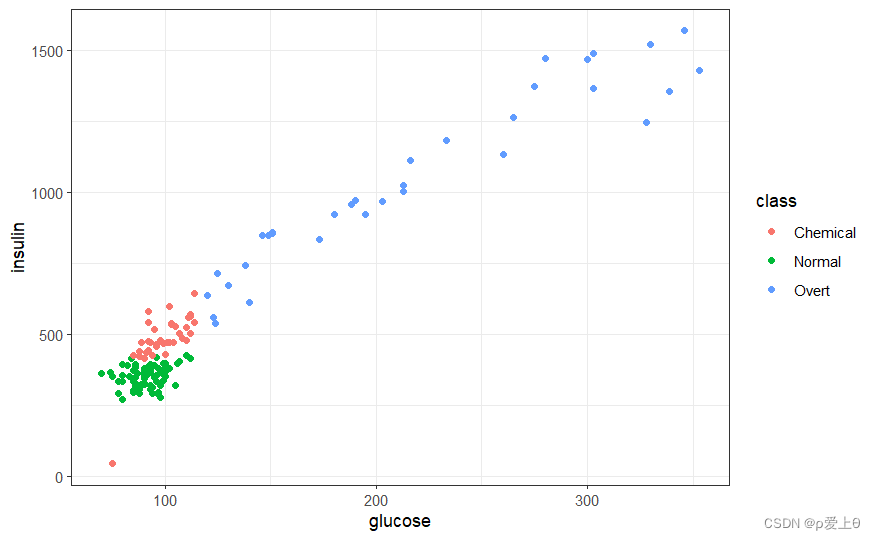
可视化
ggplot(diabetesTib,aes(x=glucose,y=insulin,col=class))+geom_point()+theme_bw()ggplot(diabetesTib,aes(x=sspg,y=insulin,col=class))+geom_point()+theme_bw()ggplot(diabetesTib,aes(x=sspg,y=glucose,col=class))+geom_point()+theme_bw()
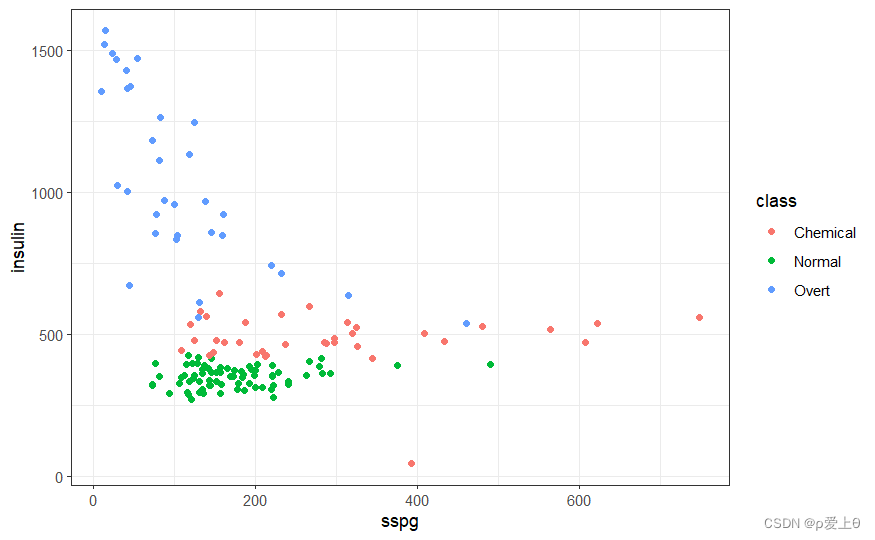
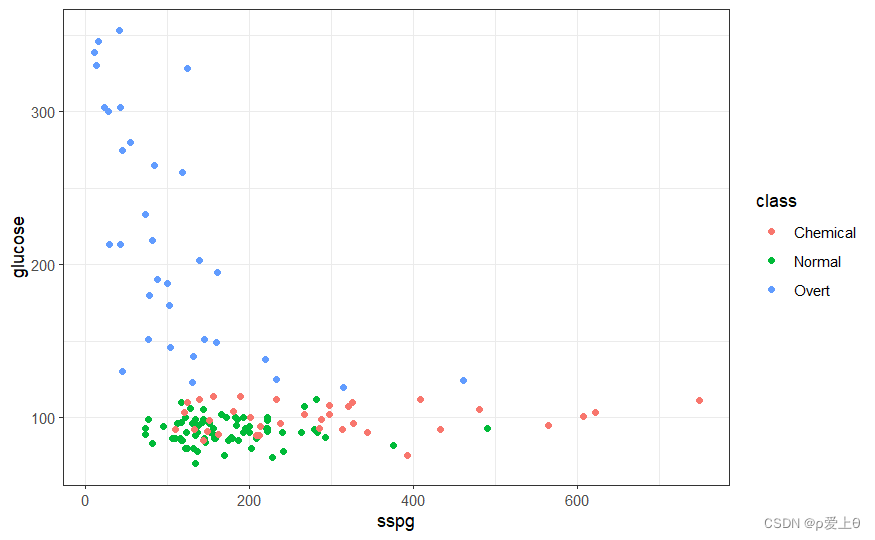
使用mlr训练模型的步骤
- 定义任务
- 包含预测变量的数据
- 期望预测的目标变量 可以通过使用makeClassifTask()函数定义分类任务;makeClusterTask()定义聚类任务;makeRegrTask()定义回归任务,将提供tibble的名称作为data参数,而将包含类别标签的变量名作为target参数
diabetesTask <- makeClassifTask(data = diabetesTib,target = "class")- 定义学习器包括三部分:
- 使用的算法类别:
- 用于分类的"classif"
- 用于回归的"regr"
- 用于聚类的"cluster"
- 用于预测幸存和多标签分类的"surv"和"multilabel"
- 使用的具体算法
- 控制算法的任何其他选项
- 使用的算法类别:
通过使用makeLearner()函数定义学习器,第一个参数是用于训练模型的算法,par.vals代表模型的参数值。
knn <- makeLearner("classif.knn",par.vals = list("k"=2))
- 训练模型 在定义好任务和学习器之后,就可以训练模型了,通过train()函数来训练模型,该函数将学习器作为第一个参数,将任务作为第二个参数。
KnnModel <- train(knn,diabetesTask)
建立好模型后,将数据传递给模型,并观察如何执行。predict()函数作用为:获取未标注的数据,将他们传递给模型,已获得数据的预测类别。predic()函数的第一个参数是模型,想要传递给模型的数据可作为newdata参数给出:
knnpred <- predict(KnnModel,newdata = diabetesTib)
可以利用performance()函数对模型预测的类别与真实类别进行比较,该函数的第一个参数是预测值,第二个参数measures需要指定返回哪些性能度量,需要将返回的性能度量作为列表传递给measures参数。
performance(knnpred,measures = list(mmce,acc))
使用交叉验证判断是否过拟合或者欠拟合
常见的交叉验证方法:
留出法交叉验证:
简而言之就是将数据集划分为两部分:训练集和测试集
在使用mlr进行交叉验证时,第一步是进行重采样描述,重采样描述只是一组关于如何将数据划分为训练集和测试集的指令。makeResampleDesc()函数的第一个参数是使用的交叉验证方法,留出法记作Holdout,对于留出法,需要指定训练集的比例提供给split参数:
hold <- makeResampleDesc(method = "Holdout",split = 0.8,stratify = TRUE)
上面代码中的makeSampleDesc函数的最后一个参数stratify作用是是否将数据集划分为训练集和测试集时,尽量保持每个类别的患者在每个集合中的比例不变,TRUE则代表不变,这在分类问题中很重要。
执行留出法交叉验证:定义好交叉验证学习器后,就可以使用resample()函数进行交叉验证
holdoutCV <- resample(learner = knn,task = diabetesTask,resampling = hold,measures = list(mmce,acc))
性能度量可以通过resampling对象提取$aggr组件来得到:
holdoutCV$aggr
计算混淆矩阵
可以使用mlr程序包中的calcuteConfusionMatrix()函数来计算混淆矩阵,第一个参数是holdoutCV对象的$pred组件,包含测试集的真实类别和预测类别,可选参数relative要求显示每个类别在真实值和预测值标签中的比例:
calculateConfusionMatrix(holdoutCV$pred,relative = TRUE)
- k-折交叉验证:
k-折交叉验证是一种常用的机器学习模型评估方法。它的基本思想是将原始数据集分成k个子集,然后执行k次模型训练和测试,在每次训练和测试中,从这k个子集中选择一个作为测试集,剩下的k-1个子集作为训练集。最后,将k次测试结果的平均值作为模型的性能指标。
k-折交叉验证可以有效地利用数据,因为它可以反复使用数据进行训练和测试,从而得到更准确的模型性能评估。此外,它还可以帮助减少数据集划分的随机性对模型性能评估的影响,从而提高模型性能评估的稳定性。
k的取值通常为5或10,但也可以根据数据集的大小和复杂度进行调整。需要注意的是,使用k-折交叉验证可能会增加训练时间,因为需要进行k次训练和测试。
kFold <- makeResampleDesc(method = "RepCV",folds=5,reps=10,stratify = TRUE) ## 执行10次的5折交叉验证,也就是50次的试验
kFoldCV <- resample(learner = knn,task = diabetesTask,resampling = kFold,measures = list(mmce,acc))
calculateConfusionMatrix(kFoldCV$pred,relative = TRUE)
- 留一法交叉验证:
留一法交叉验证是一种特殊的交叉验证方法,它的特点是将原始数据集分成N个子集,每个子集只包含一个样本。然后,执行N次模型训练和测试,在每次训练和测试中,从N个子集中选择一个作为测试集,剩下的N-1个子集作为训练集。最后,将N次测试结果的平均值作为模型的性能指标。
留一法交叉验证的优点是可以最大程度地利用数据,因为每个样本都被用于测试一次,从而得到更准确的模型性能评估。缺点是计算成本较高,因为需要进行N次训练和测试,特别是当数据集较大时,计算成本会更高。
留一法交叉验证通常用于数据集较小,样本数较少的情况下,特别是在性能评估的精度要求较高的情况下。但是,对于大型数据集,留一法交叉验证可能不太实用,因为计算成本太高。
LOO <- makeResampleDesc(method = "LOO")
LOOCV <- resample(learner = knn,task = diabetesTask,resampling = LOO,measures = list(mmce,acc))
calculateConfusionMatrix(LOOCV$pred,relative = TRUE)
模型的参数与超参数?
在机器学习中,模型的参数和超参数是两个重要的概念。
模型的参数是指在训练过程中需要学习的模型内部权重,也称为学习参数,这些参数是通过训练数据自动学习得到的。例如,在线性回归模型中,模型的参数是回归系数,它们控制着预测变量对目标变量的影响。
超参数是指在模型训练之前需要手动设置的参数,它们控制着模型的学习过程,例如正则化系数、学习率、迭代次数等,这些参数不能通过训练数据自动学习得到,需要手动设置。超参数的选择会影响模型的性能和泛化能力,因此选择合适的超参数是非常重要的。
在模型训练过程中,通常先设置超参数,然后使用训练数据来学习模型的参数。在训练过程中,模型的参数会不断调整以使得模型在训练数据上的表现更好,而超参数的调整则需要通过交叉验证等方法来选择最佳组合。
总之,模型的参数是指需要通过训练数据自动学习得到的模型内部权重,而超参数是需要手动设置的控制模型学习过程的参数。两者都对模型的性能和泛化能力产生重要影响。
KNN算法超参数的调节
在KNN算法中,k值如果设置的较小,学习器将会对数据中的噪声数据进行建模,k值过大将导致模型对数据的局部差异不敏感。下面将演示如何对k进行调整:
- 首先定义mlr访问的k值范围:
knnParameSpace <- makeParamSet(makeDiscreteParam("k",values = 1:10))
在makeParamSet函数中:内置的makeDiscreteParam函数指定需要调整的超参数k,从1到10。
- 接下来定义mlr搜索参数空间的方式:网格搜索、随机搜索
gridSerch <- makeTuneControlGrid() #网格搜索
- 接下来定义交叉验证调节过程,其原理为:对于参数空间的每一个k值,执行重复的k-折交叉验证。对于每个k值,取所有迭代的平均性能度量,并与其他的k值平均性能度量做比较,从而得到最优的k值:
cvForTuning <- makeResampleDesc(method = "RepCV",folds=10,reps = 20)#10折交叉验证,重复20次,一共做200次实验## 现在调用tuneparams()函数执行调节过程:
tunedK <- tuneParams(learner = knn,task = diabetesTask,resampling = cvForTuning,par.set = knnParameSpace,control = gridSerch)
可视化调节过程
knnTuningData <- generateHyperParsEffectData(tunedK)
plotHyperParsEffect(knnTuningData,x="k",y="mmce.test.mean",plot.type = "line")+theme_bw()
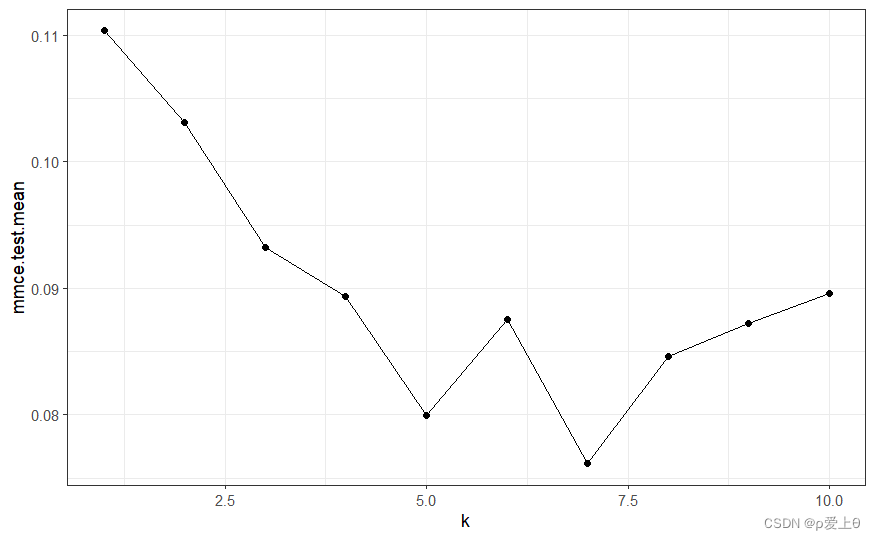
现在使用最优的k值训练最终模型:
tunedknn <- setHyperPars(makeLearner("classif.knn"),par.vals = tunedK$x)
tunedknnModel <- train(learner = tunedknn,task = diabetesTask)
使用模型进行预测,假设有一些新的患者来诊所:
newdata <- tibble(glucose = c(82,108,300),insulin = c(361,288,1052),sspg = c(200,186,135)) # 建立一个新的tibble数据
pred <- predict(tunedknnModel,newdata = newdata) # 对新数据进行预测
getPredictionResponse(pred) #获得预测的结果
使用鸢尾花数据集进行KNN算法训练-预测
data("iris")
irisTib <- as_tibble(iris)
# 首先使用留出法对数据划分为训练集和测试集
hold <- makeResampleDesc(method = "Holdout",split=0.8)
# 创建分类任务
irisTask <- makeClassifTask(data = irisTib,target = "Species")
# 建立学习器
knn <- makeLearner("classif.knn",par.vals = list("k"=5)) #先初始k为5
# 训练学习器
holdCV <- resample(learner = knn,task = irisTask,resampling = hold,measures = list(mmce,acc))
# 通过k-折交叉验证以及网格搜索改进K值并可视化
knnparamrange <- makeParamSet(makeDiscreteParam("k",values = 1:15))
gridserch <- makeTuneControlGrid() #网格搜索
cvForTuning1 <- makeResampleDesc(method = "RepCV",folds = 10,reps =20)#10-折交叉验证,迭代20次,共200次实验
tunedK <- tuneParams("classif.knn",task = irisTask,resampling = cvForTuning1,par.set = knnparamrange,control = gridserch) #调用tuneParams函数执行调节过程
knnTuningData <- generateHyperParsEffectData(tunedK)
plotHyperParsEffect(knnTuningData,x="k",y="mmce.test.mean",plot.type="line")+theme_bw() #可视化
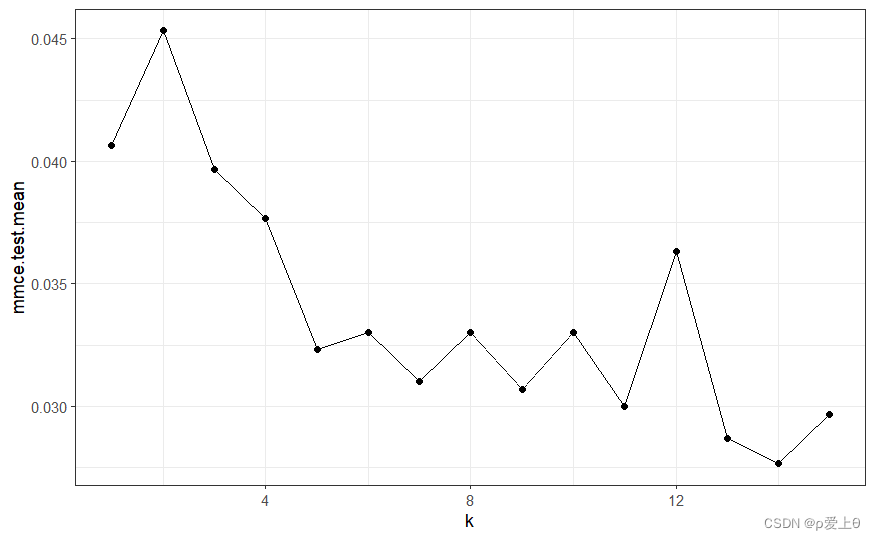
# 使用最优的k值训练最终的模型:
tunedkNN <- setHyperPars(makeLearner("classif.knn"),par.vals = tunedK$x)
tunedkNNmodel <- train(tunedkNN,task = irisTask)
# 对新的数据进行预测
new <- tibble(Sepal.Length=c(7.5,6.2,7.8),Sepal.Width = c(3.0,1.8,4.3),Petal.Length = c(4.9,2.0,1.9),Petal.Width = c(0.15,0.09,0.4)
)
#View(new)
pred_y <- predict(tunedkNNmodel,newdata=new)
getPredictionResponse(pred_y)