RDA环境因子群落结构统计检验可视化
环境因子的筛选及数据的转化方面请参阅宏基因组公众号之前的推文,本文主要侧重统计分析与可视化
看到师兄文章里的图自己可能用到,想复现一下,于是就尝试了一下,顺便写个推文记录,在黄静同学的帮助下完成
百度云链接:https://pan.baidu.com/s/1vNmxPV5kw51Ek_oXMmNycw
提取码:no73
因公众号文章不可修改,如以上链接失效,或想获取代码的更新版,请在“宏基因组”公众号后台回复本文关键字“rdaenv”获取最新下载地址。
rm(list=ls())
library(pacman)
p_load(ggplot2,patchwork,vegan,geosphere,psych,corrplot,permute,lattice,ggpubr,RColorBrewer,tidyverse,graphics)data=read.csv("sum_c.csv",row.names = 1)#读入物种(以Phylum水平为例)矩阵表
head(data,n=3)
env=read.csv("env.csv",row.names = 1)#读入环境因子数据(示例为随机数)
head(env,n=3)print(decorana(t(data)))
#DCA分析,根据Axis lengths行的第一个值选择排序分析模型
#Axis Lengths >4.0-CCA(基于单峰模型,典范对应分析);
#如果在3.0-4.0之间-RDA/CCA均可;
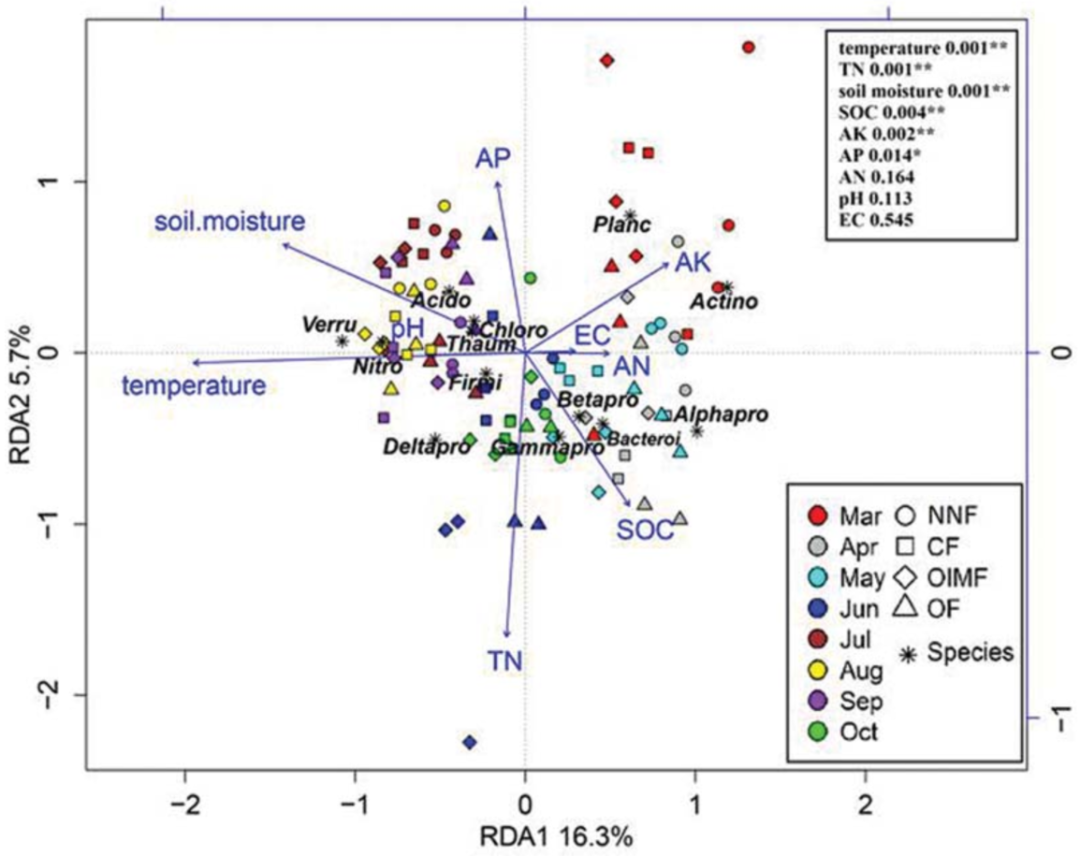
#如果小于3.0-RDA(基于线性模型,冗余分析)B.rda=rda(t(data),env[-1],scale = T)#RDA分析,如果没有环境因子参数,就是PCA分析
#提取样本得分
B.rda.data=data.frame(B.rda$CCA$u[,1:2],env$Treat,rep(c("Mar","Apr","May","Jun","Jul","Aug"),each = 3))#为了仿师兄的图添加的
colnames(B.rda.data)=c("RDA1","RDA2","group","Month")
head(B.rda.data,n=3)#提取物种得分
B.rda.spe=data.frame(B.rda$CCA$v[,1:2])
B.rda.spe=as.data.frame(B.rda.spe)
B.rda.spe$Species<-rownames(B.rda.spe)
head(B.rda.spe,n=3)#提取环境因子得分
B.rda.env <- B.rda$CCA$biplot[,1:2]
B.rda.env <- as.data.frame(B.rda.env)
head(B.rda.env,n=3)#带有环境因子,物种信息且进行不同月份不同处理标记的RDA图(仿师兄)
yanse<-c("darkolivegreen3","gold","dodgerblue4","darkseagreen","chartreuse4","darkorange","burlywood2","brown3","#984EA3","cyan3")
p1=ggplot(data=B.rda.data,aes(RDA1,RDA2))+geom_point(aes(color=Month,fill=Month,shape=group),size=5)+scale_color_manual(values=yanse)+scale_shape_manual(values = c(21,22,23))+scale_fill_manual(values = yanse)+geom_point(data=B.rda.spe,aes(RDA1,RDA2),pch=8,size=5)+geom_text(data=B.rda.spe,aes(x=B.rda.spe[,1],y=B.rda.spe[,2],label=Species),size=5.5,colour="black",hjust=0.5,vjust=1)+labs(title = "B RDA plot",x=paste("RDA1",round(B.rda$CCA$eig[1]/sum(B.rda$CCA$eig)*100,2)," %"),y=paste("RDA2",round(B.rda$CCA$eig[2]/sum(B.rda$CCA$eig)*100,2)," %"))+geom_hline(yintercept = 0,lty=3)+geom_vline(xintercept = 0,lty=3)+geom_segment(data=B.rda.env,aes(x=0,y=0,xend=B.rda.env[,1],yend=B.rda.env[,2]),colour="blue",size=0.8,arrow=arrow(angle = 35,length=unit(0.3,"cm")))+geom_text(data=B.rda.env,aes(x=B.rda.env[,1],y=B.rda.env[,2],label=rownames(B.rda.env)),size=6.5,colour="blue", hjust=(1-sign(B.rda.env[,1]))/2,angle=(180/pi)*atan(B.rda.env[,2]/B.rda.env[,1]))+ggprism::theme_prism()
p1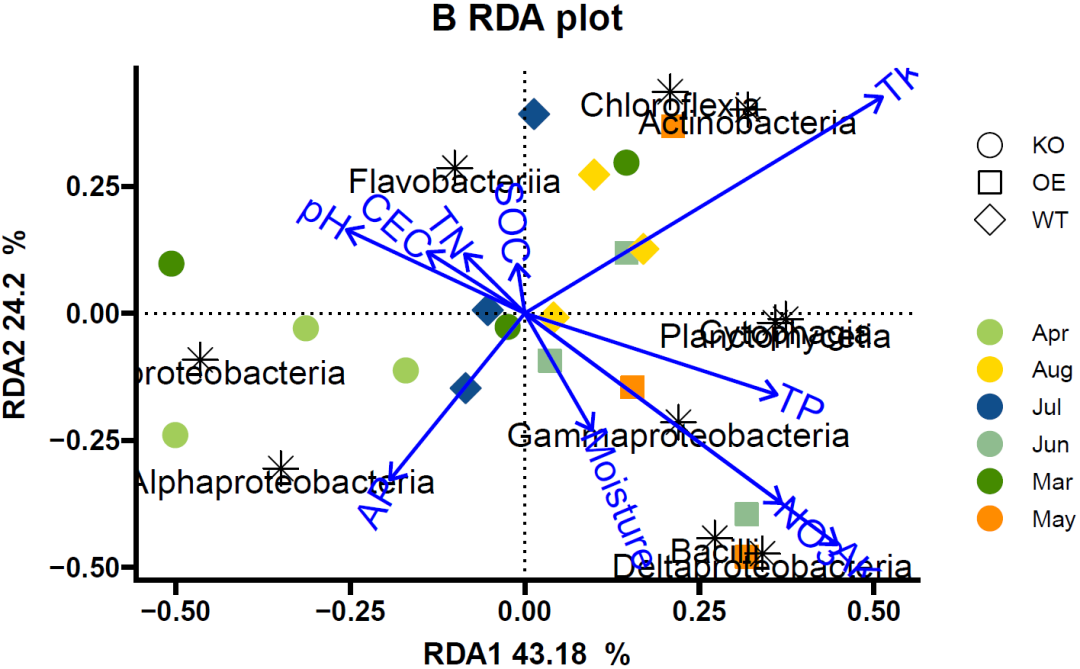
#统计
B.sum=summary(B.rda)
B.sum$constr.chi/B.sum$tot.chi #constrained表示环境因子对群落结构差异的解释度
B.sum$unconst.chi/B.sum$tot.chi#unconstrained表示环境因子对群落结构不能解释的部分#环境因子对群落结构差异解释量的饼图绘制
cor_data=data.frame(row.names = c("Explained","Unexplained"),B=c(B.sum$constr.chi/B.sum$tot.chi,B.sum$unconst.chi/B.sum$tot.chi))
cor_data$group=rownames(cor_data)
head(cor_data,n=3)
cor_data <- data.frame(cor_data)
cor_data=arrange(cor_data,B)
head(cor_data,n=3)
labs<-paste0(cor_data$group,"\n(",round(cor_data$B/sum(cor_data$B)*100,2),"%)")
pie(cor_data$B,labels=labs,init.angle = 90,col=brewer.pal(nrow(cor_data),"Reds"),boder="black")
#在R中手动导出,右侧出图区Export-PDF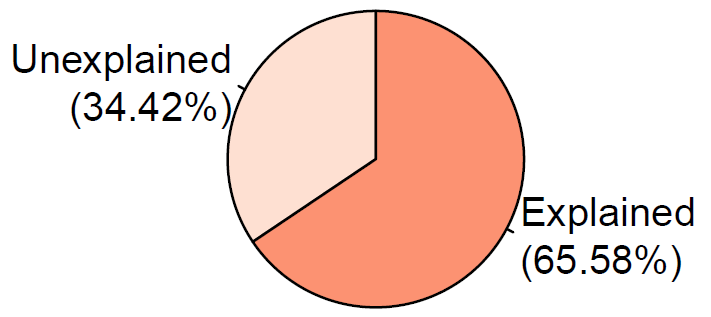
#anova.cca检验
B.perm=permutest(B.rda,permu=999) # permu=999是表示置换999次
B.perm #是做环境因子整体与群落结构差异的相关性(解释量),anova.cca {vegan}#envfit检验 envfit函数跟mantel(见下文)的功能是一样的
B.ef=envfit(B.rda,env[-1],permu=999)#是做每一个环境因子与群落结构差异的相关性(解释量)
B.ef$vectors$r#R2值
B.ef$vectors$pvals#P值#每一个环境因子对群落结构差异解释量的柱形图绘制的数据整理
cor_com=data.frame(tax=rownames(B.rda.env),B.r=B.ef$vectors$r,B.p=B.ef$vectors$pvals)
cor_com=arrange(cor_com,B.r)
head(cor_com,n=3)
cor_com[c(3)]=cor_com[c(3)]>0.05
head(cor_com,n=3)
#将p<0.05标记为FALSE,p>0.05标记为TRUE,使用此数据绘制柱形图,将其可视化#envfit检验可视化
cor_com$tax = factor(cor_com$tax,order = T,levels = row.names(cor_com))#按R2值排序
p2 <- ggplot(cor_com, aes(x =tax, y = B.r),size=2) +geom_bar(stat = 'identity', width = 0.8,color="black",fill="red") +scale_fill_manual(guide = FALSE)+geom_text(aes(y = B.r+0.005, label = ifelse(B.p==TRUE,"","*")),size = 5, fontface = "bold") +xlab("Environmental factor")+ylab(expression(r^"2"))+scale_y_continuous(expand = c(0,0))+ggprism::theme_prism()+theme(axis.text.x = element_text(angle = 45))
p2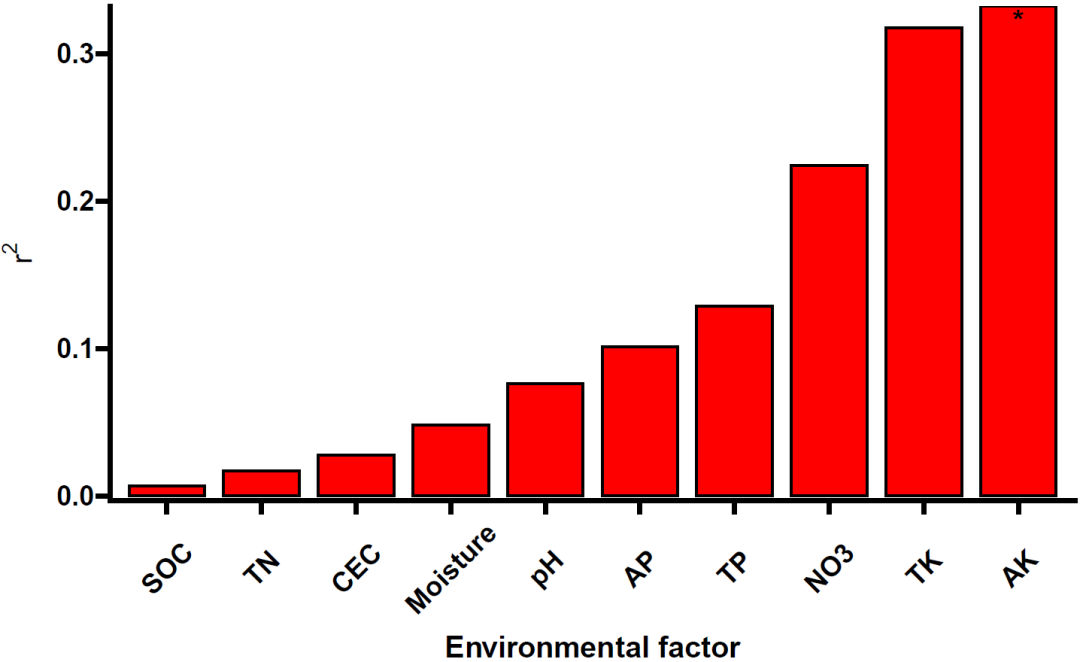
#mantel检验
data <- as.data.frame(t(data))
species.distance<-vegdist(data,method = 'bray')
soil <- NULL
for (i in 2:ncol(env)) {dd <- mantel(species.distance, vegdist(env[,i], method = "euclidean"), method = "pearson", permutations = 9999, na.rm = TRUE)soil <- rbind(soil,c(colnames(env)[i],dd$statistic, dd$signif))
}
head(soil,n=3)
soil <- data.frame(aa=rownames(B.rda.env),M_r=soil[,2],M.p=soil[,3])
rownames(soil)=soil$aa
soil=arrange(soil,M_r)
soil[c(3)]=soil[c(3)]>0.05 # 将p<0.05标记为FALSE,p>0.05标记为TRUE,同上
soil$aa = factor(soil$aa,order = T,levels = row.names(soil))
soil$M_r=round(as.numeric(soil$M_r),4)
head(soil,n=3)#mantel检验可视化
p3 <- ggplot(soil, aes(x =aa, y = M_r),size=2) +geom_bar(stat = 'identity', width = 0.8,color="black",fill="red") +scale_fill_manual(guide = FALSE)+geom_text(aes(y = M_r+0.005, label = ifelse(M.p==TRUE,"","*")),size = 5, fontface = "bold") +xlab("Environmental factor")+ylab(expression(r))+ggprism::theme_prism()+theme(axis.text.x = element_text(angle = 45))
p3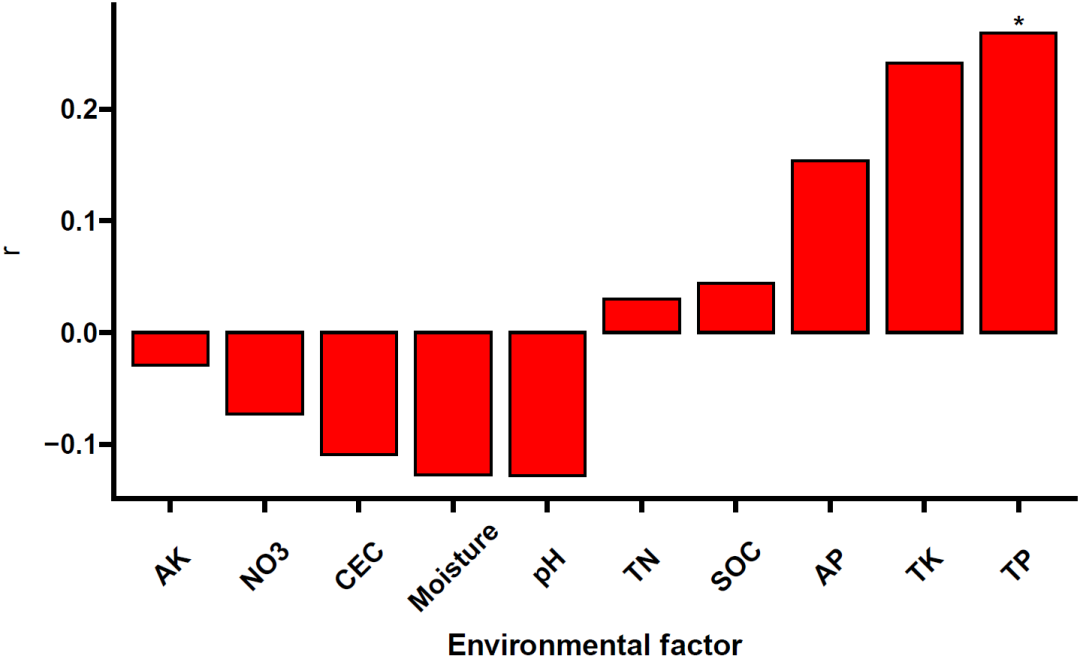
##################群落结构差异的统计检验(三种方法及可视化)
####Adonis
otu <- data.frame(data)#mantel检验时已经转置,勿要转置
head(otu)
#样本分组文件
group <- read.delim('metadata.txt', sep = '\t', stringsAsFactors = FALSE)
head(group)
#使用 Bray-Curtis 距离测度 unifrac_binary
adonis_result <- adonis(otu~Group, group, distance = 'Bray-Curtis', permutations = 999)
adonis_result$aov.tabgroup_name <- unique(group$Group)adonis_result_two <- NULL
for (i in 1:(length(group_name) - 1)) {for (j in (i + 1):length(group_name)) {group_ij <- subset(group, Group %in% c(group_name[i], group_name[j]))otu_ij <- otu[group_ij$SampleID, ]adonis_result_otu_ij <- adonis(otu_ij~Group, group_ij, permutations = 999, distance = 'bray')adonis_result_two <- rbind(adonis_result_two, c(paste(group_name[i], group_name[j], sep = '_'), 'Bray-Curtis', unlist(data.frame(adonis_result_otu_ij$aov.tab, check.names = FALSE)[1, ])))}
}
adonis_result_two <- data.frame(adonis_result_two, stringsAsFactors = FALSE)
names(adonis_result_two) <- c('group', 'distance', 'Df', 'Sums of squares', 'Mean squares', 'F.Model', 'R2', 'P')
adonis_result_two$R2<- as.numeric(adonis_result_two$R2)
adonis_result_two$P <- as.numeric(adonis_result_two$P)
#p值Benjamini校正 作图时自己选择用哪一个P值(下同)
adonis_result_two$P_adj_BH <- p.adjust(adonis_result_two$'P', method = 'BH')
head(adonis_result_two)
adonis_result_two=arrange(adonis_result_two,R2)
adonis_result_two
adonis_result_two$P=adonis_result_two$P>0.05#将p<0.05标记为FALSE,p>0.05标记为TRUE,使用此数据绘制柱形图(下同)
adonis_result_two$tax = factor(adonis_result_two$group,order = T,levels = adonis_result_two$group)
adonis <- ggplot(adonis_result_two, aes(x =tax, y = R2),size=2) +geom_bar(stat = 'identity', width = 0.8,color="black",fill="red")+scale_fill_manual(guide = FALSE)+geom_text(aes(y = R2-0.02, label = ifelse(P==TRUE,"","*")),#选择作图的P值(P_adj_BH)(下同)size = 5, fontface = "bold") +xlab("")+ylab(expression(r^"2"))+scale_y_continuous(expand = c(0,0))+ggprism::theme_prism()+theme(axis.text.x = element_text(angle = 0))
adonis ##图片大小在保存时自己调合适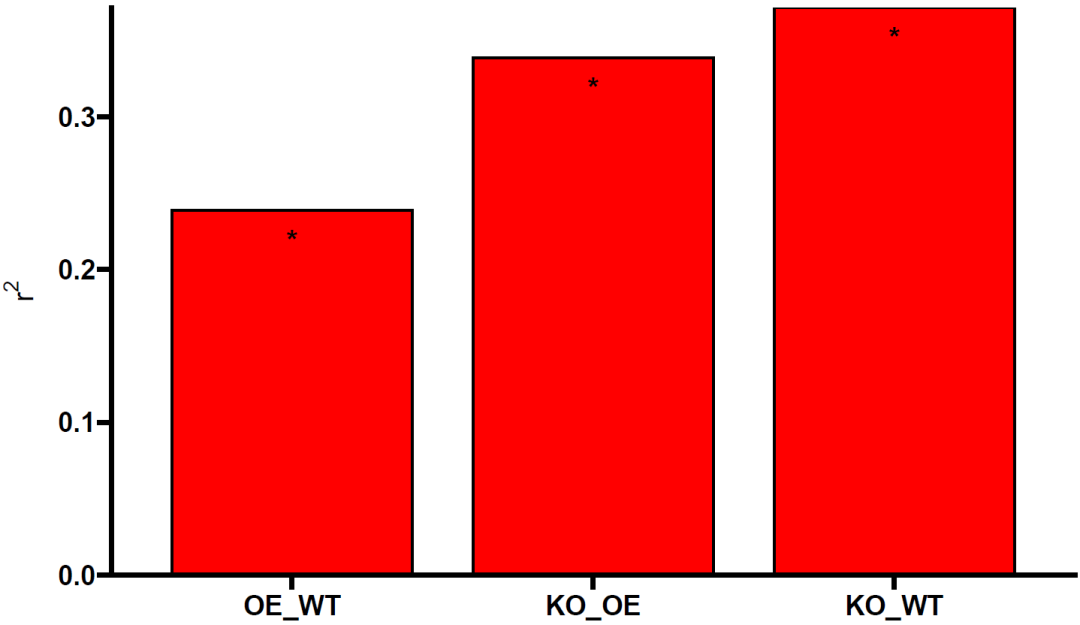
####Anosim
anosim_result <- anosim(otu, group$Group, distance = 'bray', permutations = 999)
anosim_result$signif #p 值
anosim_result$statistic #R 值group_name <- unique(group$Group)
anosim_result_two <- NULL
for (i in 1:(length(group_name) - 1)) {for (j in (i + 1):length(group_name)) {group_ij <- subset(group, Group %in% c(group_name[i], group_name[j]))otu_ij <- otu[group_ij$SampleID,]anosim_result_otu_ij <- anosim(otu_ij, group_ij$Group, permutations = 999, distance = 'bray')anosim_result_two <- rbind(anosim_result_two, c(paste(group_name[i], group_name[j], sep = '_'), 'Bray-Curtis', anosim_result_otu_ij$statistic, anosim_result_otu_ij$signif))}
}
anosim_result_two <- data.frame(anosim_result_two, stringsAsFactors = FALSE)
names(anosim_result_two) <- c('group', 'distance', 'R', 'P')
anosim_result_two$R<- as.numeric(anosim_result_two$R)
anosim_result_two$P <- as.numeric(anosim_result_two$P)
anosim_result_two$P_adj_BH <- p.adjust(anosim_result_two$P, method = 'BH')
head(anosim_result_two)anosim_result_two=arrange(anosim_result_two,R)
anosim_result_two
anosim_result_two$P=anosim_result_two$P>0.05
anosim_result_two$tax = factor(anosim_result_two$group,order = T,levels = anosim_result_two$group)
anosim <- ggplot(anosim_result_two, aes(x =tax, y = R),size=2) +geom_bar(stat = 'identity', width = 0.8,color="black",fill="red")+scale_fill_manual(guide = FALSE)+geom_text(aes(y =R-0.02, label = ifelse(P==TRUE,"","*")),size = 5, fontface = "bold") +xlab("")+ylab(expression(r))+scale_y_continuous(expand = c(0,0))+ggprism::theme_prism()+theme(axis.text.x = element_text(angle = 0))
anosim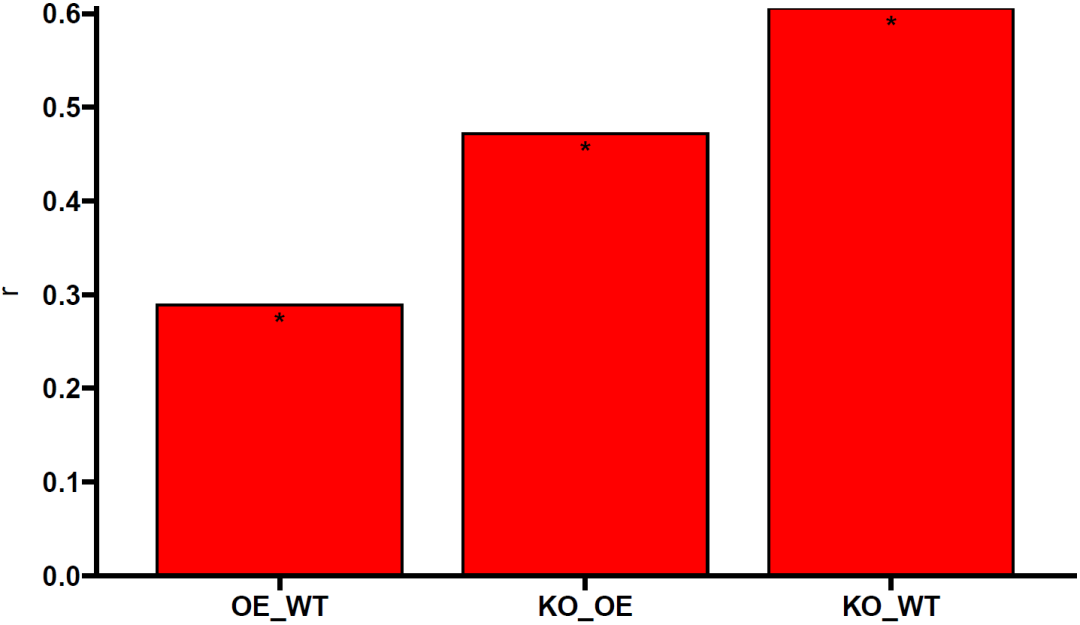
##MRPP
mrpp_result <- mrpp(otu, group$Group, distance = 'bray', permutations = 999)
mrpp_result$Pvalue # p 值
mrpp_result$A #相当于Anosim检验的R值roup_name <- unique(group$Group)
mrpp_result_two <- NULL
for (i in 1:(length(group_name) - 1)) {for (j in (i + 1):length(group_name)) {group_ij <- subset(group, Group %in% c(group_name[i], group_name[j]))otu_ij <- otu[group_ij$SampleID,]mrpp_result_otu_ij <- mrpp(otu_ij, group_ij$Group, permutations = 999, distance = 'bray') mrpp_result_two <- rbind(mrpp_result_two, c(paste(group_name[i], group_name[j], sep = '_'), 'Bray-Curtis', mrpp_result_otu_ij$A, mrpp_result_otu_ij$delta, mrpp_result_otu_ij$E.delta, mrpp_result_otu_ij$Pvalue))}
}
mrpp_result_two <- data.frame(mrpp_result_two, stringsAsFactors = FALSE)
names(mrpp_result_two) <- c('group', 'distance', 'A', 'Observe_delta', 'Expect_delta', 'P')
mrpp_result_two$A<- as.numeric(mrpp_result_two$A)
mrpp_result_two$P <- as.numeric(mrpp_result_two$P)
mrpp_result_two$P_adj_BH <- p.adjust(mrpp_result_two$P, method = 'BH')
head(mrpp_result_two)
mrpp_result_two=arrange(mrpp_result_two,A)
mrpp_result_two
mrpp_result_two$P=mrpp_result_two$P>0.05
mrpp_result_two$tax = factor(mrpp_result_two$group,order = T,levels = mrpp_result_two$group)
MRPP <- ggplot(mrpp_result_two, aes(x =tax, y = A),size=2) +geom_bar(stat = 'identity', width = 0.8,color="black",fill="red")+scale_fill_manual(guide = FALSE)+geom_text(aes(y =A-0.02, label = ifelse(P==TRUE,"","*")),size = 5, fontface = "bold") +xlab("")+ylab(expression(r))+scale_y_continuous(expand = c(0,0))+ggprism::theme_prism()+theme(axis.text.x = element_text(angle = 0))
MRPP # 其余数据集重复绘制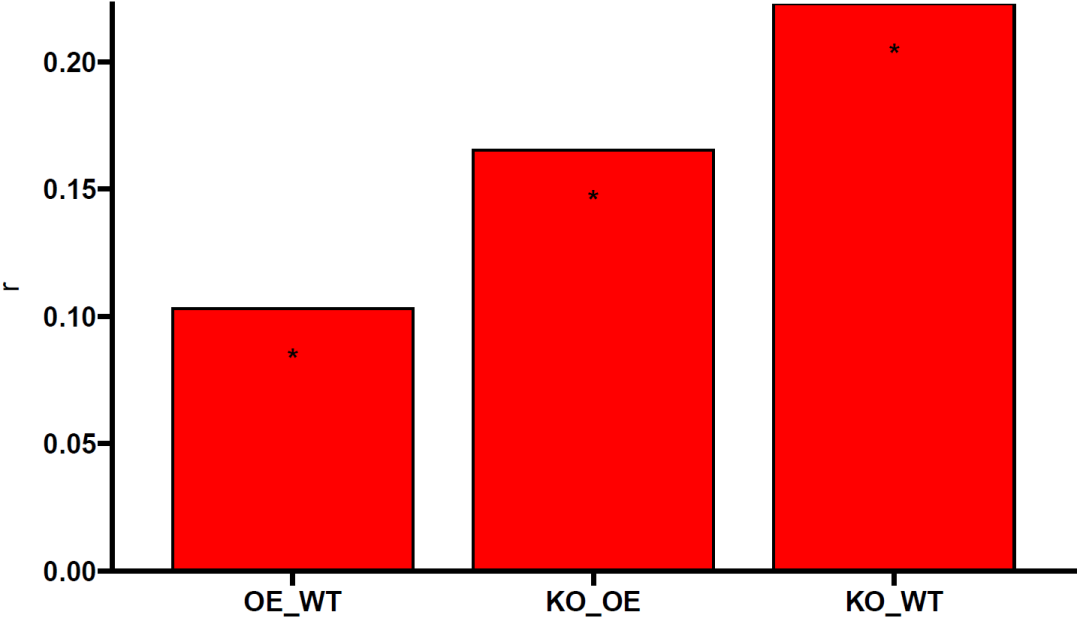
#Output figure width and height
# Letter纸图片尺寸为单栏89 mm,双栏183 mm,页面最宽为247 mm 推荐比例16:10,
# 即半版89 mm x 56 mm; 183 mm x 114 mm
#
##################保存ggsave("./p1.pdf", p1, width = 183, height = 114, units = "mm")ggsave("./p2.pdf", p2, width = 183, height = 114, units = "mm")ggsave("./p3.pdf", p3, width = 183, height = 114, units = "mm")ggsave("./p4.pdf", adonis, width = 183, height = 114, units = "mm")ggsave("./p5.pdf", anosim, width = 183, height = 114, units = "mm")ggsave("./p6.pdf", MRPP, width = 183, height = 114, units = "mm")参考资料
R绘图-RDA排序分析
R包vegan的置换多元方差分析(PERMANOVA)判断群落结构差异
R包vegan的相似性分析(ANOSIM)判断群落结构差异
R包vegan的MRPP分析判断群落结构差异
作者:黄静、旭日阳光
责编:马腾飞 南京农业大学
审核:刘永鑫 中科院遗传发育所
猜你喜欢
10000+:菌群分析 宝宝与猫狗 梅毒狂想曲 提DNA发NatureCell专刊肠道指挥大脑
系列教程:微生物组入门 Biostar 微生物组 宏基因组
专业技能:学术图表 高分文章 生信宝典 不可或缺的人
一文读懂:宏基因组 寄生虫益处 进化树
必备技能:提问 搜索 Endnote
文献阅读 热心肠 SemanticScholar Geenmedical
扩增子分析:图表解读 分析流程 统计绘图
16S功能预测 PICRUSt FAPROTAX Bugbase Tax4Fun
在线工具:16S预测培养基 生信绘图
科研经验:云笔记 云协作 公众号
编程模板: Shell R Perl
生物科普: 肠道细菌 人体上的生命生命大跃进 细胞暗战 人体奥秘
写在后面
为鼓励读者交流、快速解决科研困难,我们建立了“宏基因组”专业讨论群,目前己有国内外5000+ 一线科研人员加入。参与讨论,获得专业解答,欢迎分享此文至朋友圈,并扫码加主编好友带你入群,务必备注“姓名-单位-研究方向-职称/年级”。PI请明示身份,另有海内外微生物相关PI群供大佬合作交流。技术问题寻求帮助,首先阅读《如何优雅的提问》

学习16S扩增子、宏基因组科研思路和分析实战,关注“宏基因组”
点击阅读原文,跳转最新文章目录阅读