1,安装easyPubMed包:
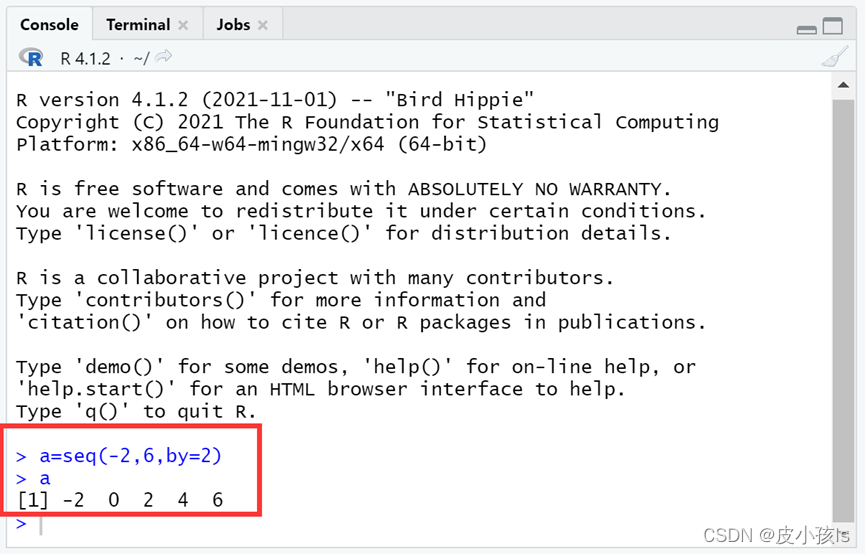
install.packages("easyPubMed")
2,关键字搜索下载摘要信息
library(easyPubMed)
query <- "GATK" #以关键字“GATK”为例
file_name <- "GATK" #保存结果的输出文件的文件名前缀
path <- "C:\\Users\\EDY\\Desktop\\GATK_abstra"
setwd(path) #设置结果文件的保存路径
out.A <- batch_pubmed_download(pubmed_query_string = query,format = "xml",batch_size = 100, #每个文件保存的记录个数dest_file_prefix = file_name,encoding = "UTF8")
运行完成后在"C:\Users\EDY\Desktop\GATK_abstra"目录下会生成对应的xml文件: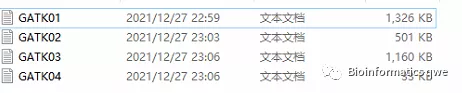
一共会生成多少文件取决于在PubMed一共有多少记录以及“batch_size”参数的设置。
文件的内容大致如下:

3,从xml文件中提取信息(pmid、doi、摘要等)
path <- "C:\\Users\\EDY\\Desktop\\GATK_abstra"
merge_txt <- paste0(path,"\\GATK01.txt") #也可以把多个文件合并到一起再操作
abstra_df <- table_articles_byAuth(pubmed_data = merge_txt, included_authors = "first", max_chars = 100000000, encoding = "UTF8")
outfile <- paste0(path,"\\GATK.abstra")
write.table(abstra_df,outfile,row.names=F,sep="\t",quote=T)
运行完成后会生成“GATK.abstra”文件,对应的内容如下:

第一行为表头,一共14列,其中第四列即为摘要信息。